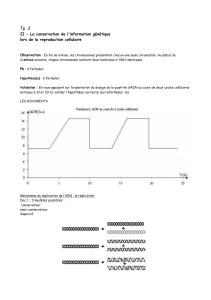
réplication

!"#$%"&$'%()*()+',%$#'$-$.'()(&)/'$-$.'()+$-0,1-"'%(!
""""""""""""""""""""""""""""""""""""""!
23'4(%5'&0)+$6"7(*)8)9):.*"-;)<",1-&0)*(5)=,'(3,(5)/>?>)@A@B)9))
C"#"&)9)+:CDE!
Filière'SVI'–'S4!
Module'de'Biologie'Moléculaire'!
II.'Le'Dogme'Central'
?%>)/F!G:HI)
:330()13'4(%5'&"'%(J)KA@B9KA@L)

Plan'du'cours'de'Biologie'Moléculaire'
!!!!
I.'Initiation'aux'techniques'usuelles'de'biologie'
moléculaire!'
II.'Le'Dogme'Central''!
'Introduction!
'II.1'La'réplication'!
'II.2'La'transcription''!
'II.3'La'traduction''!
'II.4'Régulation'génétique'chez'les'bactéries'!

Support'de'l’information'génétique'et'Macromolécules'
Génome:'"#$"%&'"!("!)*#"$!
Gène:'+#,-.!/0#1-,0##"''"!("!'’,#/02%3-,0#!).#.-,4+"5!
Composition:'31,("$!(.$0672,&0#+1'.,4+"$!89:;<=!$.4+"#1"$!
10%>0$.$!("!&3$"$!>+2,4+"$!89(.#,#"=!?+3#,#"<!"-!>72,%,(,4+"$!
8@A7%,#"=!B7-0$,#"<=!(C0$"$!"-!(C+#!)20+>"%"#-!>A0$>A3-"!
INTRODUCTION''

F-$3."M$3)5()N"'&)O"%)
-P(Q&%'7'&0)RPDS)*()-P$5(J)
)
" <$%7"M$3)*()-")-'"'5$3)
O6$5O6$*'(5&(%)
" )F-'7'3"M$3)*P13)
OT%$O6$5O6"&()
)
#$%&'()*+!
,-./0(1/02,+!

?CDUFIVF)
:CV)
:HV)
Matrice'ADN''
ADN'polymérase'III'
Protéines'de'
réplication'
dNTP'
ATP'
Mg'2+'
Matrice'ADN'(brin'sens)'
ARN'polymérase''
Facteurs'de'transcription'
NTP'
Mg'2+'
Matrice'ARNm'
Ribosomes'
Facteurs'de'traduction'
Acides'aminés'
ARNt'
Synthases'
ATP,GTP,'Mg'2+''
Le'dogme'central'
Transcription' Traduction'
Réplication'
Flux'de'l’information'génétique'unidirectionnel'
!"##$%&'($'#)&*+,$('-*%*./0$'1$'&+)%1#$&'2$'3)4"%'
$5)6&$'270%$'-*%*+)."%'8'0%$')0&+$'9'''''
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
1
/
49
100%