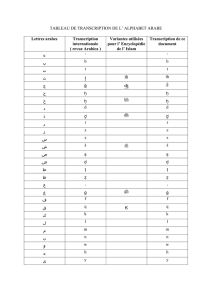
transcription

UNIVERSITE CADI AYYAD, FACULTE DES SCIENCES SEMLALIA
DEPARTEMENT DE BIOLOGIE
BIOLOGIE MOLECULAIRE
COURS S4 (TROISIEME PARTIE)
DU GENOTYPE AU PHENOTYPE
[ TRANSCRIPTION ]
PROFESSEUR A. A. BENSLIMANE
2006
- 0 -

- 1 -
DU GENOTYPE AU PHENOTYPE
[ TRANSCRIPTION ]
SOMMAIRE
1. REPERES HISTORIQUES
2
2. MECANISME GENERAL DE LA SYNTHESE
DES PROTEINES
4
3. LA TRANSCRIPTION 6
I. LA TRANSCRIPTION CHEZ LES PROCARYOTES 7
1. INITIATION (et caractéristiques de la RNA-polymérase) 7
2. ELONGATION 10
3. TERMINAISON 11
α- REGULATION DE L’EXPRESSION GENIQUE CHEZ LES
PROCARYOTES
12
β- OPERONS : CONCEPT DE BASE 13
γ- REGULATION GENIQUE NEGATIVE : ‘REPRESSIBLE’ ou
bien ‘INDUCTIBLE’
14
δ- REGULATION GENIQUE POSITIVE
15
II. LA TRANSCRIPTION CHEZ LES EUCARYOTES 17
1. INITIATION (RNA-POLYMERASE II) 18
2. ELONGATION 23
3. TERMINAISON 23
4. MODIFICATIONS DU PRE-MESSAGER : MATURATION DU
mRNA
23
α- LA FIXATION DE LA COIFFE EST TRES PRECOCE 23
β- LES RNA SONT LE PLUS SOUVENT POLYADENYLES 24
γ- L’EPISSAGE ELIMINE LES INTRONS DU TRANSCRIT
PRIMAIRE
25
MECANISME GENERAL DE L’EPISSAGE 26
CAS PARTICULIERS DE L’EPISSAGE DES INTRONS 27
L’EPISSAGE EN TRANS (« trans-splicing ») 27
L’EPISSAGE ALTERNATIF (OU DIFFERENTIEL) 28
L’AUTO-EPISSAGE (NOTION DE RYBOZYMES) 29
δ- ‘RNA-EDITING’ (CORRECTION DU RNA) 30
5. TRANSPORT DU mRNA VERS LE CYTOPLASME 31

- 2 -
DU GENOTYPE AU PHENOTYPE
[ TRANSCRIPTION ]
Un zygote humain possède dans le DNA de son noyau les instructions essentielles à la
construction d’une personne particulière avec ses caractères propres. Le phénotype de chaque
individu résultera de la combinaison de son bagage génétique unique et des influences du
milieu.
Le DNA contrôle le métabolisme en commandant aux cellules de fabriquer des enzymes
spécifiques et d’autres protéines. C’est en dictant la synthèse de ces protéines que le DNA
hérité d’un organisme va produire les caractères spécifiques d’un individu : le phénotype.
Remarque terminologique : on désigne les gènes avec des minuscules en lettres italiques (ou
soulignées). On écrira par exemple les gènes lac (ou lac). Pour chaque gène particulier, on
fera suivre d’une majuscule (exemple : gène lacZ, lacY, lacA). Les mêmes lettres sont
utilisées pour le produit du gène ou bien pour le phénotype mais la première lettre sera écrite
en majuscule et l’ensemble en lettres droites. On écrira ainsi la protéine LacZ (il s’agit de la
β–galctosidase) et la bactérie Lac+ ou lac- selon qu’il s’agit de bactérie sauvage (+) ou
mutante (-).
Les protéines forment le lien entre le génotype et le phénotype.
1. REPERES HISTORIQUES
La génétique moderne remonte aux travaux de Mendel, qui le premier établit les lois de
l'hérédité. Il publie ses résultats en [1866], mais ils passent alors à peu près inaperçus. Leur
redécouverte n'aura lieu qu'en [1900].
Emergence de la génétique formelle : Les lois de Mendel impliquent l'existence d'éléments
autonomes et reproductibles, qui contrôlent de façon discrète les caractères héréditaires de
génération en génération. Chaque caractère est représenté dans l'oeuf fécondé par deux - et
seulement deux - éléments, provenant l'un du père, l'autre de la mère.
Ces éléments autonomes, unités de l'hérédité, se verront, en [1909], attribuer par le biologiste
danois Wilhem Johannsen la dénomination de « gènes ».
Support de l’hérédité : le chromosome : Ce sont les travaux de Morgan [1904] (Prix Nobel
1933), sur la drosophile (Drosophila melanogaster), qui conduisent au développement de la
« théorie chromosomique » de l'hérédité. Les gènes sont alors localisés sur les
chromosomes, et avec Sturtevant, ils pourront même y être ordonnés par l'étude de la
ségrégation des caractères, constituant les premières « cartes génétiques ». C'est encore dans
le laboratoire de Morgan que sont développées les procédures de « mutagenèse
expérimentales » par Muller [1927] (Prix Nobel 1946) (induction artificielle de mutations par
les rayons X).
Convergence de la biochimie et de la génétique : Si la présence des gènes sur les
chromosomes est alors établie, rien n’est connu de la nature biochimique des gènes ou de leur
mode d’action.

La première relation entre gène et un enzyme est établie en [1909] par Garrod à partir d’une
observation portant sur une maladie humaine : l’« alcaptonurie ». Cette maladie se manifeste
par le noircissement des urines lorsqu'elles sont exposées à l'air. Le noircissement est dû à la
présence dans les urines d'acide homogentisique, qui est un produit intermédiaire de la
dégradation de la tyrosine et de la phénylalanine. Cette substance est dégradée chez les
individus normaux, mais pas chez les alcaptonuriques, chez lesquels elle s'accumule. Le
sérum des premiers contient l'enzyme capable de la métaboliser « l'homogentisate 1,2
désoxygénase », mais cet enzyme n'est pas présent dans le sérum des seconds. Garrod
propose que chaque enzyme serait le fruit de l'activité d'un gène.
- 3 -
Cette corrélation allait être généralisée par
Beadle et Tatum [1941] (Prix Nobel 1958)
grâce aux mutants métaboliques de
Neurospora crassa, un système plus
accessible à l’expérimentation. L'ensemble
de ces travaux aboutissent finalement à la
conclusion que les gènes contrôlent la
synthèse des enzymes, et que chaque
protéine est codée par un gène différent ;
d’où l’expression : « un gène. ↔.une
enzyme » généralisé par la suite en « un
gène ↔ un polypeptide ».

- 4 -
Des travaux similaires seront par la suite produits selon cette stratégie par de nombreux autres
chercheurs. Tous confirment que chaque étape des voies biochimiques est contrôlée par un
gène unique, codant l'enzyme impliquée à cette étape.
DNA comme support de l’information génétique : Le premier phénomène qui allait
permettre de progresser dans l'identification du support de l'hérédité est celui de la
transformation bactérienne, rapporté en [1928] par l'anglais Griffith (travaux sur Diplococcus
pneumomiae – souris). Ce phénomène représente alors un test d'activité biologique, grâce
auquel il est possible de déterminer la nature du matériel génétique. Ce test ne sera pas mis à
profit par Griffith lui même, mais par Avery [1944] qui l'utilise pour élucider la nature
biochimique du matériel génétique : il s'agit de DNA. Cette découverte est toutefois accueillie
avec beaucoup de scepticisme.
Il faudra de nombreux autres travaux pour que cette réalité soit acceptée : en particulier ceux
de Chargaff [1950] (rapports A+T/C+G et A/T, C/G) ou de Hershey [1946] (travaux menés
sur les bactériophages ; phage T2 en particulier). L'acceptation définitive ne viendra qu'avec
l'élucidation de la structure du DNA par Watson et Crick [1953] (Prix Nobel 1962). C’est à
partir de ce moment que la biologie moléculaire va connaître son apothéose et son
développement dans presque tous les domaines et disciplines scientifiques.
2. MECANISME GENERAL DE LA SYNTHESE DES
PROTEINES
Qu’est ce qu’un gène ? : La définition du gène a évolué au fur et à mesure de l’avancement
scientifique des connaissances. En fait chacune des définitions a son utilité selon le
contexte dans lequel on étudie les gènes.
Les concepts mendéliens définissent le gène en tant qu’unité héréditaire discontinue
possédant une influence sur un caractère phénotypique.
Morgan et ses collaborateurs ont localisé le gène sur un endroit précis du chromosome : locus.
Ensuite, une définition fonctionnelle du gène a été formulée ; « le gène est une séquence de
DNA codant pour une chaîne polypeptidique spécifique » : « un gène - un polypeptide ».
Cette dernière définition doit être utilisée avec discernement.
En effet, la plupart des gènes eucaryotes comprennent des régions non codantes
« introns », c'est-à-dire de grands segments qui n’ont pas d’équivalents dans les
polypeptides.
Les biologistes moléculaires incluent également dans le gène les promoteurs et
d’autres régions régulatrices qui ne sont pas transcrites, mais on peut considérer que
ces séquences font partie du gène parce que le gène ne sera exprimé qu’en leur
présence.
A l’échelle moléculaire, la définition du gène doit également englober le DNA qui
code pour le RNA ribosomal, RNA de transfert et autres petits RNA. Ces gènes ne
produisent pourtant pas de polypeptides.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
1
/
33
100%