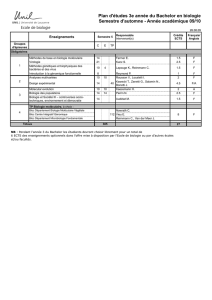
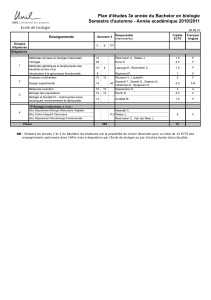
Master “In silico Drug Design” Semestre 2 Bioinformatique

1
Master “In silico Drug Design”
Semestre 2 Bioinformatique Structurale & Drug Discovery
30UAIS42 INITIATION A LA PROGRAMMATION (5 ECTS)
Responsable : A. BADEL
30ISA142 - Initiation à Python (3 ECTS)
Enseignants : D. MESTIVIER, T. CRAGNOLINI
Intitulé : Initiation à Python
Objectifs :
Former les biologistes à la programmation par le biais d'un langage ouvert et accessible aux novices :
python. A l'issue de ce module, les étudiants devront être capables de faire leurs propres scripts
d'analyse de résultats.
- La notion de programmation
- Introduction au langage Python
- Notion d'objet en Python
- Le module BioPython
- L'API PyMol
- Programmation objet en Python: classes et héritage
- Scripting, wrap de programmes externes
- Interface Python/C/R
-
- Partie pratique :
- Applications directes de la partie théorique avec comme finalité :
- - la rédaction d’un script d'analyse de résultat de Blast?
- - la rédaction d'une commande/plugin PyMol
- Applications directes de la partie théorique avec comme finalité :
- - conception d'une classe minimale pour l'analyse de fichiers
- de séquence au format FASTA
- - interface avec un programme externe comme CLUSTALW
- - extension d'une classe pour parser les structures au format PDB
Modalités d’évaluation : examen ou projet
Nombre d’heures :
Heures de cours : 21 heures
Heures de TD : 25 heures

2
30ISA242 - Projet de programmation (2 ECTS)
Enseignant : T. CRAGNOLINI
Intitulé : Projet de programmation (Python)
Objectifs :
Réalisation d'un projet de programmation demandant une soixantaine d'heure de travail aux étudiants
et mettant en jeu les compétences en Python et en algorithmique acquises acquise dans l’EC1.
Compétences visées :
L’objectif de ce cours est de renforcer les concepts fondamentaux d’algorithme et leur application
pratique dans un langage orienté objet.
Modalités d’évaluation : projet
Nombre d’heures :
Heures de cours : 5 heures
Heures de TD : 25 heures

3
Semestre 2 Bioinformatique Structurale & Drug Discovery
30UBIS42- DRUG DESIGN (5 ECTS)
Responsables : O. TABOUREAU et G. MOROY
Cet enseignement se décompose en 2 modules :
Drug Design 1 (commun avec M1BI) (3 ECTS)
Responsable : G. MOROY
Enseignants : J.L. FAULON & G. MOROY
Objectif :
La conception de médicaments ou drug design nécessite de plus en plus l’utilisation d’outils
informatiques pour optimiser le temps et les coûts liés à la découverte de nouvelles molécules
thérapeutiques. L’objectif de cet enseignement est de présenter les bases théoriques, les algorithmes et
les programmes utilisés pour mener à bien des recherches en drug design. En particulier les approches
chemoinformatiques qui concernent l’étude des molécules potentiellement thérapeutiques et les
approches bioinformatiques basées sur la structure de la protéine ciblée.
Programme :
*Représentation et manipulation des structures chimiques.
*Les descripteurs moléculaires pour le criblage virtuel et les relations structure activité et structure
propriété.
*Introduction à la conception de médicaments par des approches basées sur la structure de la protéine
ciblée (docking, criblage virtuel, flexibilité du récepteur).
*Structure des protéines.
*Outils online aidant à la conception de médicaments.
Modalités d’évaluation : 40 % de contrôle continu (TP), 60 % analyse d’article.
Nombre d’heures :
Heures de cours : 10 heures
Heures de TD : 20 heures
Drug Design 2 : (2 ECTS)
Responsable : O. TABOUREAU
Enseignants : O. TABOUREAU & J.L. FAULON
Objectif :
L’objectif de ce module est de proposer des outils qui peuvent prédire à l’avance les possibles
problèmes d’ADMET ((Administration-Distribution-Métabolisme-Excretion-Toxicité)
associés à une petite molécule. De tels outils peuvent permettre d’optimiser le design d’une
molécule et de limiter ou d’éviter les effets secondaires ou toxiques associés à celui-ci. Un
second point sera de décrire les procédures et les paramètres à prendre en considération pour
convertir les structures de petites molécules entre les différents formats (SMILES, 2D, 3D
formats). Le dernier point abordé sera celui des problèmes d’énumération des structures
chimiques et des réseaux d’interaction.

4
Programme :
*Description des propriétés ADMET
*Utilisation d’outils pour prédire les sites de métabolisation, les groupes d’atomes réactifs et
toxiques, les cibles protéiques associées à des effets secondaires.
*Utilisation d’outils afin de convertir les structures de molécules 2D en 3D.
*Utilisation du logiciel MarvinSketch.
Modalité d’évaluation : Projet: évaluation des risques potentiels associés à une molécule et
comment optimiser le design de cette molécule.
Nombre d’heures :
Heures de cours : 11 heures
Heures de TD/TP : 15 heures
Semestre 2 Bioinformatique Structurale & Drug Discovery
30UCIS42- PROTEIN DOCKING (4 ECTS)
Responsables : J. FERNANDEZ-RECIO, A-C. CAMPROUX, D. FLATTERS
Intitulé : Protein docking
Enseignant: J. FERNANDEZ-RECIO
Programme :
Prédiction des interactions protéine. Prédiction des interfaces Protéine-protéine. Docking protéine-
protéine. Méthodes de calcul pour définir des fonctions de score et d’affinité. Modélisation de la
flexibilité des association protéine-protéine. Identification de hot-spot de liaison dans la conception
des médicaments. Approches multi-docking moléculaire.
Compétences visées :
Les étudiants apprendront l’état de l'art des méthodes de calcul pour la prédiction des interactions
protéine par des simulations de docking -amarrage. Ils seront en mesure d'approche des problèmes
d'amarrage réelles de protéines et d'utiliser les méthodes disponibles et les serveurs Web pour la
modélisation de la structure de complexe protéine-protéine à partir de sous-unités non reliées. Ils
apprendront à intégrer des informations provenant du docking, de données de mutation, de
conservation de séquence et de prédiction de site de liaison.
Modalités d’évaluation : en cours
Nombre de crédits et coefficient de l’ECUE : 4 crédits, coefficient 1
Nombre d’heures :
Heures de cours : 15 heures
Heures de TP : 20 heures

5
Semestre 2 Bioinformatique Structurale & Drug Discovery
30UDIS42 - PROTEIN BIOPHYSIC (5 ECTS)
Responsables : O. TABOUREAU, D. FLATTERS
Programme:
Cet enseignement se décompose en 2 modules :
a) - Module Modélisation moléculaire (en commun avec M1BC2T) (3 ECTS)
1ère partie : Bases de modélisation moléculaire
Enseignants : C. ETCHEBEST, C. MAYER, D. FLATTERS
Objectif :
L’objectif de cet enseignement est de fournir les bases indispensables pour comprendre les principes
gouvernant les techniques de modélisation moléculaire. Ces techniques sont utilisées lors des étapes
d'affinement des structures élaborées par les techniques biophysiques classiques (RX, RMN). Seront
décrites les approches permettant le calcul théorique de différentes propriétés physico-chimiques
étudiées expérimentalement.
Programme :
• Champ de forces semi-empirique. Mécanique Moléculaire
Description des forces debase
Potentiels harmoniques (ressort). Interaction électrostatique
Forces de packing et interaction van der waals
Détermination des paramètres de champ de forces
Minimisation d’énergie et méthodes d’exploration de l'espace conformationnel
• Méthodes de simulation :
Introduction
Dynamique Moléculaire
Calculs de propriétés différentes
Mesures de quantité dynamique
Estimation des erreurs
2ème partie : Méthodes biophysiques d’étude des structures de macromolécules
Enseignant : C. MAYER
Programme : (en cours d’élaboration)
b) - Module Chemical Biology (2 ECTS)
Enseignant : O. TABOUREAU
Objectif :
L’objectif de ce module est de fournir une vue générale de l’application de la chemical biology dans la
recherche de nouveaux médicaments à travers le développement et le criblage de bases de données
chimiques contenant des informations biologiques caractérisées au niveau moléculaire, cellulaires et
phénotypique. Seront décrites les bases de données accessibles publiquement, les approches
permettant d’effectuer des recherches dans ces bases ainsi que les principes pour développer une base
de donnée sous MySQL.
Programme :
* Description de bases de données chimiques contenant des assai biologiques (moléculaire, cellulaire,
phénotypique)
* Découverte et exploration de bases de données de chemical biology
* Description des concepts et de l’architecture pour le développement d’une base de donnée sous
MySQL
 6
6
 7
7
 8
8
1
/
8
100%