Comment deux gènes orphelins du bactériophage T4 inhibent la

1
Comment deux gènes orphelins du bactériophage T4
inhibent la croissance bactérienne ? Une approche
phénomique.
Bony Juliette, Gymnase d’Yverdon-les-Bains.
Supervision : Garama Yonathan, Unige.
Responsable du laboratoire : Professeur Dominique Belin.

2
Fig.2: sensibilité aux
antibiotiques
Résumé :
Ce projet avait pour but d’identifier certains ORFans dont l’expression inhibe la croissance bactérienne
et de déterminer leurs cibles moléculaires.
Différents tests ont été faits : la sensibilité aux antibiotiques, la sensibilité aux dommages à l’ADN, la
sensibilité aux UV-C et la capacité à pousser en milieu minimum, Mac Conkey et à différentes
températures.
L’expression des gènes nrdC.11 (YG2) et 30.2 (YG3) à un niveau non toxique affecte le métabolisme
de la bactérie. L’expression de ces deux gène est toxique à haut niveau, par contre, YG3 est plus
sensible à la température. Ces tests permettent de mieux appréhender le fonctionnement de la toxicité
de ces gènes. Ainsi, il serait possible de développer de nouveaux agents antibactériens.
Introduction :
Alors que le nombre des génomes séquencés augmente, il est frappant que de nombreux gènes
prédits bioinformatiquement codent pour des protéines sans homologie avec les protéines connue.
Ces gènes orphelins, appelés ORFans, sont particulièrement abondants dans les génomes des
phages, les virus qui infectent des bactéries. Les phages sont les organismes les plus abondants sur
terre, leurs ORFans représente donc un gigantesque réservoir de matériel génétique ni décrit, ni
exploité que l’on a appelé la “matière noire du vivant”.
Le phage T4 est l’archétype d’une large famille de phages caractérisée par une morphologie
commune et par un ensemble conservé de gènes structuraux et de réplication. En plus de ces gènes
« centraux », les génomes des phages de la famille de T4 contiennent de nombreux ORFans qui sont
souvent conservés.
En utilisant 2 ORFans, connus pour être partiellement toxiques pour les bactéries, nous cherchons
une hypersensibilité à différents antibiotiques et agents endommageant l’ADN, ainsi qu’à des
conditions peu favorables à la croissance, afin d’identifier quelle voie métabolique de la bactérie est
potentiellement touchée par l’expression de l’ORFan.
Matériel et méthodes :
Les souches de bactéries utilisées sont des Escherichia coli BW25113
dans lesquelles a été transformé un plasmide nommé pBAD101
contenant ou non les gènes orphelins nrdC.11 (YG2) ou 30.2 (YG3). La
surexpression des gènes a ensuite été induite grâce à l’arabinose, un
sucre qui favorise la transcription depuis le promoteur PBAD du
plasmide.
• Sensibilité aux antibiotiques et aux dommages à l’ADN: 12,5
µl de bactéries ont été mélangés à un top LB (0,75% agar + arabinose +
antibiotique) puis coulés sur une boîte LB agar (1.5% agar + arabinose +
antibiotique). Des pastilles contenant les différents antibiotiques à tester et
agents nocifs ont ensuite été déposés sur le top et les boîtes ont été
mises à 37°C. La zone d’inhibition (de la croissance bactérienne) a été
mesurée après 5 heures et après 16 heures. [1]
Fig.1: Plasmide pBAD101
contenant le gène nrdC.11

3
• Sensibilité aux UV-C: 40 µl de bactéries diluées ont été déposé en ligne droite sur des boîtes
de LB agar. Ces boîtes ont été exposées à 90, 60, 30, 20, 10 et 5 secondes aux UV-C. Ce
travail a été effectué dans la pénombre pour éviter le déclanchement d’autres mécanismes de
réparation dépendant de la lumière visible. Les boîtes ont été incubées toute la nuit à 37°C. [1]
• Capacité à pousser en milieu minimum, Mac Conkey et à différentes températures: des
spots de 4 µl de bactéries à différentes dilutions ont été faits sur des boîtes de milieu
minimum (avec le minimum de nutriments essentiel pour le développement des bactéries +
différents sucres + antibiotique), LB (milieu riche) et Mac Conkey (contient sels biliaires). Puis
les boîtes ont été incubées toute la nuit à différentes températures (25°C, 30°C, 37°C, 42°C et
44°C). [1]
Résultats :
La figure 3 représente des spots
placés à différentes températures
pendant plusieurs heures. Nous
pouvons voir que YG3 pousse moins
bien que YG2 qui lui-même ne pousse
pas beaucoup à 44°C, ils sont donc
sensibles à la température.
La figure 4 représente également des spots sur
milieu minimum (+glycérol /+glucose) ou Mac
Conkey, mis à une température de 37°C pendant
plusieurs heures. Nous voyons que YG2 et YG3
ne poussent pas en milieu minimum + glycérol,
alors qu’ils poussent en milieu minimum +
glucose.
La figure 5 montre des O/N dilués à 1/500
étalés sur une boîte LB agar + arabinose,
exposés aux UV-C pendant 0, 5, 10, 20, 30,
60 et 90 secondes. YG3 semble peut-être
légèrement plus sensible aux UV-C
(densité). Comme vu sur le schéma de la
page 1, les UV-C provoquent des
dommages à l’ADN : deux bases azotées
(des pirimidines) vont se lier avec des
liaisons covalentes au lieu de liaisons
hydrogène.

4
La figure 6 présente les résultats de l’hypersensibilité aux dommages à l’ADN. Nous pouvons
constater que YG2 et YG3 sont clairement plus sensibles à l’H2O2 (eau oxygénée) et peut-être à l’HU
(hydroxyurée). Alors que YG3 est peu sensible au HU et après 16 heures, il n’y a plus de zone sans
bactéries. Toujours en référence à la page 1, l’eau oxygénée cause des dommages à l’ADN et
l’hydroxyurée inhibe la synthèse des nucléotides.
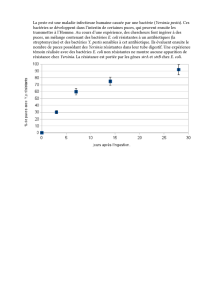
Les deux graphiques de la figure 7 ci-contre montrent les résultats de la sensibilité aux antibiotiques.
Selon le graphique, YG2 est moins sensible au triméthoprime que le contrôle (pBAD101), mais pour
YG3 c’est difficile de dire puisqu’à 5 heures il est plus sensible que pBAD101 mais à 16 heures, il l’est
moins. Nous pouvons voir qu’après 16 heures toutes les aires du contrôle (pBAD101) sont plus
grandes qu’après 5 heures sauf pour la kasugamicine. Cela montre que son effet est moins fort que
les autres antibiotiques. Comme nous l’avons vu sur le schéma de la page 1, le triméthoprime inhibe
la formation d’acide folinique à partir de l’acide folique.

5
Discussion :
L’expression des gènes nrdc.11 et 30.2 est toxique à haut niveau. Une expression plus faible est non
toxique mais elle rend les bactéries plus sensibles, à la chaleur (figure 3) et à l’eau oxygénée (figure
6), incapables de pousser sur milieu minimum (figure 4) mais aussi plus résistantes au triméthoprime
(figure 7). Cela prouve que ce sont de bons moyens pour lutter contre les bactéries. L’expression des
gènes nrdc.11 et 30.2 à un niveau non toxique affecte spécifiquement le phénotype d’Escherichia coli.
C’est une indication qu’ils affectent le métabolisme de la bactérie.
Une hypothèse possible serait qu’en combinant plusieurs facteurs cela améliorerait encore plus l’effet
antibactérien (chaleur + eau oxygénée). Une autre hypothèse, pour explique les résultats de la figure
4, serait que la bactérie a plus besoin de glucose que de glycérol pour pouvoir pousser ce qui
expliquerait que sur la figure 4 le milieu minimum + glucose soit intacte mais le milieu minimum +
glycérol soit touché.
Ces résultats permettront de mieux comprendre le mécanisme expliquant cette toxicité et
potentiellement de découvrir de nouvelles cibles pour des agents antibactériens.
Toutes les expériences seraient à refaire si on voulait des résultats plus précis et pour pouvoir vérifier
les résultats obtenus.
Références :
[1] Mattenberger, Y., Mattson, S., Métrailler, J., Silvia, F. and Belin, D. (2011), 55.1, a gene of
unknown function of phage T4, impacts on Escherichia coli folate metabolism and blocks DNA repair
by the NER. Molecular Microbiology, 82: 1406-1412. doi:10.1111/j.1365-2958.2011.07897.x
[2] Alert to biologists: Ribosomes can translate the ‘untranslated region’ of messenger RNA. phys.org
[3] Eiserling, F.A., Black, L.W. (1994) Pathways in T4 Morphogenesis. In: Karam JD, editor. Molecular
biology of bacteriophage T4. Washington, D. C.: ASM press. P. 209.
Remerciements :
Je remercie Yonathan Garama pour m’avoir laissé prendre part aux expériences et pour m’avoir bien
suivit pendant toute la semaine ; Yves Mattenberger pour l’organisation de ce projet ainsi que pour
son savoir et son aide lors des manipulations ; La Fondation Ernst et Lucie Schmidheiny ; Professeur
Dominique Belin qui m’a accueilli dans son laboratoire ; le département de pathologie du CMU ;
l’université de Genève et finalement « la science appelle les jeunes » pour avoir organisé ce projet.
1
/
5
100%