Protocole avec Annexes 1 et 2 - Collège Lionel

101-NYA-05 Évolution et diversité du vivant Automne 2016
Page 1 sur 10
Département de biologie Collège Lionel-Groulx
1. OBJECTIFS
1.1 Effectuer une analyse d’ADN dans le but d’établir une relation entre les génotypes et les phénotypes d’un
caractère héréditaire : la capacité de goûter une substance amère, le PTC.
1.2 Analyser cette relation pour déterminer le type de dominance, complète ou incomplète, existant entre les
allèles goûteur et non goûteur de PTC.
1.3 Se familiariser avec des techniques modernes de biologie moléculaire : amplification en chaîne par
polymérase ou ACP (couramment appelée PCR, de l’anglais) et électrophorèse sur gel d’agarose.
1.4 Analyser des résultats en tenant compte des variables non contrôlées et inévitables dans une
expérimentation sur du vivant.
PRÉPARATION AU LABO
Semaine 1:
Lire le protocole et compléter l’Annexe 2 à l’aide de la section «Détermination du génotype» (p.6-7).
Semaine 2:
Formuler une hypothèse répondant à la question expérimentale (p.2). Référence : «Génome gourmand» [1] .
Identifier les génotypes attendus à la Fig.7 intitulée «Électrophorèse : résultats attendus», p.5.
2. INTRODUCTION
2.1 LE GOÛT, C’EST D’ABORD UNE AFFAIRE DE GÈNE. Aimez-vous le brocoli? Le PTC?
Un peu d’histoire. Au début des années 1930, Arthur Fox, un chimiste à l’emploi de la Cie DuPont synthétise
une poudre de phénylthiocarbamide (PTC). Alors qu’il en transfère dans une bouteille, des poussières
s’échappent dans l’air et des collègues se plaignent de leur goût amer. Pourtant Fox n’en goûte rien. Quelques
années plus tard, des généticiens démontrent que la capacité de goûter le PTC est un caractère héréditaire et
qu’en Amérique environ 75% des gens sont goûteurs et 25% non goûteurs [2].
PTC et brocoli. Chez les mammifères, on connaît environ 30 gènes codant pour des
récepteurs protéiniques du goût amer sur la langue. Ont-ils joué un rôle évolutif en
permettant de reconnaître des molécules dangereuses souvent associées au goût amer
(ex : strychnine)? Pourtant, certains aliments sains et anti-cancer contiennent beaucoup
d’isothiocyanates dont la saveur amère se rapproche de celle du PTC (ex : les crucifères :
chou, brocoli, chou de Bruxelles, navet). On a même pu établir un lien chez des enfants
entre leur dédain pour le brocoli et leur capacité à goûter le PTC. Évidemment, rendus au
cégep, tous adorent le brocoli! Mais qu’en est-il du PTC?
Lab 6 ANALYSE GÉNÉTIQUE
DE
LA CAPACITÉ DE GOÛTER L’AMER.
Sécurité au laboratoire
Sarrau
Le port du sarrau est obligatoire.
Lampe
UV
L’exposition aux UV est dommageable pour la peau et les yeux. Utiliser avec verre
protecteur.
Rappel
Comme toujours, il est interdit de manger ou de boire au laboratoire.
Produit
toxique
L’ADN est coloré avec un produit potentiellement cancérigène. Tout objet entrant en
contact avec ce produit sera manipulé avec des gants et suivant un protocole très précis.

101-NYA-05 Évolution et diversité du vivant Automne 2016
Page 2 sur 10
Département de biologie Collège Lionel-Groulx
Un gène pour le PTC. Un gène pour le récepteur de PTC a été identifié en 2003 et nommé TAS2R38. Il mesure
1002 pb (paires de bases). Il existerait 7 allèles dans le monde (allèles = formes différentes d’un gène), mais
seuls 2 d’entre eux sont répandus en Amérique : un allèle goûteur et un allèle non goûteur qui diffèrent par 3
mutations aux positions 145, 785 et 886. Dans ce labo, seule la mutation 145 sera utilisée pour distinguer les
allèles goûteur avec un C et non-goûteur avec un G (Fig.1).
Fig.1. Différences entre les deux principaux allèles du gène TAS2R38.
On soupçonne que notre sensibilité au PTC soit sous contrôle polygénique, c’est-à-dire que plusieurs paires de
gènes seraient impliquées. Cependant, le gène TAS2R38 serait le principal responsable des variations observées
dans la population [2].
Le goût, pas juste une affaire de gène… La perception des saveurs est très complexe et plusieurs facteurs
interviennent, tels que l’influence de l’odorat, les mécanismes cérébraux, les différences culturelles, ou
simplement le nombre de papilles gustatives qui peut varier d’un individu à l’autre! Le sexe pourrait aussi
intervenir puisque c’est souvent chez les femmes qu’on trouve des «super goûteuses» [3].
2.2 GÉNOTYPE VS PHÉNOTYPE.
Nous venons au monde avec un bagage de gènes donnés, c’est notre génotype [4;p.303]. Les gènes sont reçus
en deux versions, l’une maternelle, l’autre paternelle. Lorsqu’un individu reçoit 2 allèles identiques du gène
TAS2R38, on dit qu’il est «homozygote». Le caractère héréditaire observé, le «phénotype», est bien sûr celui
porté par les allèles : l’individu est goûteur ou non-goûteur. Mais quel sera le phénotype d’un individu
«hétérozygote», c'est-à-dire un individu qui hérite de deux allèles différents?
Dans un cas de dominance complète, l’hétérozygote présente un phénotype correspondant à celui de
l’allèle dit «dominant». L’allèle qui ne se manifeste pas est dit «récessif».
S’il s’agit d’un cas de dominance incomplète, l’hétérozygote présente un phénotype intermédiaire entre
ceux des homozygotes [4;p.309]; il sera alors semi-goûteur ou «goûteur faible».
2.3 QUESTION EXPÉRIMENTALE.
Puisque nous possédons maintenant les moyens techniques d’analyser notre ADN et nos gènes, nous pouvons
énoncer la question expérimentale suivante : l’interaction entre les deux allèles se rapproche-t-elle d’une
dominance complète ou incomplète? En termes de résultats attendus, la question peut s’énoncer ainsi :
Les élèves hétérozygotes possédant un allèle goûteur et un allèle non-goûteur du gène TAS2R38 auront-ils un
phénotype «goûteur fort» de PTC, «non-goûteur», ou un phénotype intermédiaire «goûteur faible»?
2.4 HYPOTHÈSE … à rédiger après lecture de l’article «Génome gourmand» [1] disponible à l’Annexe 3.
Rédigez une hypothèse et justifiez-la à l’aide de l’article «Génome gourmand» [1]. Indiquer le type de
dominance attendu, le phénotype attendu pour un hétérozygote et citer un passage de l’article à l’appui.
Une des 23 paires
de
CHROMOSOMES
ALLÈLE Goûteur
CCA
GCA
145
ALLÈLE Non Goûteur
3 MUTATIONS:
785
886
1
1002pb
2 Allèles du gène TAS2R38 (1002 pb)

101-NYA-05 Évolution et diversité du vivant Automne 2016
Page 3 sur 10
Département de biologie Collège Lionel-Groulx
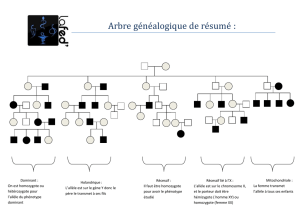
2.5 DÉMARCHE GÉNÉRALE PROPOSÉE
Au cours de ce labo, nous tenterons de répondre à la question posée en suivant la démarche suivante (Fig.2):
1) D’abord une dégustation de PTC, pour déterminer le phénotype de chacun. Qui est goûteur? Non-goûteur?
Y-a-t-il des «goûteurs faibles»?
2) Ensuite une analyse d’ADN, pour déterminer le génotype de chacun : homozygote ou hétérozygote? (un
seul élève expérimentateur par équipe)
3) Enfin, une analyse croisée des résultats pour déterminer le type de dominance, complète ou incomplète,
selon le phénotype observé chez les hétérozygotes. Les homozygotes, goûteurs et non-goûteurs,
constituent un groupe témoin servant à valider la méthode d’analyse, puisque leur phénotype devra
nécessairement correspondre à leur génotype, en toute logique.
Fig.2. Résumé de la démarche générale proposée.
Homozygote
Goûteur
Hétérozygote
Homozygote
Non-goûteur
1o EXTRACTION D’ADN
de cellules buccales
2o AMPLIFICATION :
Ciblage et multiplication
de fragment du gène
3o DIGESTION :
Coupure des allèles goûteurs
pour les différencier des non-
goûteurs
4o ÉLECTROPHORÈSE : séparation par bandes selon leur grosseur
Allèle Goûteur
C
2) DÉTERMINATION DES
GÉNOTYPES
C
G
G
G
+
2X
2X
C
+
C
Goûteurs-
faibles?
1) DÉTERMINATION DES
PHÉNOTYPES
DÉGUSTATION de
PTC
Goûteurs
forts?
Non-
Goûteurs?
3) ANALYSE GÉNOTYPES VS PHÉNOTYPES
Y-a-t-il une forte corrélation entre les génotypes et les phénotypes observés?
La relation entre les allèles du gène TAS2R38 est-elle un exemple de
dominance complète ou de dominance incomplète?
+
C
?
Allèle Non-goûteur
G

101-NYA-05 Évolution et diversité du vivant Automne 2016
Page 4 sur 10
Département de biologie Collège Lionel-Groulx
2.6 DÉMARCHE DÉTAILLÉE DE L’ANALYSE D’ADN (Consulter la «Vue d’ensemble», Annexe 1)
I. EXTRACTION D’ADN.
Des cellules buccales sont d’abord recueillies par rinçage vigoureux avec une solution saline.
L’ADN est ensuite extrait des cellules par ébullition en présence d’une résine; celle-ci sert à éliminer les cations
bivalents (ex : Ca++) nuisibles à l’enzyme «ADN polymérase» utilisée à la prochaine étape.
II. AMPLIFICATION DE L’ADN PAR ACP. (Consulter Campbell Biologie, fig. 20.8).
Objectifs. L’amplification en chaîne par polymérase (ACP) nous permet d’atteindre 2 objectifs :
1o Cibler une région précise de l’ADN : ici la région de la mutation 145 du gène PTC.
2o Faire de multiples copies (ou «réplicons») de cette région pour pouvoir l’analyser.
Chez un homozygote, toutes les copies obtenues seront identiques;
Chez un hétérozygote, on aura 2 types de copies qui diffèrent par une seule lettre, C ou G
Étapes. L’ACP s’effectue dans un thermocycleur (petit four à
température programmable) en 3 étapes (Fig.3):
1o Dénaturation à 94oC : à cette température, l’agitation moléculaire
l’emporte sur les liaisons H de l’ADN et les brins d’ADN se séparent.
2o Hybridation à 68oC; l’agitation diminue et les brins d’ADN tendent
à se lier, mais les amorces, plus concentrées, prennent de vitesse l’ADN
chromosomique. Elles se fixent au début et à la fin de la région ciblée
grâce à la complémentarité des bases azotées. Pour cibler la région de
la mutation 145, nous utilisons une «amorce-début» de 44pb,
complémentaire à la région 101-144 du gène, et une «amorce-fin» de
24pb, complémentaire à la région 297-320 (Fig.4)
3o Élongation à 72oC; à partir des amorces, l’enzyme ADN polymérase
Taq ajoute les nucléotides complémentaires. Cet enzyme est extrait de
la bactérie Thermophilus aquaticus vivant dans des eaux thermales et
il est activé à la température de 72oC.
Chacune de ces 3 opérations ne dure que 30 secondes mais elles sont
répétées plusieurs fois, de telle sorte qu’au bout de 30 cycles on
obtient 1 073 741 824 réplicons de la région ciblée : la région 101 à 320
du gène PTC, mesurant 220 pb (Fig.4). Attention, un hétérozygote
présentera deux types de réplicons qui diffèrent uniquement à la position 145!
Fig.4. Régions ciblées sur le gène PTC par l’amorce début et l’amorce fin.
ALLÈLE GOÛTEUR
101
144
145
5’ ....CCTTCGTTTTCTTGGTGAATTTTTGGGATGTAGTGAAGAGGCAGC................................................................................................................ 3’
5’ CCTTCGTTTTCTTGGTGAATTTTTGGGATGTAGTGAAGAGGCGG 3’
I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I
3’ .....................................................................................................................................................................CTACTAACGTTTGGTTCGGTTGGA... 5’
Amorce début
I I I I I I I I I I I I I I I I I I I I
3’ CTACTAACGTTTGGTTCGGTTGGA 5’
Amorce fin
297
320
ALLÈLE NON-GOÛTEUR
3’ ....................................................................................................................................................................CTACTAACGTTTGGTTCGGTTGGA.... 5’
5’ CCTTCGTTTTCTTGGTGAATTTTTGGGATGTAGTGAAGAGGCGG 3’
I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I I
I I I I I I I I I I I I I I I I I I I I
3’ CTACTAACGTTTGGTTCGGTTGGA 5’
145
5’ ....CCTTCGTTTTCTTGGTGAATTTTTGGGATGTAGTGAAGAGGCAGG.............................................................................................................. 3’
1o Dénaturation
2o Hybridation
3o Élongation
Amorce fin
Polymérase
Amorce début
94oC
68oC
72oC
Fig.3. Étapes de l’ACP [5]

101-NYA-05 Évolution et diversité du vivant Automne 2016
Page 5 sur 10
Département de biologie Collège Lionel-Groulx
Astuce de l’amorce-début.
Normalement, les nucléotides d’une
amorce sont identiques à ceux du gène. Ici,
l’amorce-début diffère à la position #43 :
elle contient un G au lieu d’un A! Cette
«erreur» vise à introduire le doublet GG
devant la mutation 145. Ainsi, les réplicons
obtenus par ACP contiendront en
positions 143-146 les séquences GGCC
pour l’allèle goûteur ou GGGC pour le non-
goûteur (Fig.5). Ces deux séquences sont
essentielles au bon déroulement de la
prochaine étape.
III. DIGESTION DE L’ADN.
Objectif : couper l’allèle goûteur de manière à pouvoir le distinguer de l’allèle non-goûteur.
Les réplicons sont incubés en présence de l’enzyme de restriction HaeIII (extrait de la bactérie Haemophilus
aegypticus). Cet enzyme coupe l’ADN par hydrolyse au milieu d’une séquence GGCC. Cette séquence se trouve
aux positions 143-146 de l’allèle goûteur! Ce dernier est alors coupé en 2 fragments de 44pb et 176pb (Fig. 6).
Par contre, l’allèle non-
goûteur présente la séquence
GGGC et ne peut être coupé
par l’enzyme.
NB. En guise de groupe
témoin, des réplicons ne
seront pas traités avec
l’enzyme.
IV. ÉLECTROPHORÈSE. (Campbell Biologie, fig. 20.9)
Objectif : séparer et visualiser les deux types d’allèles.
Les fragments d’ADN sont placés dans un gel d’agarose et soumis à un courant électrique. Puisque l’ADN porte
des charges négatives (-), les fragments sont entraînés vers l’électrode positive (+) et ce, à différentes vitesses,
les plus petits se déplaçant plus rapidement à travers le gel. Une fois la migration terminée, un colorant
fluorescent sous UV permet de visualiser les bandes d’ADN (Fig. 7).
La position des fragments est évaluée à
l’aide d’un marqueur comportant des brins
de dimension connue; la bande de 500pb est
plus brillante et sert de repère. L’allèle non-
goûteur n’est pas coupé et ne présente
qu’une seule bande de 220pb, alors que
l’allèle goûteur est coupé et présente 2
bandes distinctes de 176pb et 44pb.
Sur la fig.7, quels sont les génotypes des
individus 1-2-3 : hétérozygote, homozygote
goûteur ou homozygote non-goûteur?
1D : ____________________ 2D : ____________________ 3D : ____________________
44 pb
176 pb
220 pb
3 GÉNOTYPES POSSIBLES
3D
N : contrôle (réplicons Non Digérés)
D : réplicons digérés
500 pb
3N
2D
2N
1D
1N
400 pb
220 pb
300 pb
100 pb
176 pb
44 pb
(-)
200 pb
(+)
MARQUEUR
Fig 7. Électrophorèse : résultats attendus
101
3’ ......................................................................................................................................... 5’
3’ ......................................................................................................................................... 5’
ALLÈLE Goûteur
Amorce début
5’ ....CCTTCGTTTTCTTGGTGAATTTTTGGGATGTAGTGAAGAGGCAGCC................ 3’
145
5’ CCTTCGTTTTCTTGGTGAATTTTTGGGATGTAGTGAAGAGGCGGCC 3’
143
Gène
Gène
ALLÈLE Non-Goûteur
Amorce début
5’ CCTTCGTTTTCTTGGTGAATTTTTGGGATGTAGTGAAGAGGCGGGC 3’
145
5’ ....CCTTCGTTTTCTTGGTGAATTTTTGGGATGTAGTGAAGAGGCAGGC............... 3’
143
Fig.5. Différence entre l’amorce-début et le gène en position 143.
Fig.6. Hydrolyse enzymatique de l’allèle goûteur.
145
+
101
320
fragment de 176 PB
réplicon de 220 PB de l’allèle goûteur
fragment de 44 PB
5’ CC……..................................................... 3’
3’ ………………………………………………. 5’
5’ …………………….…GG 3’
3’ ……………………….….. 5’
5’ ..................................GG CC…….................................................... 3’
3’ ……………………………………………………………………… ……..5’
Fig.6. Digestion enzymatique de l’allèle goûteur.
 6
6
 7
7
 8
8
 9
9
 10
10
1
/
10
100%