05-4 Bouchez - INRA Versailles

BIOFUTUR 265 • AVRIL 200638
Dossier
Les atouts de la génomique végétale
L
a génomique fonctionnelle, discipline récente qui
doit beaucoup aux progrès de l'instrumentation,
vise à une compréhension fine des fonctions des
gènes et des protéines impliqués dans les pro-
cessus biologiques à l'œuvre dans les organismes
vivants. Elle repose sur la mise en œuvre de méthodes
à haut débit (les fameux « -omiques ») et de niveaux
d’analyse variés (du gène à la population).
L
e séquençage de génomes complets, comme celui
d'Arabidopsis thaliana, a permis de mesurer l'ampleur
de la tâche : des informations fonctionnelles fiables
ne sont disponibles que pour à peine un cinquième des
26 000 gènes repérés. Pour les plantes comme pour la
plupart des organismes supérieurs pour lesquels on
dispose d'informations génomiques détaillées, l'étude
de la fonction des gènes et des protéines fait appel à
des approches de génétique (mutagenèse ciblée ou aléa-
toire, analyse des phénotypes résultants), mais aussi
de transcriptomique (régulation de la transcription),
de biochimie des protéines et d'imagerie cellulaire
(voir
article suivant)
.
U
n grand volet des approches de génomique fonc-
tionnelle, en particulier celles menées sur A. thaliana
et les modèles génériques, concerne en effet la géné-
tique. Jusqu’aux années 90, il était particulièrement
difficile d'isoler un gène repéré sur la base de sa loca-
lisation sur une carte génétique. Toutefois les approches
de clonage positionnel disponibles aujourd’hui ont fait
de grands progrès par rapport à cette époque. Elles
demeurent, encore à l'heure actuelle, délicates à mettre
en œuvre, mais sont théoriquement applicables dans
n'importe quelle espèce où il est possible de réaliser de
la cartographie génétique ; ces techniques sont d'un
intérêt considérable pour l'identification de gènes
importants chez les espèces végétales, en particulier
chez les plantes cultivées où elles constituent souvent
le seul moyen d'identification disponible.
C
hez les plantes supérieures comme le maïs, le muflier
ou Arabidopsis, la mise au point de techniques de muta-
genèse d’insertion a permis dès le début des années 90
de proposer une alternative aux approches de clonage
positionnel. En raison de l’absence de technique de
ciblage génique par recombinaison efficace chez les
plantes supérieures, les collections de mutants d’in-
sertion sont également largement exploitées en géno-
mique fonctionnelle pour l'identification de fonctions
géniques. On parle alors de génétique « inverse » :
contrairement aux approches classiques dans lesquelles,
à partir d'une fonction identifiée sur la base d'un crible
phénotypique, on cherche à arriver aux gènes et aux
protéines impliquées, il s'agit, à partir de la connais-
sance du génome et de l'identification des gènes, d'iso-
ler des mutants et d'en analyser les modifications.
P
lus récemment, les approches de crible de populations
de mutants classiques (voir tilling) ou l'utilisation d'ap-
proches basées sur l'interférence ARN (ARNi) ont per-
mis de diversifier la palette des outils de mutagenèse
et d'élargir le spectre des mutations utilisables.
Clonage positionnel
de gènes d’intérêt agronomique
P
armi les caractères observables sur les plantes, cer-
tains ont un impact important en agriculture (préco-
cité, résistance à un pathogène…). On parle alors de
Nouvelles approches génétiques
La génomique s'intéresse aux grands principes qui régissent la structure
globale et l'évolution des génomes, mais aussi au fonctionnement
coordonné des milliers – voire des dizaines de milliers chez les plantes
supérieures – de gènes et de protéines qu'ils spécifient. Au-delà des
approches de génomique structurale (cartographie physique et
génétique, inventaire de gènes, séquençage…), les approches de
génomique fonctionnelle ont investi la plupart des champs de la
biologie végétale actuelle.
David Bouchez*, Abdelhafid Bendahmane**,***, Claire Lurin**,****, Bénédicte Sturbois**,*****
* Station de génétique
et amélioration des plantes,
Institut Jean-Pierre Bourgin
Inra, Centre de Versailles,
78026 Versailles Cedex
** Unité de recherche
en génomique végétale (URGV)
UMR Inra 1165 - CNRS 8114 -
Université d'Évry
2 rue Gaston Crémieux,
CP 5708, 91057 Évry Cedex
**** [email protected]
***** [email protected]
05-4 Bouchez 16/03/06 9:46 Page 38

BIOFUTUR 265 • AVRIL 2006 39
caractères agronomiques. Un caractère agronomique
peut être déterminé entièrement par un seul locus, com-
munément appelé « gène majeur » (déterminisme men-
délien), ou par plusieurs loci à effet partiel sur la varia-
tion du caractère, on parle alors de QTL
*1
. Des
marqueurs génétiquement liés à de tels loci permettent
de sélectionner, parmi un grand nombre de plantes, les
individus les plus performants. Ils peuvent ainsi être
utilisés pour sélectionner un nouveau génotype inté-
ressant par croisements successifs.
L
’identification précise des gènes en jeu et des poly-
morphismes responsables des variations observées est
l’étape ultime qui permet de connaître les bases molé-
culaires de ces caractères agronomiques, d’analyser
leur variabilité dans les populations naturelles et de les
intégrer à des programmes de création variétale.
I
dentifier la séquence d’un gène majeur ou d’un QTL
relève en pratique de deux démarches de clonage dif-
férentes. L’identification moléculaire rapide d’un gène
majeur ou parfois d’un QTL peut reposer sur les tech-
niques d’étiquetage des gènes, lorsqu’elles sont acces-
sibles dans l’espèce étudiée. En l’absence de ce système
d’étiquetage, la stratégie la plus couramment utilisée
est le clonage positionnel
(1)
.
C
e type de clonage repose sur l’identification de mar-
queurs ADN et d’événements de recombinaison dans
la région du locus cible. Les marqueurs sont positionnés
avec précision par rapport aux événements de recom-
binaison pour développer une carte génétique de haute
résolution. La seconde étape consiste à construire une
carte physique de la région, dans laquelle les distances
génétiques
*2
sont converties en nombre de paires de
bases. Quand un fragment d’ADN cloné recouvrant
l’intervalle entre les marqueurs les plus proches a été
identifié, le gène d’intérêt peut être localisé par sous-
clonage et recherche des séquences codantes dans les
sous-clones. La validation fonctionnelle est réalisée par
cartographie fine du gène dans la zone chromosomique
étudiée
(figure 1)
.
Cartographie génétique fine
L
a cartographie fine consiste à rechercher et ordon-
ner des marqueurs localisés dans un très petit inter-
valle autour du locus d’intérêt. La saturation de la
région cible en marqueurs et en événements de
recombinaison constitue un paramètre clef de l’ex-
ploitation de cette stratégie et peut considérablement
accélérer le clonage de gènes. Les techniques de mar-
quage permettant de cribler un grand nombre de mar-
queurs simultanément, comme les AFLP
*3
, sont parti-
culièrement utiles à cette étape
(2)
.
Cartographie physique
L
a construction d’une banque d’ADN génomique est
une étape essentielle pour l’établissement de la carte
physique. Le système BAC
(3)
est le plus couramment
utilisé pour ce type d’approche. La taille des inserts
peut y atteindre 350 kb.
L
a banque BAC est criblée avec les marqueurs les plus
proches du gène d’intérêt. On établit ensuite une carte
locale des clones par une série de digestions enzy-
matiques qui permettent de définir les fragments com-
muns à plusieurs clones et de les ordonner les uns par
rapport aux autres. Un ensemble de clones ordon-
nés constitue un contig.
L
a réussite de la construction de la carte physique
dépend de la qualité de la carte génétique fine du locus
cible. Les extrémités des BAC sont séquencées et de
nouveaux marqueurs sont définis et utilisés pour ancrer
la carte génétique à la carte physique et pour identifier
le clone BAC supposé porter le gène d'intérêt, ou encore
pour allonger la taille des contigs.
Q
uand le clone BAC qui recouvre l’intervalle entre
les marqueurs les plus proches a été identifié, le gène
cible peut être identifié par séquençage et cartographie
des gènes prédits relative aux événements de recom-
binaison. Le polymorphisme des portions codantes
identifiées est étudié, afin de rechercher d’éventuelles
schez les plantes
*1
Quantitative trait locus
*2La distance génétique
reflète la fréquence des
événements de recombinaison
sur la portion d’ADN
considérée. Elle est exprimée
en centimorgans, 1cM = 1 %
de recombinaison.
*3
Amplification fragment length
polymorphism
, marqueurs du
polymorphisme de longueur
des fragments d'amplification.
(1) Rommens JM
et al.
(1989)
Science
245, 1059-65
(2) Vos P
et al.
(1995)
Nucl Acids Res
23, 4407-14
(3) Shizuya H
et al.
(1992)
Proc Natl Acad Sci USA
89,
8794-7
Figure 1 Étapes du clonage positionnel
Localisation primaire de la région d'intérêt par analyse de liaison (quels marqueurs
co-ségrègent avec le phénotype ?)
Assemblage des clones d'ADN génomique (BAC) chevauchants (formation de
contigs
)
Inventaire des gènes présents
Étude des polymorphismes
© D.R.
05-4 Bouchez 16/03/06 9:46 Page 39

BIOFUTUR 265 • AVRIL 200640
Dossier
Les atouts de la génomique végétale
relations entre polymorphisme et variation du carac-
tère. Des constructions plasmidiques, porteuses de ces
portions, sont produites et utilisées pour transfor-
mer des plantes afin de valider leur rôle dans la varia-
tion du caractère par complémentation.
L
e clonage d'un QTL est plus difficile que celui d'un
gène majeur car il n'influence que partiellement la varia-
tion du caractère. Une « mendelisation » du caractère
est nécessaire avant l’établissement du clonage posi-
tionnel. Cette opération consiste à comparer deux géno-
types identiques sauf pour la zone chromosomique étu-
diée, porteuse du QTL à identifier.
Mutagenèse au hasard
Mutagenèse par insertion
U
tilisé chez la souris depuis plusieurs années, le ciblage
génique sur cellules souches embryonnaires n'a permis
d'obtenir des mutants que pour environ 10 % des gènes
de cette espèce. On découvre actuellement les avan-
tages de la mutagenèse insertionnelle pour la génétique
inverse et les cribles génétiques à grande échelle.
C
hez les plantes supérieures, la mutagenèse d'insertion
a été largement utilisée depuis la fin des années 80 pour
l'obtention de mutants, tout d'abord dans le cadre
de cribles phénotypiques, puis à des fins d'analyse fonc-
tionnelle et de génétique inverse.
• Un principe relativement simple
O
n utilise comme agent mutagène un segment d'ADN
de séquence connue, capable de s'insérer de manière
aléatoire dans le génome. L'insertion de ce segment au
niveau d'un gène provoque, dans la plupart des cas,
une perte de fonction. Contrairement aux mutations
classiques induites par des agents physico-chimiques,
la mutation est facilement repérable dans le génome,
« étiquetée » par la présence du segment d'insertion
au locus muté. Comme pour toute approche de muta-
genèse, le paramètre essentiel est le taux de saturation
de la population, c'est-à-dire la probabilité de muter
chaque gène au moins une fois. Pour une population
d'insertion, ce taux dépend de la taille du génome,
de la taille moyenne d'un gène, du nombre moyen d'in-
sertions par lignée, et du nombre total de lignées : pour
Arabidopsis, environ 180 000 insertions aléatoires sont
nécessaires pour atteindre une saturation de 95 %.
Même si ce chiffre peut paraître impressionnant, il reste
comparativement faible par rapport à des génomes où
la densité en gènes est beaucoup plus faible que chez
Arabidopsis.
L
e segment d'insertion porte un gène de résistance sélec-
tionnable, et peut aussi inclure des marqueurs divers
permettant par exemple d'obtenir des informations sur
l'activité transcriptionnelle de la région d'insertion,
par « piégeage » de promoteurs ou d'éléments régu-
lateurs de transcription. L'élément d'insertion peut éga-
lement porter un activateur de transcription qui per-
met le cas échéant « d'allumer » la région d'insertion
au lieu de l'éteindre : on obtient alors des mutations
de type gain de fonction, le plus souvent dominantes
génétiquement.
• Éléments transposables
D
eux grands types d'éléments sont utilisés pour la
mutagenèse insertionnelle chez les plantes. En pre-
mier lieu, les éléments transposables : ces segments
d'ADN, découverts chez le maïs dès les années 40,
ont la propriété intrinsèque d'être mobiles au sein des
génomes. Les rétrotransposons peuvent même consti-
tuer la plus grande partie du génome chez certaines
espèces végétales. Bien qu’en général les éléments
transposables soient naturellement inactifs, on peut
les « réveiller » afin de les utiliser pour la mutagenèse
insertionnelle. Par exemple, le rétroélément de riz
Tos17 est activé par la culture in vitro, et est de nou-
veau inactif dans les plantes régénérées, où on peut
trouver entre 5 et 30 copies nouvelles du rétroélément.
E
n général, les éléments endogènes sont difficiles à uti-
liser tels quels, et on préfère utiliser soit des versions
Clonage positionnel du gène
Rfo
chez le radis
La stérilité mâle cytoplasmique Ogu-INRA, caractérisée chez le radis (
Raphanus sativus
), est
utilisée depuis plusieurs années pour la production de semences hybrides chez les brassicas
(crucifères) cultivées. Son utilisation chez le colza cause de nombreux problèmes dus à la maî-
trise imparfaite du système de restauration. En effet, le gène de restauration
Rfo
a été introduit
dans le colza par croisements interspécifiques. Les lignées de colza restauratrices actuelle-
ment disponibles portent une introgression*1de génome de radis de grande taille ayant rem-
placé environ 50 cM du génome de colza (1). Ces manipulations (introgression et/ou délétion)
s’accompagnant de caractères défavorables (fort taux en glucosinolates, mauvaises perfor-
mances agronomiques, faible fertilité femelle), des efforts intensifs de sélection ont été menés
au cours des dernières années.
Test de complémentation du phénotype mâle stérile.L’expression transgénique de la
PPR B chez le radis mâle stérile (à droite) restaure la production de pollen. À gauche,
fleur non-transgénique mâle stérile.
Carte physique du locus
Rfo
chez le radis. Le clone BAC B64 portant le gène
Rfo
a été
sequencé et annoté.
*1Incorporation de gènes
d’une espèce au sein du
génome d’une autre espèce.
(1) Delourme R
et al.
(1998)
Theor Appl Genet
97, 129-34
(2) Desloire S
et al.
(2003)
EMBO report
4, 588-94
© D.R.
© D.R.
Le clonage du gène
Rfo
a été réalisé chez le radis en collaboration avec Régine Delourme,
Michel Renard (Inra de Rennes) et Françoise Budar (Inra de Versailles) en utilisant la plate-
forme de clonage positionnel d’Évry (figures et photos). À l’issue de ce travail, il a été démon-
tré que le gène
Rfo
code une protéine contenant des motifs PPR (
pentatricopeptide repeat
) (2).
Ce gène appartient à une très grande famille chez les plantes supérieures, avec environ 450
membres répertoriés chez
Arabidopsis thaliana
. La plupart des protéines codées par ces gènes
seraient adressées aux organites cellulaires afin de contrôler l’expression des gènes mito-
chondriaux ou plastidiques.
05-4 Bouchez 16/03/06 9:46 Page 40

BIOFUTUR 265 • AVRIL 2006 41
modifiées faciles à repérer (stratégie RescueMu chez le
maïs par exemple), soit introduire des transposons en
provenance d'autres espèces : divers transposons du
maïs (monocotylédone) ont par exemple été utilisés
avec succès chez Arabidopsis (dicotylédone), comme
ceux de la famille Ac/Ds, ou surtout En/Spm.
D
eux stratégies sont possibles : dans la première, aucun
contrôle n'est exercé sur la transposition, et chaque
lignée porte jusqu'à plusieurs centaines de copies de
l'élément (cas du transposon Mu du maïs). Dans ce
cas, il est relativement aisé de générer un très grand
nombre d'insertions, et l'analyse de plusieurs allèles
mutés indépendants doit théoriquement permettre
de s'affranchir du « bruit de fond génétique » généré
par les autres insertions. La seconde stratégie fait appel
à des dispositifs génétiques sophistiqués pour limiter
le nombre d'insertions. Dans ce cas, l'analyse phéno-
typique est facilitée, et on n'a pas forcément besoin
d'un grand nombre d'allèles mutants pour fiabiliser
les conclusions. De plus, beaucoup de transposons
montrent une certaine « paresse » et s'insèrent préfé-
rentiellement à proximité de leur site de départ, mais
là encore il est possible de mettre en place des dispo-
sitifs permettant de s'affranchir de ce problème.
• ADN-T
L
e second élément mutagène largement utilisé chez les
plantes, tout particulièrement chez Arabidopsis, est
l’ADN-T d'Agrobacterium tumefaciens. Cette bacté-
rie, qui possède la capacité unique de transférer une
portion d'ADN plasmidique appelée ADN-T dans le
génome des cellules végétales qu'elle infecte, est un vec-
teur biologique largement utilisé en transgenèse végé-
tale. L'insertion de l’ADN-T dans le génome nucléaire
est aléatoire et le nombre d'insertions naturellement
limité (une à trois) par événement de transformation,
ce qui en fait un système de choix pour une approche
de mutagenèse par insertion. Cependant, pour atteindre
un nombre d'insertions indépendantes permettant une
saturation satisfaisante, il est nécessaire de disposer
d'une méthode de transformation très efficace, et qui
s'affranchisse si possible des étapes de culture in vitro
et de régénération, toujours susceptibles d'induire des
variations génétiques indésirables.
C
es conditions ne sont remplies à l'heure actuelle que
chez Arabidopsis : une méthode de transformation très
simple, sans passage par la culture in vitro, a été mise
au point dès 1987 par Kenneth Feldmann aux États-
Unis
(4)
. Au début des années 90, des chercheurs ver-
saillais apportent des améliorations majeures à cette
méthode difficile à reproduire. Cette technique de trans-
formation d'Arabidopsis par infiltration a depuis
conquis tous les laboratoires de biologie végétale. Elle
se déroule entièrement en conditions horticoles, non
stériles, et permet d'obtenir facilement et rapidement
des milliers de plantes transformées indépendantes
(photo)
. Avec un tel outil, il devenait possible d'envi-
sager une mutagenèse d'insertion à saturation. Grâce
à ces travaux, de nombreuses populations ont été
construites, pour un effectif global qui doit maintenant
dépasser le million de lignées, largement plus que la
saturation théorique du génome d'Arabidopsis.
M
alheureusement, cette technique de transformation
« in planta » n'a pas pu être transposée à d'autres
modèles végétaux. Pour d'autres espèces chez lesquelles
des programmes de mutagenèse ADN-T sont mis en
place (riz, Medicago), le recours à des techniques de
transformation in vitro, incluant une phase de régé-
nération, s'avère incontournable.
U
ne fois établies, les populations d'insertion peuvent
être utilisées dans le cadre de cribles génétiques clas-
siques, comme toute population mutagénisée, avec un
accès facilité au gène muté. Les populations ADN-T
d'Arabidopsis ont été largement utilisées au fil du temps
pour le clonage de gènes ou la recherche de nouveaux
mutants. Par exemple, beaucoup des gènes impliqués
dans le développement de la fleur ont été isolés par ce
biais, notamment le gène agamous.
C
es populations d'insertions sont également très utiles
pour la génétique inverse et la recherche de mutants pour
des gènes particuliers. Cela peut se faire facilement, par
des méthodes basées sur l'amplification par PCR.
Cependant, dans un souci d'efficacité, l'exploitation des
populations repose désormais essentiellement sur des
programmes d'identification systématique de la totalité
des sites d'insertion au sein d'une population. Pour
chaque lignée, le site d'insertion est identifié par PCR et
séquençage de quelques centaines de nucléotides. La
comparaison avec la séquence génomique (il faut donc
qu’elle soit disponible) permet alors d'identifier la région
concernée, et éventuellement le gène affecté par l'in-
sertion. L'ensemble des informations est intégré dans
des bases de données internationales. Le généticien peut
facilement y sélectionner les mutations dans ses gènes
favoris, et commander les lignées correspondantes dans
les centres de ressources afin d'en étudier le phénotype.
Une partie de la collection de lignées d'insertion ADN-T de
l'Inra de Versailles, en cours de multiplication. Chaque bar-
quette contient une cinquantaine de plantes issues d'une lignée
transgénique indépendante. Les lignées sont examinées atten-
tivement pour la recherche d'altérations phénotypiques.
De plus, l'ADN de chaque famille est prélevé et analysé pour
repérer le site d'insertion de l’ADN-T. Cette information est
ensuite insérée dans les bases de données internationales
de génomique d'
Arabidopsis
.
(4) Feldmann KA, Marks MD
(1987)
Mol Gen Genet
208, 1-9
© D. BOUCHEZ
05-4 Bouchez 16/03/06 14:22 Page 41

BIOFUTUR 265 • AVRIL 200642
Dossier
Les atouts de la génomique végétale
O
n compte à l’heure actuelle 370 000 insertions ADN-T
cartographiées chez Arabidopsis, soit une saturation
théorique de plus de 99,9 %. Cependant, plusieurs mil-
liers de gènes prédits ne possèdent toujours pas d'in-
sertion identifiée. Parmi ceux-ci, il est probable qu'une
bonne partie représente des gènes essentiels ne pou-
vant être atteints par ce type de mutation qui engendre
le plus souvent une perte totale d'activité, incompatible
avec la viabilité des gamètes.
Tilling
L
e tilling (
t
argeted
i
nduced
l
ocal
l
esions
in g
enome,
criblage de mutations induites localement dans le
génome) est une technique de génétique inverse aux
nombreux avantages
(encadré)
. Même si elle a d’abord
été mise en œuvre chez Arabidopsis thaliana
(5, 6)
,
elle est adaptée aux plantes cultivées. En effet, le
génome des plantes cultivées est souvent de grande
taille, complexe et récalcitrant aux méthodes clas-
siques de transgenèse, ce qui le rend inapproprié aux
techniques de mutagenèse d’insertion à grande échelle.
L
e principe du tilling est d’introduire au hasard des
mutations dans le génome de milliers de plantes (grâce
à un produit chimique mutagène) et ensuite de repérer
par une approche moléculaire les plantes renfermant
les mutations recherchées dans un gène d’intérêt.
• Développement de plates-formes
E
n exploitant la technologie du tilling, des plates-formes
de criblage ont donc été mises au point pour les plantes
cultivées telles que le pois, le colza et la tomate, au sein
de l’URGV d’Évry, et pour le soja aux États-Unis. Le
pois et le colza sont deux espèces cultivées de grande
importance en France. Le colza est appelé à un grand
développement pour la production de biocarburants.
Le pois est un substitut direct du soja, massivement
importé des États-Unis pour la nourriture du bétail
(l'Europe importe 68 % de ses besoins en protéines
pour l'alimentation animale).
C
ependant, les outils de génétique inverse basés sur la
mutagenèse insertionnelle ne sont pas applicables à ces
deux plantes cultivées en raison de leur génome de très
grande taille (plus de 40 fois celui d’Arabidopsis thaliana
pour le pois et 10 fois pour le colza) ce qui nécessite-
rait des collections de mutants d’insertion gigantesques
pour effectuer des travaux de génétique inverse. Il était
donc crucial de développer et d’exploiter le tilling pour
ces deux espèces cultivées.
L
es gènes étudiés dans un premier temps chez le pois
sont issus des programmes de recherche de gènes can-
didats. On s’intéresse plus particulièrement aux gènes
impliqués dans le remplissage de la graine chez le
pois protéagineux et à des mutants de pois à florai-
son tardive. Ces mutants sont recherchés chez les
orthologues
*4
des gènes très bien caractérisés dans
l’initiation florale chez Arabidopsis thaliana, tels que
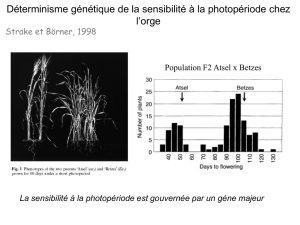
ceux impliqués dans la photopériode (Constans,
Gigantea…) ou dans la vernalisation
*5
comme
Luminidependens, FCA ou FLC
(7, 8)
.
P
our le colza, compte tenu de sa parenté évolutive
avec Arabidopsis thaliana chez qui un grand nombre
de gènes a déjà été caractérisé par knock-out, on s’in-
téresse aux gènes présentant un intérêt agronomique
potentiel. On cible notamment les gènes impliqués
dans la déhiscence des siliques
(9)
. En effet, l’ouver-
ture des siliques chez le colza est à l’origine d’une perte
de rendement annuel de 20 à 50 %. D’autres phé-
notypes d’intérêt, concernant par exemple la teneur
en huile, en glucosinolates
(10)
ou en flavonoïdes, sont
recherchés. La technologie du tilling peut s’appliquer
à des génomes de plantes encore beaucoup plus com-
plexes, par exemple sur des plantes polyploïdes,
puisque des mutants de la production d’amidon dans
les graines de blé qui sont hexaploïdes ont pu être
caractérisés grâce à cette technique
(11)
.
• Une technologie pour tous les organismes
U
ne variante du tilling a été développée pour analyser
le polymorphisme allélique : l’ecotilling. Le criblage
ne se fait plus sur des populations mutagénisées à l’EMS
mais sur des populations naturelles de plantes. Ainsi,
on a directement accès à la variation allélique natu-
relle au sein d’un gène. Cette technologie a été mise en
œuvre chez Arabidopsis thaliana, le pois, la tomate et
le blé. Elle est beaucoup moins onéreuse à mettre en
Principe du
tilling
L’ADN génomique est muté par application sur les graines d’éthylméthane sulfonate (EMS), qui
provoque des mutations ponctuelles au hasard. L’avantage de l’EMS par rapport aux mutations
insertionnelles est que l’on a alors accès à l’analyse de gènes dans lesquels une insertion dans la
séquence codante aurait provoqué un phénotype létal et donc difficile à caractériser. On peut donc
identifier des mutants dont le phénotype est diversement altéré et donc plus susceptibles d’avoir
un intérêt agronomique potentiel. De plus, l’EMS pouvant induire aux doses usuelles plusieurs
mutations par génome, une collection de plusieurs milliers de plantes offre tout un panel de muta-
tions différentes pour un gène étudié, et l’opportunité d’évaluer les conséquences de chacune de
ces mutations au niveau de ce même gène. On peut ainsi mettre en évidence un phénotype plus
ou moins marqué en fonction des mutations et constituer des séries alléliques.
On utilise pour le criblage les plantes M2, issues d’autofécondations des plantes mutagénisées
(M1) : les plantes M1 présentant des mutations sont hétérozygotes, donc une partie des
plantes M2 (1/4 selon les lois de Mendel) sont homozygotes, avec un génome stable. L’ADN
est isolé à partir de chaque famille M2*1et réparti en
pools
(ce qui permet de réaliser moins
de PCR que si les plantes étaient testées individuellement), chacun contenant
a priori
des
plantes mutées et non mutées pour le gène d’intérêt.
On procède ensuite à des PCR sur ces
pools
, en utilisant des oligonucléotides marqués par des
fluorophores. Après l’étape de renaturation, deux types de molécules seront obtenues, les
homoduplexes (deux brins sauvages ou deux brins mutants) et les hétéroduplexes (un brin
sauvage + un brin mutant, donc avec un mésappariement). Une endonucléase reconnaissant
les mésappariements*2(1, 2) clive les hétéroduplexes uniquement, conduisant à l’apparition de
deux fragments de digestion sur gel d’acrylamide (qui sépare les fragments d’ADN selon leur
taille). On peut ainsi localiser le site de coupure au sein du gène d’intérêt sur la base de la lon-
gueur des fragments générés.
Lorsqu’un
pool
contient une mutation d’intérêt, l’expérience est répétée mais uniquement au
sein du
pool
concerné, avec à terme l’identification du plant présentant la mutation.
*1Une famille se compose de
quatre plantes, pour optimiser
les chances d’y trouver un
mutant pour le gène considéré.
*2Même si l’endonucléase
préférentiellement utilisée
jusqu’à présent est CEL1
(purifiée à partir du céleri),
une nouvelle enzyme
plus performante, ENDO1,
a récemment été purifiée
et caractérisée à l’URGV d’Évry.
Elle est d’ailleurs commer-
cialisée par une start-up privée
(www.serialgenetics.com).
(1) Oleykowski
et al.
(1998)
Nucleic Acid Res
26,
4597-602
(2) Till
et al.
(2004)
Nucleic Acid Res
32, 2632-41
© D.R.
05-4 Bouchez 16/03/06 9:46 Page 42
 6
6
 7
7
1
/
7
100%