d. cartographie genetique des maladies : de la

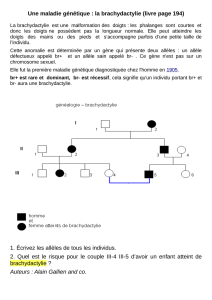
de certains clones cellulaires de ces tumeurs. Dès
1914 T. Boveri associait la notion d’anomalie chro-
mosomique à celle de carcinogenèse. De nom-
breuses aberrations chromosomiques surviennent au
hasard au sein des cellules cancéreuses, témoignant
d’une instabilité génétique. Certains réarrangements
chromosomiques sont cependant particuliers à l’évo-
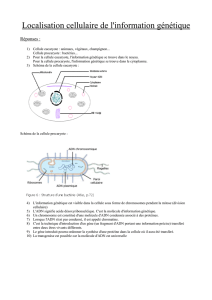
lution de certaines tumeurs. C’est le cas pour le chro-
mosome de Philadelphie qui a été la première ano-
malie chromosomique (translocation entre les chro-
mosomes 9 et 22) décrite comme spécifique de la
leucémie myéloide chronique. Ces altérations ont
permis d’isoler à l’échelle moléculaire des anoma-
lies génétique et leurs conséquences fonctionnelles.
Par exemple dans les translocations de la leucémie
myéloide chronique ou de la leucémie aigue pro-
myélocytaire, les translocations chromosomiques
respectives t(9-22) et t(15-17) entraînent les
séquences codantes de gènes qui fusionnent et
codent ainsi pour de nouvelles protéines issues de
cette recombinaison génétique. Ces protéines recom-
binantes, dite de fusion, acquièrent alors une activité
de type oncogénique.
La nature génétique d’une maladie repose avant tout
sur des arguments épidémiologiques tels qu’une
augmentation de la prévalence chez les apparen-
tés de malades (agrégation familiale) ou dans cer-
tains isolats génétiques, un taux de concordance
élevé chez les jumeaux monozygotes ou un âge de
début plus précoce dans les formes familiales que
dans les formes sporadiques (Fig. 35).
• L’approche classique pour identifier les gènes res-
ponsables des maladies héréditaires partait de la pro-
téine pour retrouver le gène qui détermine sa synthè-
se. Cette démarche est celle qui a prévalue à la
découverte des facteurs de la coagulation impliqués
dans l’hémophilie. Lorsque les gènes furent isolés,
leur position sur le chromosome X fut précisée et
leurs mutations reconnues.
•Dans la démarche actuelle, on part du constat que
la maladie est héréditaire sur des arguments épidé-
miologiques et on cherche à déterminer la localisa-
tion puis la séquence du gène et de ses mutations
afin d’en déduire la protéine ainsi que son rôle dans
la physiopathologie de la maladie. Le processus de
découverte se déroule dans l’autre sens d’où le terme
de “ génétique inverse ” ou de “ clonage position-
nel ”. Cette démarche peut être très performante car
il n’est pas nécessaire de connaître la nature biochi-
mique de l’anomalie qui détermine la maladie. Elle
a permis d’identifier les gènes impliqués dans plu-
sieurs maladies telles que la chorée de Huntington, la
myopathie de Duchenne, la mucoviscidose ou le
vieillissement accéléré du syndrome de Werner.
Elle s’applique idéalement aux maladies monofacto-
rielles à forte pénétrance. Elle est moins performan-
te lorsqu’il existe une importante hétérogénéité
génétique (plusieurs gènes indépendants pouvant
déterminer la même maladie) ou a fortiori lorsqu’il
s’agit d’une maladie multifactorielle [dans ce cas, le
facteur génétique héritable confère une vulnérabilité
relative (susceptibilité ou risque) à la maladie avec
plusieurs gènes impliqués de façon concomitante et
souvent intriqués avec des facteurs non génétiques].
Pour ces maladies, où une composante génétique
existe au sein de différents facteurs de risque, l’ap-
proche dite “ gène candidat ” est souvent choisie.
Dans cette approche, les gènes testés comme candi-
dats sont des gènes déjà connus et pour lesquels on
suppose que la défectuosité pourrait être impliquée
dans le déterminisme de la maladie (Fig. 36).
• L’identification des altérations génétiques
(mutations causales) des maladies repose in fine sur
la mise en évidence de séquences d’ADN particu-
lières au niveau d’un gène déterminé et spécifique-
ment associées au phénotype malade.
Cette approche procède par plusieurs étapes. S’il n’y
a pas de “ gène candidat ”, la première étape consis-
te à localiser sur le génome le gène susceptible d’être
altéré dans la maladie. Pour cela, il faut disposer de
II. COMMENT IDENTIFIER LES
GENES RESPONSABLES ?
I. LA MALADIE A T’ELLE UNE
COMPOSANTE HEREDITAIRE ?
D. CARTOGRAPHIE GENETIQUE
DES MALADIES : DE LA
RECHERCHE À LA CLINIQUE
742

743
Figure 35 : Caractéristique d’une maladie faisant suspecter des facteurs génétiques
Figure 36 : Méthodes pour identifier les gènes impliqués dans les maladies génétiques
COMPOSANTE GENETIQUE ?
Isolats génétiques
Jumeaux
Agrégation familiale
&
Age précoce
J J

cartes du génome, balisées par des marqueurs géné-
tiques qui déterminent des locus. Lorsque le gène de
la maladie est lié statistiquement à un ou plusieurs de
ces marqueurs, il est situé (locus) sur une carte géné-
tique de liaison ou “ carte statistique ”, dont l’unité
de mesure est le centiMorgan. Cette localisation per-
met de retrouver dans les banques génomiques de
chromosomes artificiels une région suffisamment
petite qui peut être séquencée (carte génétique phy-
sique, dont l’unité de mesure est la paire de bases)
afin de déterminer le gène en cause. Une fois le gène
identifié, ses séquences nucléotidiques sont compa-
rées entre individus non malades et malades pour
identifier les changements de paires de bases qui
définissent la mutation morbide.
1. MARQUEURS GÉNÉTIQUES
Depuis une trentaine d’années, différentes collec-
tions de marqueurs génétiques ont été développées
pour cartographier le génome humain. Ces mar-
queurs sont des fragments d’ADN qui définissent
chacun un locus précis et unique au niveau du géno-
me. Ces fragments ont été choisis en fonction non
seulement de leur spécificité mais aussi en fonction
de leur polymorphisme (différents allèles), polymor-
phisme qui se traduit par de subtiles mais détectables
variations de séquences entre les différents allèles du
marqueur.
Ils doivent être tout à la fois spécifiques d’une région
chromosomique précise (locus) et être assez
variables (polymorphismes des allèles du locus)
entre individus. Cette double propriété permet
d’identifier la transmission des allèles d’une région
précise du génome (locus) au sein d’une même
famille ou au sein d’une population. Trois types de
marqueurs génétiques ont été particulièrement déve-
loppés : les RFLP (Restriction Fragment Length
Polymorphism), les microsatellites et les SNP
(Single Nucleotid Polymorphisms).
a) Les marqueurs de type RFLP
Les RFLP ont été utilisés pour établir les premières
cartes génétiques chez l’homme, cartes dites de pre-
mière génération. Ils ont contribué à la localisation
de gènes responsables de maladies génétiques telles
que la myopathie de Duchenne (1982), la chorée de
Huntington (1983) ou la mucoviscidose (1985).
Les enzymes de restriction, isolées de bactéries, ont
la particularité de couper l’ADN en des endroits pré-
cis et en fragments de taille variables (polymorphes)
(Fig. 37). Un RLFP est un fragment d’ADN géno-
mique. Utilisé comme sonde sur de l’ADN géno-
mique humain digéré par une enzyme de restriction,
il détecte des fragments dont la taille varie (poly-
morphisme) au sein d’une population d’individus.
Ces variations pour un même locus sont dues soit à
la répartition des sites de restriction (coupure), soit à
la présence de minisatellites (séquences répétitives
d’ADN) dans la région reconnue par la sonde.
b) Les marqueurs de type microsatellites
Les microsatellites sont des marqueurs largement
utilisés en cartographie génétique. Ils présentent un
nombre variable de répétitions dinucléotidiques
mais aussi tri ou tréta (ex. CA) qui leur donnent
leur polymorphisme. Ils sont uniformément répar-
tis sur l’ensemble du génome. Leur degré de poly-
morphisme est très élevé. Plus de 50000 marqueurs
de ce type ont été identifiés et balisent en quelque
sorte le génome humain (Fig. 38).
Ainsi, le microsatellite D8S133 est localisé sur le
chromosome 8 au niveau de la bande 22p. Cette loca-
lisation est identifiable pour tous les individus par une
séquence fixe, détectable en PCR, en amont et en aval
d’une région variable. La région variable présente un
polymorphisme génétique basé sur le plus ou moins
grand nombre de répétitions (CA) dans sa séquence. Il
est dit hautement polymorphe car il y a 70% des indi-
vidus pour lesquels cette région variable est diff é r e n-
te sur le chromosome 8 hérité du père et sur le chro-
mosome 8 hérité de la mère.
c) Les marqueurs de type SNPs (Polymorphismes
mono-nuclétotidiques)
Les autres polymorphismes fréquents au sein du
génome (le génome humain contiendrait de 5 à 29
millions de SNPs) sont des mutations simples spéci-
fiques d’un allèle. Ces polymorphismes mono-
nucléotidiques ou SNPs (prononcer “ s n i p s ” pour
Single Nucleotide Polymorphisms) peuvent être des
mutations fonctionnelles ou non fonctionnelles (Fig.
3 9 ) .
Un polymorphisme mono-nucléotidique fonction-
nel peut être localisé au niveau de la séquence
III. LES CARTES ET LES
MARQUEURS DU GENOME
744

745
Figure 37: Marqueurs RLFP
Figure 38: Représentation schématique d’un microsatellite avec un motif CA répété
n= nombre de répétition du motif CA sur l’allèle d’origine maternelle
m= nombre de répétition du motif CA sur l’allèle d’origine paternelle
n et m peuvent varier de 10 à 30 fois
amorces : courtes séquences de 15 à 25 bases connues et constantes qui encadrent le motif
répété et permettent l’amplification in vitro de ce marqueur par PCR (Polymerase Chain
Reaction).
5’
3’
5’
3’
3’
5’
3’
5’
ADN I
ADN II
1 - 2 - 3 - 4 - 5 : Enzyme de restriction
ADN III
Région spécifique
Région spécifique
Région variable
Région variable
(CACACA...)n
(GTGTGTGT...)
(CACACA...)
(GTGTGTGT...)

746
Figure 39 : Polymorphismes mono-nuclétotidiques (SNPs)
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
1
/
28
100%