1ère session

Licence STS Mention Biologie
U.E. Introduction à la
Génétique Moléculaire
Examen écrit
1ère session 2006-2007
Les deux questions de cours et l'exercice doivent être traités. L'utilisation de tout
document ou support de communication est interdite.
Durée de l'épreuve 2 heures
Cours (1 heure conseillée) :
Question 1 :. (30 minutes conseillées)
L’opéron tryptophane contient cinq gènes codant pour des enzymes impliquées dans la synthèse
du tryptophane (trpE, D, C, B et A), groupés sous la dépendance d'un seul système
promoteur/opérateur.
Cet opéron est exprimé par défaut en absence de tryptophane et il est réprimé par 2 mécanismes
complémentaires en présence de cet acide aminé (fixation d’un répresseur sur l’opérateur +
phénomène d’atténuation).
1. Expliquer par quel mécanisme un promoteur est reconnu par l’ARN polymérase chez les
bactéries. Préciser pourquoi tous les promoteurs ne sont pas reconnus avec la même
efficacité, et quelle propriété doit avoir le promoteur de l’opéron tryptophane pour que sa
transcription se fasse par défaut ?
2. Expliquer à l’aide de schémas précis et légendés, le phénomène d’atténuation observé
dans l’opéron tryptophane (autrement dit, comment le couplage traduction/transcription
régule l’avancée de l’ARN polymérase sur ce gène ?).
Question 2 : (30 minutes conseillées)
Le mécanisme d'excision/épissage permet d'éliminer les introns et de rabouter les exons présents
dans l'ARN pré-messager.
Après avoir donné les différentes séquences impliquées dans la reconnaissance et
l'élimination d'un intron, exposez à l'aide de schémas annotés, la mise en place séquentielle
des différents complexes au sein desquels se déroule la réaction d'excision /épissage.
Exercice (1 heure conseillée) :
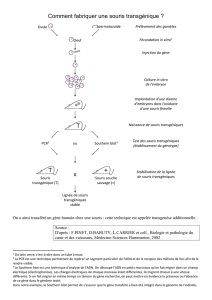
La protéine Myoser est codée par un gène qui contient 3 exons (Figure 1). Ce gène est
impliqué dans l'atrophie des muscles striés chez une souche de souris appelée Mdx. Pour
déterminer l'origine de ce phénotype, l'expression du gène Myoser est étudiée par Northern blot et
par Western blot avec différents tissus de souris contrôles et de souris Mdx (Figure 2).
1. Analyser et interpréter les résultats obtenus dans ces expériences de Northern Blot
et de Western Blot.

2. Est-ce que la taille de l'ARNm Myoser, chez les souris contrôles, est en accord avec
la structure du gène Myoser ?
3. Déterminer, en justifiant votre réponse, la taille du cadre de lecture ouvert (ORF) de
l'ARNm Myoser chez les souris contrôles.
Afin de mieux cerner le défaut d'expression du gène Myoser dans les souris Mdx. Le cadre de
lecture ouvert du gène Myoser est amplifié par RT/PCR à partir d'ARNm de muscle squelettique
de souris contrôles et de souris Mdx. Pour réaliser cette amplification deux amorces spécifiques
qui comportent à leur extrémité 5' un site EcoRI (GAATTC) ont été réalisées.
Amorce 1 : 5'-GAATTCGATGGCAGCACCCAGCCA-3'
Amorce 2 :5'-GAATTCTTATACTGTCCTCTCTGC-3'
4. En considérant qu'il y a 100 molécules d'ARNm Myoser dans l'extrait d'ARNm de
muscle squelettique placé dans le tube, exposer le protocole de RT/PCR mis en
place pour amplifier 1 µg d'ADN correspondant au cadre de lecture ouvert du gène
Myoser. (Il vous est demandé de préciser les températures et les durées des
différents cycles ainsi que le nombre de cycles).
5. Ecrire les 30 premières bases des extrémités 5' et 3' du brin codant du fragment
amplifié à partir des ARNm de souris contrôles.
Le fragment amplifié à partir des ARNm de souris contrôles a été digéré par l'enzyme de restriction
EcoRI et inséré dans le site EcoRI du vecteur d'expression procaryote pG-5 (Figure 3). De l'ADN
plasmidique, préparé à partir de cinq clones indépendants, a été digéré par les enzymes de
restriction EcoRI et NcoI et analysé par électrophorèse sur un gel d'agarose (Figure 4).
6. Quelles sont les caractéristiques du vecteur d'expression pG-5?
7. Après avoir analysé le gel, indiquer, en justifiant votre réponse, le ou les plasmides
qui permettront de produire une protéine recombinante Myoser.
Le gène myoser de souris contrôles et de souris Mdx a été cloné par PCR en utilisant les mêmes
amorces 1 et 2 que celles utilisées pour l'amplification de l'ADNc. Les fragments amplifiés ont été
séquencés par la technique de Sanger avec l'amorce 2. Une partie des autoradiographies des gels
de séquençage est donnée dans la figure 5.
8. Déduire la séquence nucléotidique du brin non transcrit de la région séquencée pour
le gène Myoser de souris contrôle et de souris Mdx. Orienter cette séquence
correctement en indiquant les extrémités 5' et 3'.
9. D'après ces séquences, quelle est l'origine moléculaire la plus vraisemblable du
phénotype des souris Mdx.
Masse moléculaire moyenne d'un acide aminé : 100 Da
Masse moléculaire moyenne d'un nucléotide : 330 Da
1 dalton (Da) = 1,66 10-24 g

A
E1 E2 E3
300 nt 200 nt 600 nt
500 nt 800 nt
NcoI (200 nt)
NcoI (500 nt) NcoI (1400 nt)
BamHi (900 nt) AatII (2200 nt)
ATG
(100 nt)
ggcagcgtataaattgacagctcagtcccg ATG GCA GCA CCC AGC CAA TTC AAT CAC ACA CGG GGA TCC AGT ACT CCC
M A A P S Q F N H T R G S S T P
AGA AGG AGG ACA ACC ATG GAG AGG AAA TTA //NNN……NNN//AGG TTC TTA CAG ATA AAC TGC AAG GAG ACT AGA
R R R T T M E R K L R F L Q I N C K E T R
TTC AAG AGA AAA TTT TCC TCA ACA GAC GCC CTT CTA TCC AGG CGG GGG CCA GGG CAC GGG CTC AAA TTA GCG
F K R K F S S T D A L L S R R A P A H A L K L A
TTA TTT ACA GCC GCC GCC AAA AAA AAA GAC CTA CGG CTC GGG CCC CAC AGA TCC TTT GCA GCG CAC CTA GCA
L F T A A A K K K D L R L G P H R S F A A H L A
AAC CGT GCT GAG TTT GCA GAG AGG ACA GTA TAA aagttggaaaagtccattgatgat
N R A E F A E R T V
Figure 1 : A. Structure schématique du gène Myoser. Les exons sont représentés par des boites et les introns
par des traits. La taille des exons et des introns est donnée en nucléotide. Une carte de restriction partielle est également
indiquée. B. Séquence nucléotidique partielle montrant le début et la fin du cadre de lecture ouvert de l'ADNc
Myoser de souris contrôles. La séquence prédite en acides aminés est également indiquée.
B
12 3 4 5 12 3 4 5
Souris contrôle Souris mutante
1,3
12 3 4 5 12 3 4 5
30
1,3
kDa kDa
kb kb
A.
B.
25
Figure 2 : Expression du gène Myoser. A. Analyse par Northern blot. Des quantités équivalentes d'ARN
extraits à partir de muscle squelettique (piste 1), cœur (piste 2), foie (piste 3), cerveau (piste 4) et estomac (piste 5)
ont été séparées par électrophorèse sur gel d'agarose en conditions dénaturantes. Les ARN transférés sur une membrane
de nitrocellulose ont été hybridés avec une sonde ADNc Myoser radiomarquée au 32P
B. Analyse par Western blot. Des quantités équivalentes de protéines extraites à partir de muscle squelettique
(piste 1), cœur (piste 2), foie (piste 3), cerveau (piste 4) et estomac (piste 5) ont été séparés par électrophorèse sur
un gel acrylamide /SDS page. Les protéines transférées sur une membrane de nitrocellulose ont été révélées avec un
anticorps dirigé contre la protéine Myoser.

AmpR
PSMC
ori
O
pG-5
3 kpb
PvuII
(1500)
NcoI
(800)
XbaI
(2600)
EcoRI
(50) HindIII
(56) NotI
(65)
RBS EcoRI HindIII NotI
5' GCGCAATAATACGCTCACTATAGGGCGCTCGGATCCAGGAGGCATCTAGAATTCCAAGCTTGAGGCGGCCGCCGTTTGTCTATAGTGAGTCGTATTCGAACGGCGCGG 3'
3' CGCGTTATTATGCGAGTGATATCCCGCGAGCCTAGGTCCTCCGTAGATCTTAAGGTTCGAACTCCGCCGGCGGCAAACAGATATCACTCAGCATAAGCTTGCCGCGCC 5'
Figure 3 : Carte schématique du vecteur pG-5. P et O : promoteur et opérateur de l’opéron lac Z
d’E. coli, SMC : site multiple de clonage, AmpR : gène de résistance à l’ampicilline, , ori : origine de
réplication. Une carte de restriction partielle du plasmide est donnée et la séquence du site multiple de
clonage est représentée de manière détaillée au dessus de la carte plasmidique.
pb
ND EcoRI NcoI ND EcoRI NcoI ND EcoRI NcoI ND EcoRI NcoI ND EcoRI NcoI
3000
900
850
3050
1550
2350
plasmide 1 plasmide 2 plasmide 3 plasmide 4 plasmide 5
Figure 4 : Analyse par électrophorèse sur un gel d’agarose de l’ADN plasmidique purifié
à partir de 5 clones indépendants. Des quantités équivalentes d’ADN plasmidiques non
digérés (ND), digérés par EcoRI ou par NcoI ont été déposées. L’ADN a été révélé par
coloration au bromure d’éthidium.
ACG T
-
+
Souris contrôle
ACG T
Souris Mdx
Figure 5 : Séquençage des fragments génomiques amplifiés par PCR. Une portion du gel de
séquence est présentée. La flèche indique le sens de migration des fragments d'ADN.
1
/
4
100%