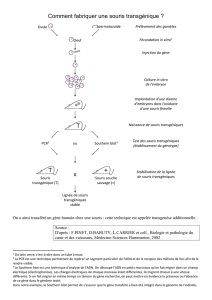
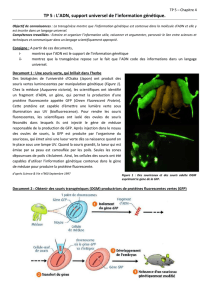
Génétique de la souris

Par Krys3000 (Groupe « The Trust » - http://www.cours-en-ligne.tk/) Page 1
GENETIQUE DES EUCARYOTES ET MARQUEURS
MOLECULAIRES
CHAPITRE IV : GÉNÉTIQUE DE LA SOURIS
I – 100 ANS DE GÉNÉTIQUE
La souris est apparue il y a 10000 ans, où, venue de l’inde, elle a envahit les zones agriculturales (croissant fertile) en tant que
passer clandestin des migrations humaines. Il existe beaucoup d’espèces de souris. La domestication a commencé dès
l’antiquité, et l’intérêt pour la génétique est né en 1905 par l’acquisition de souris par le Bussy Institute de W. Castle.
A la base, on pensait que les lois de Mendel ne pouvaient pas se généraliser en dehors du règne végétal et la théorie
concurrente du mélange des caractères voit le jour, prônant la dilution d’un caractère possible chez un animal sans jamais
élimination. Cette théorie est balayée au profit de celle de Mendel un peu plus tard par Cuénot qui en profite pour découvrir le
multiallélisme et l’épistasie. C’est Clarence Little qui prend conscience de la nécessité d’avoir des lignées génétiquement
homogènes pour étude et réalise le premier croisement en 1909. Pour contrer la tendance du génome à se recombiner, il faut
attendre une quinzaine de générations d’endogamie pour considérer une génération comme pure.
On peut utiliser les RFLP, que l’on va détecter par PCR, pour déterminer les mutations. Avant d’avoir connaissance du génome
de la souris, on faisait du clonage positionnel (positionnement d’un gène en rapport à des éléments connus). Ce genre de travail
implique d’avoir 400 individus avant d’obtenir ce que l’on cherche. A notre époque, les choses sont différences grâce au
séquençage.
II – GÉNOME DE LA SOURIS : PROXIMITÉ AVEC LE GÉNOME HUMAIN
Avant de commencer à séquencer on a construit des cartes génétiques avec des marqueurs environ toutes les mégabases sur
des YAC / BAC par méthode des clones chevauchants : on construit une banque ou chacun se chevauche pour reproduire le
génome – de telles séquences génomiques ordonnées sont nommées contig. Cette méthode est sure et précise, mais chère et
lente.
Une méthode plus récente est la méthode shotgun qui est rapide est moins couteuse, mais laisse de nombreux trous et erreurs :
il s’agit de détruire l’ADN en morceaux et de les intégrer à des vecteurs pour ensuite laisser un ordinateur compiler.
En 2002, une publication met en avant la synténie : on retrouve chez certains animaux des groupes de gènes retrouvés
identiques et dans le même ordre chez l’homme. 217 blocs de synténie ont été trouvés entre la souris et l’Homme.
De nos jours, on utilisera une troisième méthode, le séquençage de prochaine génération. On va donc pulvériser de la même
façon que le shotgun, mais au lieu d’intégrer aux vecteurs, on ajoute des adaptateurs à chacun des côtés de chaque morceau,
pour hybrider ensuite ceux-ci à un support et les séquencer par PCR avec fluorescence, c'est-à-dire avec des terminateurs
réversibles Génome reséquencé en une semaine.
III – GÉNÉTIQUE MOLÉCULAIRE : MODIFICATION GÉNÉTIQUES À VOLONTÉ
Découverte des cellules EC en 1960-70. Ces cellules proviennent des tératocarcinomes (tumeurs des organes génitaux) et
peuvent se diviser indéfiniment en culture. Etant capables de se différencier en n’importe quel type cellulaire, on s’est rendu
compte que l’on peut former des chimères en les réimplantant dans les blastocystes. On peut aussi utiliser les cellules
germinales primordiales (PCGs)
En utilisant cette approche, on a ensuite pensé à mettre en culture les cellules des blastocystes de souris, qui ont les mêmes
propriétés que les cellules EC, mais sont normales : ce sont les cellules ES. Ces cellules peuvent être maintenues dans un état de
multipotence, et parviennent encore plus à conserver cet état si elles sont déposées sur une couche de cellules nourricières. En

Par Krys3000 (Groupe « The Trust » - http://www.cours-en-ligne.tk/) Page 2
effet, celle-ci produisent une cytokine, la LIF, qui est donc le seul élément à pouvoir remplacer une culture sur cellules
nourricières. Une telle chose ne peut pas marcher chez l’Homme ou la voie de signalisation n’est pas la même, le LIF ne peux pas
assurer seul ce maintien.
Le récepteur du LIF forme un hétérodimère avec la protéine GP130, une protéine qui est commune aux récepteurs d’autres
cytokines, mais est aussi connue pour être la protéine reconnue par le VIH.
A l’aide des cellules ES, on peut donc faire de la différenciation cellulaire in vitro.
Les cellules ES peuvent donc permettre une génétique inverse pratiquement illimitée, puisqu’on peut les utiliser pour transférer
des mutations génétiques à l’animal implanté, et donc utiliser des stratégies de Knockout, ou, plus informatif encore, des
Knockouts avec substitution de gène, les Knock-in où un gène est introduit en lieu et place d’un autre et se retrouve exprimé à la
place. Cette méthode peut nous permettre de comparer des fonctions.
On peut aussi utiliser les mutations ponctuelles ciblées. On introduit ici une mutation après avoir au préalable introduit un
marqueur de sélection positive et un de sélection négative. Cette stratégie a été désormais remplacée par le système Cre-loxP
qui fonctionne selon Cre, un gène causant des recombinaisons sur le site loxP en codant pour une recombinase.
Une dernière technique est la stratégie du self-knockout. Ici, on veut avoir un contrôle spatial et temporel de notre invalidation.
Cela permet d’identifier certains phénotypes spécifiques. Le contrôle temporel est assuré par la creERT2 mutée pour qu’elle ne
soit active qu’en présence d’une substance, et le contrôle spatial par l’ajout de substance faisant la délétion d’allèles dans
certaines parties de la souris. Les deux sont parfois couplés lorsqu’un gène est censé s’activer d’abord en un lieu puis en un
autre.
1
/
2
100%