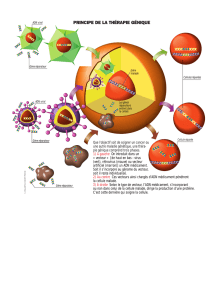
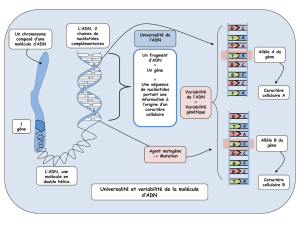
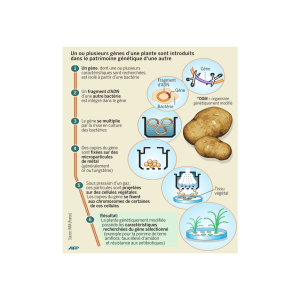
Comité scientifique - Haut Conseil des Biotechnologies

!
!
!
!
!
!
HAUT!CONSEIL!DES!BIOTECHNOLOGIES!
«!NOUVELLES!TECHNIQUES!»!4!
«!NEW!PLANT!BREEDING!TECHNIQUES!»!
Première!étape!de!la!réflexion!du!HCB!4!
Introduction!générale!!
!
"#$%&'!()!*+!,#-.%)$!*+/0!
!
!
!
!
12$!3$434&%5%4-!6)!&4-!72$)#2'!le! Haut! Conseil! des! biotechnologies! s’est! saisi! de! la! question! des! nouvelles!
techniques!d’obtention!de!plantes,!dites!«!New!Plant!Breeding!Techniques!»!(NPBT)8!9)&!:"7;'!<2%!4-5!6=,>!
?4-62%5! 342$! ?)$5#%-)&! #2! 6=.)(433)@)-5! )5! >! (#! @%&)! &2$! ()! @#$?A=! 6)! .#$%=5=&! .=B=5#()&! )-! C@=$%<2)! 62!
:4$6'!&2&?%5)-5!)-! )DD)5!2-)! &=$%)!6)! <2)&5%4-&!#2,42$6EA2%!6=F#552)&'!(%=)&!#2G! 4334$52-%5=&!)5!#2G! $%&<2)&!
<2E)(()&!342$$#%)-5!3$=&)-5)$'!#%-&%!<2E>!(#!@#-%H$)!64-5!)(()&!6).$#%)-5!I5$)!$=B()@)-5=)&8!
!
J)! K97! 3$4?H6)! %?%! >! 2-)! 3$)@%H$)! =5#3)! 6)!(#! $=D()G%4-! &2$! ()&! :"7;/'! )-! 3$4?=6#-5! >! (#! ?(#$%D%?#5%4-! 6)&!
5)$@)&!62!6=F#5!)5!>!(E)G3)$5%&)!6)!?)$5#%-)&!6)&!<2)&5%4-&!)-!,)28!L(!&E)&5!D4?#(%&='!6#-&!?)55)!première!étape!
de!son!travail'!&2$!()&!34%-5&!&2%.#-5&!M!
- (#!6)&?$%35%4-!6)&!3$%-?%3#()&!:"7;!&42@%&)&!>!=526)!>!?)!,42$!3#$!(#!94@@%&&%4-!)2$43=)--)!N!
- ()&! )-,)2G! <2%! O! &4-5! (%=&!M! 4334$52-%5=&! 345)-5%)(()&'! -45#@@)-5! )-! 5)$@)&! 6)! 6=.)(433)@)-5! 6)!
.#$%=5=&! 3$=&)-5#-5! 6)&! ?#$#?5=$%&5%<2)&! -42.)(()&! <2%! -)! 342$$#%)-5! 3#&! I5$)! 4F5)-2)&! 3#$! ()&!
5)?A-%<2)&! 6)! &=()?5%4-! 6=,>! 6%&34-%F()&! P&=()?5%4-! ?(#&&%<2)'! 5$#-&B=-H&)Q!N! $%&<2)&! <2E)(()&!
342$$#%)-5! =.)-52)(()@)-5! 3$=&)-5)$'! #2! 3(#-! &#-%5#%$)! 42! )-.%$4--)@)-5#('! @#%&! #2&&%! #2G! 3(#-&!
&4?%4R=?4-4@%<2)!)5!=5A%<2)!N!
/!S4%$!()&!$)().=&!6)&!6=?%&%4-&!6)&!72$)#2G!6)&!T!-4.)@F$)'!/)$!6=?)@F$)!)5!/T!6=?)@F$)!*+/T8!U-!-45)$#!<2)!?)!5$#.#%(!
&E)&5!6=$42(=!6#-&!2-)!$)(#5%.)!2$B)-?)!=5#-5!64--=!()&!#--4-?)&!D#%5)&!3#$!(#!94@@%&&%4-!)2$43=)--)!<2#-5!>!(E#B)-6#!6)!
&#!$=D()G%4-!&2$!(E)-?#6$)@)-5!6)&!:"7;'!)5!<2)!()&!@)@F$)&!62!K97!4-5!=5=!#@)-=&!6#-&!6)&!6=(#%&!5$H&!F$)D&!>!3$)-6$)!
?4--#%&&#-?)!6)!5)G5)&!-4@F$)2G!)5!6)-&)&!&2$!2-!&2,)5!?4@3()G)8

- (#! <2)&5%4-! 6)! &#.4%$! &%! )5! ?4@@)-5!%(! ?4-.%)-6$#%5'! #2! $)B#$6! 6)! ?)&! =(=@)-5&'! 6E)-?#6$)$! ()!
6=.)(433)@)-5!6)&!:"7;!)5!(#!?4@@)$?%#(%%4-!6)!()2$&!3$462%5&8!!
!
Pour! aborder! ces! questions,! le! HCB! a! mobilisé! les! compétences! de! son! Comité! scientifique! (CS)! et! de! son!
Comité!économique,!éthique!et!social!(CEES),!selon!les!modalités!suivantes!définies!par!le!Bureau.!!!
!
Pour! le! CS!M! ()! 72$)#2! #! 6)@#-6=! #2! 91! 6E#F4$6)$! 6)2G! <2)&5%4-&!M! (#! 6)&?$%35%4-! 6)&! :"7;'! )-! &)!
D4?#(%&#-5!6#-&!2-!3$)@%)$!5)@3&!&2$!()&!V!5)?A-%<2)&!#%-&%!<2#(%D%=)&!6#-&!()&!6%&?2&&%4-&!)2$43=)--)&*!N!()&!
=.)-52)(&!$%&<2)&!6)&!:"7;!342$!(E)-.%$4--)@)-5!)5!(#!&#-5='!#%-&%!<2)!()&!=(=@)-5&!&2&?)35%F()&!6)!&)$.%$!(#!
$=D()G%4-!<2#-5!>!(#!<2#(%D%?#5%4-!W!UXY!Z!42!-4-!6)&!3$462%5&!6)&!:"7;8!!
[-!X$423)!6)!5$#.#%(\!#!6=?$%5!?)&!5)?A-%<2)&!)5!%6)-5%D%=!()&!<2)&5%4-&!<2E)(()&!&42(H.)-58![-)!-45)!$=6%B=)!>!
3#$5%$!6)!?)&!64?2@)-5&!#!)-&2%5)!=5=!6%&?25=)!)5!.#(%6=)!)-!&=#-?)!3(=-%H$)!62!?4@%5=]8!!
!
Pour!le!CEES!M!()&!$)3$=&)-5#-5&!6)&!4$B#-%%4-&!)5!3)$&4--#(%5=&!<2#(%D%=)&!@)@F$)&!62!9^^1!4-5!=5=!
%-.%5=)&!>!3$462%$)!6)&!?4-5$%F25%4-&!&O-5A=5%<2)&!6)&5%-=)&!>!?(#$%D%)$!()2$!34&%5%4--)@)-5!N!?)&!?4-5$%F25%4-&!
#%-&%! <2E2-! $=&2@=! 6)! -4@F$)2&)&! #-#(O&)&! ,2$%6%<2)&! 6)&! :"7;! 4-5! =5=! .)$&=)&! #2G! 6=F#5&! 62! 9^^1! )5!
6%&?25=)&! )-! &=#-?)! 3(=-%H$)! 62! ?4@%5=T8! 12%5)! >! ?)! 6=F#5'! 6E#25$)&! @)@F$)&! 62! 9^^1! 4-5! D#%5! ?4--#_5$)! 42!
)G3(%?%5=! ()2$! 34&%5%4--)@)-58! [-)! -45)! &O-5A=5%&#-5! (E)-&)@F()! 6)! ?)&! ?4-5$%F25%4-&! )5! ()&! 6=F#5&! #2G<2)(&!
)(()&!4-5!64--=!(%)2!#2!9^^1!#!=5=!$).2)!)5!.#(%6=)!3#$!()&!@)@F$)&!62!?4@%5=8!!
!
!
A!l’issue!de!cette!première!étape,!on!trouvera!donc!ci4dessous!les!éléments!suivants.!!
!
1.!Pour!ce!qui!concerne!les!travaux!produits!par!le!Comité!scientifique!M!!
!
- 2-)! -45)! %-5$462%&#-5! (#! <2)&5%4-! 6)&! :"7;! #%-&%! <2)! ()&! %-5)$$4B#5%4-&! <2E)(()&! &42(H.)-5! #2! 3(#-!
&?%)-5%D%<2)!PW!91!`!:45)!&2$!()&!W!:42.)(()&!;)?A-%<2)&!Z!Pa4?2@)-5!/Q!N!!
- )-!#--)G)!6)!?)55)!-45)'!2-)!&=$%)!6)!D%?A)&!6)&?$%35%.)&!6)!?A#<2)!5)?A-%<2)8!!
!
!
!
2.!Pour!ce!qui!concerne!les!travaux!produits!par!le!Comité!économique,!éthique!et!social!M!!
!
R!J)&!?4-5$%F25%4-&!6)!@)@F$)&!62!9^^1!M!
!
*!S4%$!(#!(%&5)!6)&!5)?A-%<2)&!6#-&!()&!64?2@)-5&!62!91!<2%!&2%.)-58!a#-&!()!6)2G%H@)!.4()5!6)!&#!$=D()G%4-'!()!91!&)$#!%-.%5=!
>!?4-&%6=$)$!6E#25$)&!5)?A-%<2)&!=.)-52)(()@)-5!3)$5%-)-5)&'!)G3(4%5#-5!-45#@@)-5!()&!@=?#-%&@)&!=3%B=-=5%<2)&'!)5!<2%!
342$$#%)-5!>!(E#.)-%$!I5$)!25%(%&=)&!342$!(E4F5)-5%4-!6)!3(#-5)&8!
\!X$423)!@%&!)-!3(#?)!)-!D%-!6)!/)$!@#-6#5!62!K978!94@34&%5%4-!6=5#%((=)!6#-&!(#!-45)!62!918
]!1=#-?)!62!91!62!/0!6=?)@F$)!*+/T8
T!1=#-?)&!62!9^^1!6)&!/+!-4.)@F$)!)5!/0!6=?)@F$)!*+/T8!!

894-5$%F25%4-!?4(()?5%.)!3$434&=)!3#$!()&!4$B#-%%4-&!9443!6)!b$#-?)'!b:1^C'!X$423)@)-5!:#5%4-#(!
L-5)$3$4D)&&%4--)(!6)&!1)@)-?)&'!c)2-)&!CB$%?2(5)2$&'![-%4-!b$#-d#%&)!6)&!1)@)-?%)$&!P?%R#3$H&!96b'!
b:1^C'!X:L1'!cC'![b1Q!PC--)G)!/Q8!
8!94-5$%F25%4-!?4(()?5%.)!3$434&=)!3#$!()&!4$B#-%%4-&!J)&!C@%&!6)!(#!5)$$)'!94-D=6=$#5%4-!"#O&#--)'!
b=6=$#5%4-!:#5%4-#()!6eCB$%?2(52$)!7%4(4B%<2)'!b$#-?)!R!:#52$)!R!^-.%$4--)@)-5'!X$))-3)#?)'!f=&)#2!
1)@)-?)&!"#O&#--)&!)5![-%4-!:#5%4-#()!6)&!C3%?2(5)2$&!6)!b$#-?)!P?%R#3$H&!C6;'!9"'!b:C7'!b:^'!X"'!
f1"! )5! [:CbQ! PC--)G)! *Q8! 9)55)! ?4-5$%F25%4-'! )-&2%5)! )-$%?A%)! 6E=(=@)-5&! #334$5=&! 3#$! a8! ^.#%-!
Pb:C7Q!)5!78!74-g%!PJ)&!C@%&!6)!(#!;)$$)Q'!&)!6%.%&)!)-!T!3#$5%)&!PC--)G)&!*8/!>!*8TQ8!!
894-5$%F25%4-!6)!(#!b=6=$#5%4-!62!94@@)$?)!)5!6)!(#!a%&5$%F25%4-!Pb9aQ!PC--)G)!\Q8!
894-5$%F25%4-!62!94-&)%(!:#5%4-#(!6)&!C&&4?%#5%4-&!b#@%(%#()&!(#h<2)&!P9:CbCJQ!PC--)G)!]Q8!
894-5$%F25%4-!6)!1#$#A8!S#-2G)@'!3)$&4--#(%5=!<2#(%D%=)!)-!6$4%5!PC--)G)!TQ8!
894-5$%F25%4-!6E^&5)(()8!7$4&&)5'!3)$&4--#(%5=!<2#(%D%=)!)-!6$4%5!PC--)G)!0Q8!
!
aE#25$)&! 4$B#-%%4-&! )5! 3)$&4--#(%5=&! <2#(%D%=)&! 4-5! &42@%&! #2! 1)?$=5#$%#5! 62! K97! 6)&! =(=@)-5&!
6E%-D4$@#5%4-!)5!6)!34&%5%4--)@)-5!-)!3$)-#-5!3#&!(#!D4$@)!6E2-!5)G5)!$=6%B=!in!extenso!P)5!%-5=B$=&!
6#-&! (#! :45)! &O-5A=5%<2)! ?%5=)! infraQ!M! 1)$B)! 74#$%-%! P3)$&4--#(%5=! <2#(%D%=)! )-! &4?%4(4B%)Q!N! 143A%)!
b4-<2)$-%)!PC&&4?%#5%4-!6)&!f=B%4-&!6)!b$#-?)Q!N!b$#-d4%&!J2?#&!P944$6%-#5%4-!f2$#()Q!N!f)-=!Y#g#$&!
P94(()?5%D!L-5)$#&&4?%#5%D!12$!(#!1#-5=Q8!
!
!>! "42$! ?4@3(=5)$! ?)5! )-&)@F()'! )&5! 3$434&=! 2-! W!f=&2@=! 6)&! 3$%-?%3#()&! #-#(O&)&! ,2$%6%<2)&! 6%&34-%F()&!
$)(#5%.)&!>!(E#33(%?#F%(%5=!6)!(#!6%$)?5%.)!*++/i/Vi9^!#2G!:"7;!Z!PC--)G)!jQ8!!
!
R!U-!5$42.)$#!)-D%-!2-!64?2@)-5!6)!&O-5AH&)!=5#F(%!>!3#$5%$!6)!542&!?)&!=(=@)-5&!)5!6)&!6=F#5&!#2G<2)(&!%(&!4-5!
64--=!(%)2!#2!9^^1!P9^^1!R!W!1O-5AH&)!6)&!?4-5$%F25%4-&!)5!6)&!6=F#5&!ZQ!Pa4?2@)-5!*Q8!!
!
Comme!décidé!par!le!Bureau!du!HCB!et!étant!donné!le!temps!contraint!dans!lequel!cette!première!étape!du!
travail! s’est! déroulée,! le! HCB! engage! dès! à! présent! une! deuxième! étape!6)&5%-=)! >! #33$4D4-6%$! 2-)! &=$%)!
34%-5&! 64-5! ()&! &2%.#-5&!M! 5$#d#F%(%5=! 6)&! :"7;'! %-D4$@#5%4-! 6)&! ?4-&4@@#5)2$&'! 3$45)?5%4-! ,2$%6%<2)! 6)&!
5)?A-%<2)&!)5!6)&!3$462%5&'!$HB()@)-5#5%4-'!)G#@)-!3$4&3)?5%D!6)&!5)?A-%<2)&'!#-#(O&)!6)!(#!@#52$%5=!6)!?)(()&!
<2%!&4-5!)DD)?5%.)@)-5!@%&)&!)-!k2.$)&'!)5?8!#D%-!6)!D42$-%$!#2G!#254$%5=&!32F(%<2)&!(E)-&)@F()!6)&!=(=@)-5&!
6E#33$=?%#5%4-!-=?)&&#%$)&08!!
JE)-&)@F()!6)!?)&!34%-5&!D)$#!(e4F,)5!6)!6=.)(433)@)-5&!6#-&!(#!6)2G%H@)!3#$5%)!6)&!5$#.#2G!62!K978!
!!!!!!
!
9A$%&5%-)!:4%.%(()'!"$=&%6)-5)!62!K#25!94-&)%(!6)&!F%45)?A-4(4B%)&!
0
!

HIGH COUNCIL FOR BIOTECHNOLOGY
NEW PLANT BREEDING TECHNIQUES
General introduction: First stage of HCB
deliberations
Paris, 20 January 2016
At the suggestion of its Board, the High Council for Biotechnology decided to address the issue of new plant
breeding techniques (NPBTs) through a self-referral. These NPBTs, some of which have already led to
development and market release of new plant varieties in North America, raise a number of questions,
currently being debated, concerning their risks and opportunities and the manner in which they should be
regulated.
HCB has begun the first stage of its deliberations concerning NPBTs
1
by clarifying the terms of the debate and
assessing some of the issues involved. It has focused, in this first stage of its work, on the following points:
-
Description of the main NPBTs currently being examined by the European Commission;
-
Related issues: potential opportunities, particularly for development of varieties with new traits that
cannot be obtained using the breeding techniques already available (conventional breeding,
transgenesis); possible risks to health and the environment as well as in the socio-economic and ethical
fields;
1
See statements of decisions of the Board meetings of 5 November, 1 December and 15 December 2015. It should be noted that this work
had to be done in relatively short order, given the announcements by the European Commission on the discussion timetable for NPBT
regulation, and members of HCB had to study a large volume of technical literature on a complex subject in a very short space of time.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
 59
59
 60
60
 61
61
 62
62
 63
63
 64
64
 65
65
 66
66
 67
67
 68
68
 69
69
 70
70
 71
71
 72
72
 73
73
 74
74
 75
75
 76
76
 77
77
 78
78
 79
79
 80
80
 81
81
 82
82
 83
83
 84
84
 85
85
 86
86
 87
87
 88
88
 89
89
 90
90
 91
91
 92
92
 93
93
 94
94
 95
95
 96
96
 97
97
 98
98
 99
99
 100
100
 101
101
 102
102
 103
103
 104
104
 105
105
 106
106
 107
107
1
/
107
100%