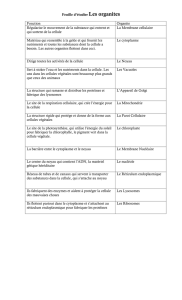
Parutions Nouméavirus, un nouveau virus d`amibe, contrôle le

Accueil>Larechercheensciencesduvivant>Parutions>Nouméavirus,unnouveauvirusd’amibe,contrôlelenoyaude
sonhôteàdistance
Parutions
Nouméavirus,unnouveauvirusd’amibe,contrôlelenoyaudesonhôteàdistance
DeschercheursdulaboratoireInformationgénomiqueetstructurale,encollaborationavecle
laboratoiredeBiologieàgrandeéchelle,ontcaractériséunnouveauvirusgéantd’amibe,
Noumeavirus,dontlemodederéplicationoriginalremetencauseladichotomietraditionnelleentre
lesvirus«nucléaires»etlesvirus«cytoplasmiques».Cetteétudeaétépubliéele21avril2017dans
larevueNatureCommunications.
Lesmicroorganismeseucaryotessedistinguentdesprocaryotes(lesbactériesetlesarchaebactéries)parla
compartimentationstrictedeleurscellulesquiséparelenoyau,oùsedéroulentlaréplicationdel’ADNetsa
transcriptionenARNmessagers,ducytoplasmeoùlesribosomesdécodentlesARNmessagerspour
synthétiserlesprotéinescorrespondantes.
Pourinitierleurmultiplication,lesvirusàgénomeADNquiinfectentunecelluleeucaryote(commeune
amibe)doiventsurmontercecloisonnement.Jusqu’àprésent,onleurconnaissaitdeuxstratégies
différentes:soitdirectementtransporterleurgénomedanslenoyauetyutiliserlamachineriecellulaire(ce
sontlesvirusdits«nucléaires»),soitmettreenœuvreleurpropremachineriedetranscriptionetde
réplicationauseinducytoplasme(virusdits"cytoplasmiques").Danscederniercas,lecomplexe
transcriptionnelcodéparlegénomeviralnepeutinitierlecycleinfectieuxsansêtreaussiembarquédansla
particulesouslaformedeprotéinesprêtesàl’emploi.C’estcetteprédiction,jusqu’alorsvalidéepourtoutes
lesfamillesdeviruscytoplasmiquestestés(Mimivirus,Vacciniavirus,Pithovirus),quevientdecontredire
cetteétude.
AlorsqueNoumeavirusserépliquedanslecytoplasmeetcodebienpoursespropresARNpolymérases,les
chercheurs,enanalysantlecontenuprotéiquedesparticules,onteulasurprisedenepasentrouverla
moindretracedanslaparticulevirale.Noumeavirusnepeuxdoncpasinitiersoncycleinfectieuxsansl’aide
delamachineriedesonhôte,pourtantconfinéedanslenoyau.Cetteanomalielesapoussésàreprendre
l’étudedétailléeducycleinfectieuxdansuneamibedontlenoyauaétérendufluorescent.Ilsontalors
observéquel’infectionparNoumeavirusdéclenchaituneperméabilisationtemporairedunoyaudèsles
premièresminutes,rendantpossiblelerecrutementdesenzymesnucléairesnécessairesàlatranscription
desgènesprécocesduvirus.Phénomèneétonnantobservépourlapremièrefois,lenoyaucellulairereprend
sonapparencenormaleaprèsquelquesheures,alorsquelamultiplicationdesparticulesviralesbatsonplein
danslecytoplasme.
Noumeavirus(etprobablementlafamilledesMarseilleviridaedanssonensemble)inauguredoncun
nouveaumodederéplication,intermédiaireentreceluidesvirusnucléairesetceluidesvirusserépliquant
entièrementdanslecytoplasme.Plutôtquedetransportersongénomedanslenoyau(unprocessus
complexe),Noumeavirusaévoluélacapacitéd’attirerdanslecytoplasmelesenzymesnucléaires
temporairementnécessairesàl’expressiondesesgèneslesplusprécoces.
Cettedécouverted’unmécanismedecontrôleàdistancedunoyauaplusieursconséquences.Ellemontre
d’abordquel’analyseducontenuprotéiquedesparticulesvirales,encomplémentdeleurséquence
génomiqueetdel’observationmicroscopique,estessentielleàlacompréhensiondeleursmodesde
réplication.Ensuite,elleouvredenouvellespistesquantaumoded’infectionmisenœuvrepard’autres
grandsvirusàADNdénuésd’appareiltranscriptionnel(commelesChlorovirus)maisdontlepassageparle
noyaun’ajamaisétémisenévidence.Enfin,elleconfortelemodèled’évolutionréductivedesgrandsvirusà
ADNprivilégiéparlesauteurs,ensuggérantunmécanismeparlequeldesvirusinitialementcytoplasmiques
ontpus’engagerdanslaperteprogressivedeleurautonomievis-à-visdunoyau,enattendantd’évoluerla
capacitéd’ytransporterleurgénome.
Figure:Noyau(fluorescenceverte,GFP)et«usineàvirion»(bleu,DAPI)dansuneamibeàunstadetardif
d’infectionparNoumeavirus.Aprèsavoirdiffusédanstoutelacellule,lafluorescenceverteseretrouveconfinéedans
lenoyauquiaretrouvésonintégritéstructurale.
©IGS.CNRS-AMU.
Ensavoirplus
Noumeavirusreplicationreliesonatransientremotecontrolofitshostnucleus.
ElisabethFabre,SandraJeudy,SébastienSantini,MatthieuLegendre,MathieuTrauchessec,Yohann
Couté,Jean-MichelClaverie,ChantalAbergel.
NatureCommunications.8,Articlenumber:15087(2017).doi:10.1038/ncomms15087
Contactchercheur
ChantalAbergel
InformationGénomiqueetStructurale
CNRSUMR7256--Aix–MarseilleUniversité,
InstitutdeMicrobiologiedelaMéditerranée
IGS-CASE934
163avenuedeLuminy
13288MarseilleCedex09
0491825422
Miseenlignele24avril2017
Liensutilesetpartenaires
Rechercher
surcesite:
1
/
1
100%