Evolution des génomes de vertébrés

Evolution des génomes de vertébrés
Hugues Roest Crollius
Groupe Dyogen - CNRS
Ecole Normale Supérieure
Ecole d’Eté de Lumigny
07 Septembre 2007
•Les mutations
•La recombinaison
•La dérive génétique
•La sélection naturelle
L’évolution est gouvernée par quatre forces:
« Nothing makes sense in biology except in the light of evolution »
(Theodosius Dobzhansky)
Les processus biologiques que nous observons sont le produit de
l’évolution.
non adaptatif
adaptatif
Substitutions
Insertions
Délétions
Réarrangements chromosomiques

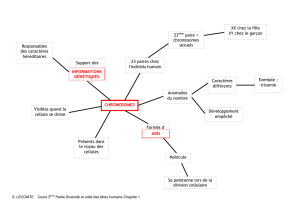
1. La «!paléogénomique?!»
2. Classification des vertébrés
3. Mécanismes gouvernant l’évolution des chromosomes
4. Les techniques d’observation
a. Cytogénétique
b. Génomique comparative
5. Les méthodes de reconstruction
a. La parcimonie en cytogénétique
b. E-painting
c. Des algorithmes
i. The reversal sorting problem
ii. MGR
d. Duplications complètes de génomes
6. Quelques résultats:
a. l’ancêtre des mammifères placentaires
b. l’ancêtre des poissons
7. Implications non-intuitives lors d’une reconstruction
8. Conclusions
PLAN
La Paléogénomique
-Séquençage et l’analyse d’ADN ancien (Mammouth, Homme de
Neandertal, etc…).
- extraction d’ADN à partir de tissus preservés
- forte dégradation par oxydation
- limite temporelle (< ~1 million d’années)
-Etude des génomes ancestraux sur la base de reconstructions à partir
des génomes contemporains
- passe nécessairement par un processus d’abstraction
- les résultats resteront des conjectures basées sur les données disponibles
- selon le niveau de résolution visé, limite temporelle > 500 millions d’années

sarcopterygii
actinopterygii
Poisson zèbre
Medaka
Epinoche
Fugu
Tetraodon
Percomorpha
Otocephala
Tetraodontidae
Osteichthyes
0100 50200 150300 250400 350450
Millions d’années
Actinopterigii Teleostei Euteleostei
RequinChondrichthyes
Crapaud
Poule
Ornithorynque
Opossum
Elephant
Tenrec
Tatou
Hérisson
Vache
Chien
Macaque
Humain
Chimpanze
Rodentia
Carnivora
Boreoeutheria
Xenarthra
Metatheria
Prototheria
Theria
Mammalia
Amniota
Lapin
Rat
Souris
Murinae
Lagomorpha
Catarrhini
Insectivora
Eutheria
Sarcopterigii
Tetrapoda
La Paléogénomique
?
?
?
?
Opossum
Eléphant
Tenrec
Tatou
Vache
Chien
Macaque
Humain
Chimpanzé
Rodentia
Carnivora
Boreoeutheria
Xenarthra
Metatheria
Prototheria
Theria
Mammalia
Lapin
Rat
Souris
Murinae
Lagomorpha
Catarrhini
Insectivora
Eutheria
Hérisson
Ornithorynque
Problème: nous n’étudions que des processus contemporains.
Les processus biologiques que nous observons sont le produit de l’évolution.

sarcopterygii
actinopterygii
Poisson zèbre
Medaka
Epinoche
Fugu
Tetraodon
Percomorpha
Otocephala
Tetraodontidae
Osteichthyes
0100 50200 150300 250400 350450
Millions d’années
Actinopterigii Teleostei Euteleostei
RequinChondrichthyes
Crapaud
Poule
Ornithorynque
Opossum
Elephant
Tenrec
Tatou
Hérisson
Vache
Chien
Macaque
Humain
Chimpanze
Rodentia
Carnivora
Boreoeutheria
Xenarthra
Metatheria
Prototheria
Theria
Mammalia
Amniota
Lapin
Rat
Souris
Murinae
Lagomorpha
Catarrhini
Insectivora
Eutheria
Sarcopterigii
Tetrapoda
La classification des vertébrés
Les génomes évoluent selon un nombre réduit de mécanismes:
- Inversions
- Translocations
- Transpositions
- Duplications
- Fusions et Fissions
- substitutions nucléotidiques
- insertions et délétions
L’évolution des chromosomes: mécanismes

Les techniques d’observation
La cytogénétique comparée
- comparer des préparations de chromosomes de 2 espèces
ou
- hybrider des chromosomes d’espèces différentes
La génomique
1) cartographier et/ou séquencer des génomes
2) comparer l’ordre des marqueurs sur les cartes/séquences
Chromosomes de tatou Chromosomes de fourmilier
Xenarthra
Metatheria
Prototheria
Theria
Mammalia
Eutheria
Opossum
Elephant
Tenrec
Armadillo
Cow
Dog
Macaque
Human
Chimpanzee
Rabbit
Rat
Mouse
Hedgehog
Platypus
Svartman et al. 2006
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
1
/
23
100%