Diapositive 1

Applications des méthodes
phylogénétiques en
linguistique.
Matthieu Willems
22 mars 2016

PLAN
1. Rappels sur l'inférence phylogénétique.
2. Évolution réticulée.
3. Réseaux phylogénétiques.
4. Biolinguistique
5. Un nouvel algorithme.
6. Application à la biolinguistique.

1. Inférence phylogénétique
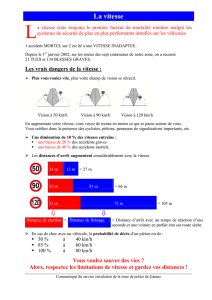
Objectif : reconstruire l'histoire évolutive d'un ensemble
d'espèce à partir de données moléculaires

Entrées des algorithmes
phylogénétiques
•Méthodes basées sur les caractères
(séquences moléculaires) :
E1 : ATCGGATCGTATTCGA
E2 : ATGCCTAGGATATGGT
E3 : TTGGGAACGCATCCTA
•Méthodes basées sur les distances :
E1 E2 E3
E1 011 6
E2 11 012
E3 612 0

Principales méthodes
1. Méthodes de distances (NJ, UPGMA).
2. Maximum de parcimonie.
3. Maximum de vraisemblance.
4. Méthodes bayésiennes.
5. Peu de nouveaux développements
depuis 15 ans.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
1
/
41
100%