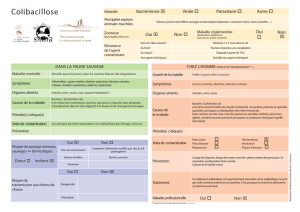
Expression chez E. coli

Bi 231: Ingénierie
des Protéines
cours 2

Plan Général
•I. Introduction et rappels.
•II. Purification des protéines.
•III. Surexpression d'une protéine.
•IV. Mutagenèse: techniques et
applications.
•V. L'insuline : exemple d'une stratégie.
•VI. Analyse d'articles.

31 –Cahier des charges.
32 –Expression chez E. coli
- Avantages.
- Organisation générale d'un vecteur
- Exemples de systèmes d'expression.
- Les différentes souches de E. coli existantes
- Les paramètres ajustables lors d'une surexpression
33 –Autres hôtes bactériens : avantages et inconvénients.
34 –Expression chez la levure : Hôtes et vecteurs
disponibles.
35 –Expression dans des cellules Eucaryotes supérieures
animales et végétales.
III. Surexpression d'une protéine.

31 Cahier des charges (1)
définir le(s) but(s) de cette surexpression :
•tests enzymatiques => purifier la protéine tout en
conservant sa fonction
• production d’anticorps spécifiques => très
grande pureté mais fonctionnalité et structure
non nécessaire
•injection chez homme, animal => Absence
d’impureté immunogène et/ou toxique, stérilité,
absence de particules en suspension, pH, ...
(normes européennes)

31 Cahier des charges (2)
définir l’origine et caractéristiques de la protéine
surexprimée :
•Procaryotes, Archée, Eucaryotes primitifs,
supérieurs (animal, végétal), ... => modif° post
traduct°les, code génétique (fréquence des
codons).
•Localisation cellulaire.
•Cofacteurs.
•Coexpression
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
1
/
16
100%