Outils bioinformatiques pour l`analyse du microenvironnement tumoral

•Dr. J. Galon
•Matthieu CAMUS
INSERM U255
Dirigée par : Pr. H. Fridman
Centre des Cordeliers
Paris VI
Outils Bioinformatiques
pour l‘Analyse du
Microenvironnement Tumoral

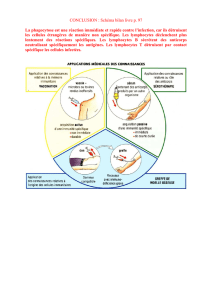
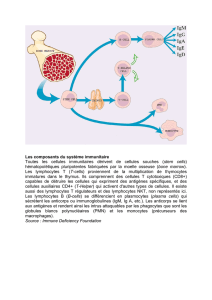
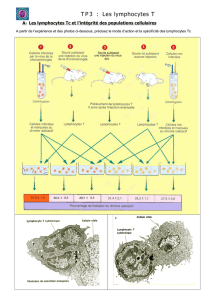
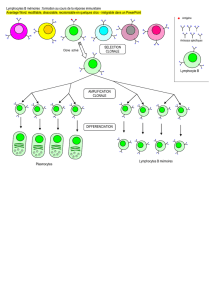
Lymphocytes T Régulateurs,
Lymphocytes B, NK, Macrophages …
Cellules présentatrices d’Antigènes
Lymphocytes T
Effecteurs Cytokines
Facteurs de croissance
Chemokines
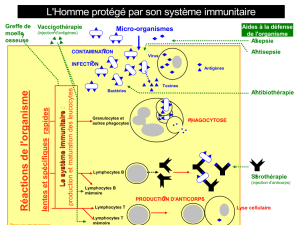
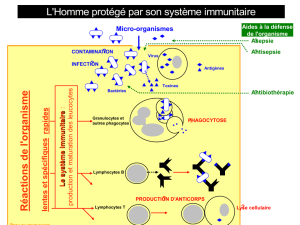
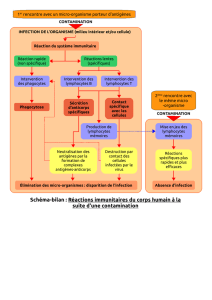
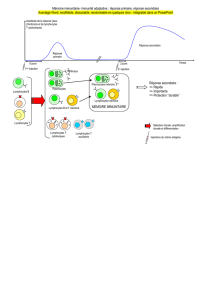
Immunologie des Tumeurs
Cellule Dendritique
Micro-Environnement Tumoral
-> analyses à Large-échelle pour l’étude des cellules infiltrantes
Stroma
Background
Tumeur

FACS Phenotype Data (n=820)
FACS Functional Data (n=980)
DNA MicroArrays (n=12000)
CIPHERGEN Collaboration
SELDI-TOF (proteomic)
Colon cancer : Approaches
parameters patients
10
950
12000
10
ALPHELIS Collaboration
Tissue MicroArrays (TMA)
(1 marqueur cellulaire, deux régions)
APPLIED Collaboration
Taqman PCR LDA (n=48)


•Base de données (autorisation/authentification/CNIL)
•Gestion des données cliniques
•Gestion des données biologiques et expérimentales
•Modules de connections aux appareils de laboratoire
•Requêtes multicritères (listing, analyse)
•Connections aux autres bases (MARS, publiques)
•Connections aux logiciels d’analyse (Genesis, PathwayExporer)
•Modules d’analyses et statistiques
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
1
/
33
100%