Biosynthèse des macromolécules

Biosynthèse des
macromolécules

RAPPELS

Rappels
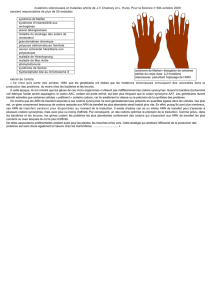
• L’ADN est la molécule la plus grosse de l’organisme
•Double hélice faite de deux monomères enroulés les uns
sur les autres unis entre-eux par des liaisons
hydrogènes
•dont les deux brins sont :
–antiparallèles : l'un est constitué d'un enchaînement
commençant à gauche et se poursuivant vers la droite, l'autre
commençant à droite et se poursuivant vers la gauche ;
–complémentaires : chaque adénine (A) d'un des deux brins est
liée par deux liaisons hydrogène avec une thymine (T) de l'autre
brin, et chaque guanine (G) d'un brin est liée par trois liaisons
hydrogène avec une cytosine (C) de l'autre brin.

Rappels
• Les monomères sont constitués d’unités = nucléotide
(sucre (désoxyribose) + Ac phosphorique + base purique
(Adénine ou Guanine) ou base pyrimidique (Cytosine ou
Thymine))
•A= T
•C = G
•...AGAGTCGTCTCGAGTCA...
•...TCTCAGCAGAGCTCAGT...
• L’arrangement des 4 bases permet d’établir le code
génétique qui supporte d’information dont la cellule a
besoin pour synthétiser les protéines
•Se fait sous forme de « codon » = triplet de bases
Liaisons hydrogène

 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
1
/
52
100%