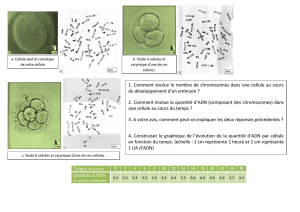
Cytogénétique moléculaire et DPI par FISH

G49-50 1/14
G49-50 Philippe Gosset Pauline Amrouni et Marie Freund
Cours du 23/11/12 de 16h à 18h
M1 Physiopathologie
Cytogénétique moléculaire et DPI par FISH
La cytogénétique classique permet d’analyser les chromosomes vus au microscope. En
cultivant des cellules et en les arrêtant en métaphase, on peut faire des analyses du
génome. On colore les chromosomes au Giemsa en général. La cytogénétique permet de
voir des anomalies intermédiaires (de l’étude d’un gène moyen à celle d’un chromosome
entier). On peut également utiliser des techniques particulières pour voir les chromosomes
(bande G, bande R, haute résolution…)
La cytogénétique classique pourra faire apparaitre 400 bandes de quelques Mb, la haute
résolution montrera 850 bandes (5 Mb minimum). Ces deux techniques pourront permettre
l’étude des anomalies de nombres des chromosomes et certaines anomalies de structures.
La biologie moléculaire comprend différentes méthodes et aura en général une résolution
d’une Mb à quelques centaines de bases. On y trouve :
- la PCR : résolution inférieure une centaine de bases. On peut étudier des gènes
(fragments de délétions et de fusions..), ou encore des microsatellites (analyse des
liaisons, recherche de disomie uniparentale)
- le séquençage : résolution d’une base, il permet notamment la recherche des
mutations.
La cytogénétique moléculaire permet de faire des peintures des chromosomes (résolution de
quelques Mb). Elle comprend aussi la technique de FISH (résolution qui dépend de la taille
de la sonde : du chromosome a plusieurs Mb jusqu'à quelques Kb (voire une base). Elle
permet l’analyse des remaniements et des
micro-remaniements.
Selon la résolution recherchée et selon ce que
l’on recherche on préférera une méthode a une
autre.
Pour information quelques points
Historiques :
- découverte des chromosomes : Tjio et
Levan, 1956
- utilisation des Bandes et de la FISH :
• Casperson et coll., 1970
• Dutrillaux et Lejeune, 1971
-Hybridation in situ radioactive :
• Gall et Pardue, 1969
-Hybridation in situ Fluorescente :
• Bauman et coll., 1980
Lichter et coll., 1988 (application) Pinkel et coll., 1988

G49-50 2/14
I. LA CYTOGENETIQUE MOLECULAIRE
A. La technique
Elle repose sur deux principes :
- les propriétés de l’ADN (double hélice à base complémentaire avec un ordre
particulier), on va cibler, choisir la séquence à marquer.
- Les techniques d’imagerie : radioactivité, molécule fluorescente, microscope à
épifluorescence
L’ADN possède quatre bases disposées en double hélice, relié par des ponts hydrogènes.
Son diamètre est d’environ 2nm (donc invisible en microscope optique).
L’hybridation in situ repose sur la complémentarité de l’ADN.
Pour faire de l’hybridation in situ on doit d’abord séparer les deux brins d’ADN pour qu’il
puisse exposer leurs bases à la sonde (c’est la dénaturation).
La cible correspond à l’ADN simple brin, elle est complémentaire à la sonde marqué (ADN,
ARN, système protéique..). La reconnaissance de la cible avec la sonde est appelé
l’hybridation.
Pour info : la première tentative d’hybridation s’est faite avec de l’adn tritié après un
étalement chromosomique. La sonde était du tritium radioactif, après l’hybridation on lavait
pour enlever ce qui n’était pas fixé. On posait un film photographique sur la lame et on
pouvait analyser l’image.
Les molécules fluorescentes :
Elles sont utilisées pour que l’on puisse voir l’adn. Ce sont des molécules naturellement
fluorescentes sous une excitation (irradiation) de longueur d’onde plus courte que celle vue à
la réémission. Les cellules perdent de l’énergie donc la longueur d’onde augmente à la
réémission.
Par exemple on a la fluoréscéine qui a une excitation pour une lumière bleue-verte et une
émission dans le vert.
L’œil ne voit que les longueurs d’onde entre 400 et 700 nm (en sachant que 8% des
hommes ont un déficit des couleurs). Il faut donc que les molécules émettent dans le
domaine du visible mais certaines nécessitent une analyse informatique, en effet elles
émettent dans l’infra rouge ou le domaine UV. Il faudra donc sélectionner suffisamment de
lumière pour activer la fluorescence mais sans perturber l’image. En France on colore en
bleu, dans les pays anglo-saxons plutôt en rouge.
Pour info : la courbe de fluorescence de la fluoréscéine : elle absorbe la lumière de 495nm et
réemet à 515-525 nm.

G49-50 3/14
B. Le microscope à épifluorescence
On a un premier filtre qui sélectionne la
lumière qui excite la fluorescence. Cette
lumière rebondit ensuite vers la lame grâce à
un miroir dichromique (élément qui fait miroir
sous un angle et qui est transparent selon un
autre angle (laisse passer la lumière). La
lumière arrive donc à la lame, la
fluorescence s’active et passe au travers du
miroir dichromique qui cette fois agit comme
un élément transparent et non plus comme
le miroir. Elle passera au travers d’un
deuxième filtre et elle sera vue.
Résumé : source=>1er filtre=> miroir
dichromique=>lame=> miroir dichromique=>2ème filtre=> œil.
On peut utiliser plusieurs molécules fluorescentes en même temps grâce à un système de
filtre qui laisseront passer que la lumière d’excitation dans un sens et celle de la réémission
dans l’autre sens.
II. LES SONDES
A. Nature des sondes
on peut trouver :
- de l’adn (génomique total (CGH), des hybrides somatiques, de l’adn cloné (plus ou
moins spécifique et de tailles variables), des oligonucléotides..)
- de l’arn
- des systèmes protéiques
remarque : les hybrides somatiques sont des cellules hybrides entre 2 espèces, c’est
instable car on perd des chromosomes qui sont jugés inutiles par la cellule. On fait la
sélection une fois les cellules dans des puits. On prend un hybride et on la cultive.
Exemple : hybride entre des cellules de rongeurs et d’hommes : on perd des chromosomes
humains dans ce cas.
On peut faire de la peinture chromosomique également, soit partielle soit totale.
La peinture spécifique peut cibler un locus, plusieurs locus, des télomères (région
télomérique ou sub-télomérique), les centromères, les chromosomes acrocentriques (dans
lesquels on trouve de l’adn sans gènes donc non codante, mais on a des séquences
répétées).
On peut trouver des sondes dans le commerce :
- des centromères
- des télomères
- spécifiques à des locus (micro délétion, remaniement ou gêne de fusion)
On peut également fabriquer nos propres sondes :
- idem à celles du commerce plus celles fabriquées à partir de l’ADN du patient (CGH
array et puces à ADN)

G49-50 4/14
B. La fabrication
Pour préparer de nouvelles sondes, on doit effectuer plusieurs étapes :
- la sélection d’un clône ou un criblage
- faire une culture des clônes
- extraire l’ADN
- effectuer une vérification (STS, profil de restriction) pour vérifier que l’on a bien l’ADN
que l’on cherche
- effectuer une marquage
- test par la FISH pour la validation de la sonde
Pour fabriquer un clone, on incorpore une molécule d’ADN spécifique dans une cellule hôte
(souvent une bactérie) puis on la met en culture.
Plusieurs types de fragment d’ADN peuvent être incorporés dans une cellule hôte :
- un hybride somatique (dans une cellule hôte de mammifère)
- YAC (dans une levure), pour les ADN de grande taille (~1M paires de bases)
- PAC, BAC, cosmides, phages, plasmides (dans des bactéries)
C. Le principe du marquage
Pour rendre visible l’ADN, il faut incorporer un nucléotide marqué (dNTP) par :
- un haptène (petite molécule comme par exemple la biotine ou la digoxigénine),
facilement manipulable
- un fluorochrome
- un agent radioactif
- un enzyme sur l’ADN
Pour la visualisation de ces nucléotides marqués, il existe plusieurs techniques (la PCR, nick
translation, random prining)
On va extraire l’ADN double brin du clone et faire agir un enzyme pour couper un des deux
brins au hasard : la DNase1
Puis la DNApolymérase1 va venir recopier l’autre brin pour recréer la partie précédemment
coupée. Pour cela, elle a besoin de nucléotide, et utilisera donc les nucléotides marqués que
l’on aura au préalable intégrés dans la solution.
Attention : La DNApolymérase1 est également une exonucléase, et donc agrandi le trou
formé dans l’ADN.
III. LES METHODES D’ANALYSE
A. La FISH (hybridation in situ fluorescente)
Elle montre les petites anomalies.
Technique :
- dénaturation : On a un tube avec la solution de la sonde fluorescente (souvent de
l’ADN double brin), et une lame avec la cible ; on dénature les deux éléments en
chauffant à 95°C pour former de l’adn simple brin.
- Hybridation : on mélange le tube et la lame en contrôlant la température et en
ajoutant des agents dénaturants (pour éviter les températures trop élevées : 37°C au
lieu de 95). Le formamide est souvent utilisé comme agent dénaturant. C’est grâce à
cela que l’on va contrôler la spécificité de l’hybridation
- Lavage : on laisse agir quelques heures et on élimine les sondes non fixées.
- Révélation : soit la fluorescence est déjà présente et l’on fait directement une
lecture, soit on doit procéder à une révélation pour l’étude avec possibilité
d’amplification.

G49-50 5/14
Exemple : La biotine avec une sonde à l’avidine On peut rajouter un anticorps à
biotine avidine pour amplifier la fluorescence.
B. La PRINS (PRimed IN Situ labelling)
Il faut synthétiser de l’ADN marqué à partir d’une amorce in situ (la PCR nécessite 2
amorces). On peut avoir une amorce spécifique d’un site, annealing, un allongement avec
incorporation de dUTP marqué à la fluorescence (nécessitant de travailler à 72°C).
Il faut attendre une heure d’extension, on lave et on monte. C’est simple, rapide et sensible,
mais l’on a qu’un seul site de visualisation à la fois.
C. La fiber FISH
C’est une technique de FISH sur une fibre à
ADN « peignée » (étirée). Il faut étaler l’ADN
pour allonger les fibres (on a un ADN de haut
poids moléculaire en suspension), on fait
réagir la sonde. La technique est ensuite
semblable à la FISH.
L’ADN étiré permet d’analyser sur de petites
distances (de l’ordre du kb), les fibres sont
régulièrement étirées, ce qui permet une
précision sur la distance, mais certaines
visualisations sont impossibles, comme celles
des délétions.
La fiber FISH est réservée pour la
cartographie et l’analyse des micro-
duplications.
D. La CGH (Hybridation génomique comparative)
On marque l’ADN du patient
d’une couleur et un ADN témoin
d’une autre couleur.
L’hybridation est compétitive
sur des métaphases normales.
On incorpore de l’ADN
compétiteur qui va bloquer les
régions répétées (satellite,
télomère, séquence Alu = trace
d’un rétrovirus qui a été actif
dans la lignée humaine et est
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
1
/
14
100%