Cours N°7, « Réplication - Département de Biochimie

Biologie Cellulaire
&
Biologie Moléculaire
L3 UE 5.2 2013-2014
Cours n° 7 : Mardi 8 octobre 2013
186
III • Réplication
IV • Cycle cellulaire
Cours 7 BC&BM 2013-2014

Avancement du Cours
I Introduction
II Chromosomes
• III Réplication
– Contrôle de l’activation des origines
• Activation unique
• Organisation spatiale
• Activation moléculaire des origines
• Initiation de la réplication
• IV Cycle Cellulaire
• V Transcription
187 Cours 7 BC&BM 2013-2014

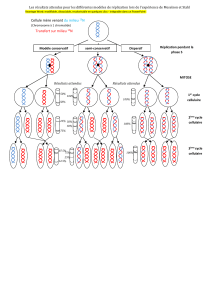
Chaque origine est activée une fois
et une fois au plus
188
Non totalement synchronisées, mais toutes les origines activées
doivent avoir fonctionné correctement 1 seule fois
P 145
contrôle contrôle contrôle Chap IV
Cycle
Cellulaire
Cours 7 BC&BM 2013-2014

Foyers réplicatifs : hypothèse
• Les origines sont organisées en foyers (ordre de 1000/
noyau)
• Chaque foyer peut contenir de 10 à 100 origines de
réplication.
• Ces usines à réplication comprennent les protéines
nécessaires à l’élongation de la réplication.
• Ces foyers peuvent correspondre à des territoires
chromatiniens stables.
• Chaque foyer est constitué de réplicons (unité de
réplication) synchronisés
189
P 146
Cours 7 BC&BM 2013-2014

Organisation spatiale des origines et stress
réplicatif
190
Blocage du
cycle cellulaire
Contrôle de l’activité des origines
P 147
ATR/Chk1
Kinases de signalisation
du système de contrôle de
la réplication
Inhibition de la réplication
Stress réplicatif
ou DSB activent
Cours 7 BC&BM 2013-2014
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
1
/
24
100%