projet-clostr-difficile

3/05/13 15:17Département des Sciences des Denrées alimentaires - ULg - Faculté de Médecine vétérinaire
Page 1 sur 1http://www.dda.ulg.ac.be/pages/fr/rech15.php
Clostridium difficile
Retour
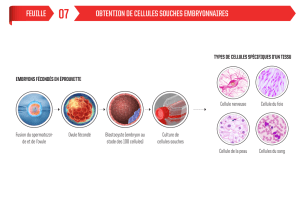
Mise en évidence du potentiel de transmission à l´homme de souches épidémiques de
Clostridium difficile par la consommation de denrées alimentaires d´origine animale
Clostridium difficile est actuellement la première cause de diarrhées ou colites nosocomiales suite à un
traitement antibiotique. Au cours de 5 dernières années, l’incidence et la sévérité de l’infection ont
augmenté progressivement. De plus, cette augmentation concerne aussi les infections à caractère
communautaire car non liées à un séjour hospitalier.
D’autre part, cette bactérie est responsable de pathologies intestinales chez l’animal, notamment chez le
porcelet et le veau, animaux destinés à rentrer dans la chaîne alimentaire.
Quelques études ont aussi identifié la bactérie dans les aliments d’origine animale, ce que donne la
possibilité d’une transmission via la chaîne alimentaire.
Ce projet essai de répondre à la question de si les infections à Clostridium difficile (CDI) humaines peuvent-
elles avoir pour origine une contamination alimentaire. Identifier ces problèmes de contamination apportera
de nouveaux éléments à la gestion des CDI en milieu hospitalier et communautaire mais aussi dans la
gestion de la contamination bactérienne dans la chaîne alimentaire.
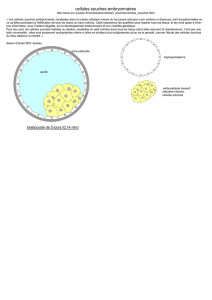
La première partie du travail consiste en l’adaptation et le développement de la méthode de détection de
Clostridium difficile sur des échantillons de bovin et porcin et la recherche de souches à partir de matières
fécales, des écouvillons de carcasses et des viandes fraîches de ces deux filières animales.
Toutes les souches isolées seront caractérisées au niveau moléculaire : PCR espèce (gène tpi) pour
confirmer que la colonie isolée se correspond à Clostridium difficile, PCR toxine pour mettre en évidence
leurs mécanismes de virulence (production des toxines A, B et CDT ou toxine binaire) et PCR ribotype afin
de les comparer entre elles.
Ces différentes méthodes microbiologiques et moléculaires développées seront utilisées pour isoler une
collection de souches alimentaires et pour les comparer et déterminer la parenté avec les souches
épidémiques humaines retrouvées en Belgique. Pour cette étape et pour le ribotypage de souches isolées il
sera important de travailler en collaboration avec le laboratoire de référence en Belgique (Prof Michel
Delmée UCL-MBLG) que possède une collection représentative de souches isolées durant les trois
dernières années dans toutes les régions du pays ainsi que dans beaucoup des pays européens.
Avec ce projet, il sera possible d’estimer le portage animal de Clostridium difficile en Belgique et l’état de
contamination des denrées alimentaires associées ainsi que le risque que ça représente pour la santé
humaine.
Collaboration
Projet ZOODIF (Professeur Michel Delmrée, UCL-MBLG)
Financement
SPF Santé publique
Contact
Prof. Georges Daube - Tél +32 (0) 4 366 40 15
© 2009, ULg - Faculté de Médecine vétérinaire - Département des Sciences des Denrées alimentaires. All rights reserved - E-mail info
Accueil
Historique
Recherche
Enseignement
Prestations
Publications
Personnel
Liens
Contacts
Accès
Evénements
Unité pilote
1
/
1
100%