epissageFichier DOC - e

UN GENE, DEUX PROTEINES
On s’intéresse à l’expression d’un gène appelé CGRP (Calcitonin Gene Related Product)
Ce gène est situé sur le chromosome 11. Il s’exprime dans les cellules C de la thyroïde où il code pour une hormone,
la calcitonine, intervenant dans la régulation de la calcémie (hormone hypocalcémiante). Il s’exprime aussi dans de
nombreux neurones du système nerveux central et périphérique où il code pour un neuromédiateur, le CGRP.
Calcitonine et CGRP ont des rôles physiologiques différents. C’est donc l’exemple d’un gène qui code pour deux
protéines différentes suivant le type de cellules où il s’exprime.
Projet de travail : Déterminer comment un même gène peut être à l’origine de deux protéines différentes.
Support 1 : un ARN pré-messager chez les eucaryotes
La synthèse des protéines est le processus par lequel une cellule assemble les acides aminés isolés présent dans son
cytoplasme en une chaîne protéique, à partir de l'information contenue dans l'ADN. Elle se déroule en deux étapes au
moins : la transcription de l'ADN en ARN messager et la traduction de l'ARN messager en une protéine.
Chez les eucaryotes, il y a une étape intermédiaire, la production par transcription d’un ARN pré-messager, qui se
passe dans le noyau. L'ARN pré-messager va subir un certain nombre de modifications (dont l’accrochage d’une
queue poly A). A l’issue de ces modifications, il est arrivé à maturation et prend alors le nom d'ARN messager. C’est
cet ARN messager qui est utilisé pour la traduction.
Support 2: utilisation du logiciel anagène
Choisir Démarrer
tous les programmes
SVT
anagène
anagène code 1 lettre
fichier
ouvrir
dossier
CGRP
On dispose de la séquence du gène (gene-CALCA.adn), de celle de l’ARN pré-messager et des deux ARN messagers,
celui présent dans les cellules de la thyroïde (ARNm2) et celui présent dans les neurones (ARNm1).
Utilisez les fonctionnalités (comparaison de séquences (ADN et ARN prémessager) et dot plot (ARN prémessager et
ARNm1 et ARNm2)) du logiciel pour comparez les différentes séquences entre elles.

corrigé
Le support 1 indique que chez les eucaryotes, la transcription est à l’origine d’un ARN pré-messager qui subit des
modifications aboutissant à la formation d’un ARN messager qui sera utilisé pour la traduction et sera à l’origine
d’une chaîne polypeptidique.
Le support 2 nous permet de comprendre certaines des modifications subies par l’ARN pré-messager.
La comparaison (avec discontinuité) de l’ADN et de l’ARN pré-messager indique que chaque nucléotide de l’ADN a
été transcrit dans l’ARN pré-messager.
La fonction dot plot permet de comparer les molécules d’ARN pré-messager et d’ARN messager nucléotide par
nucléotide. Elle donne les deux graphiques suivant :
On s’aperçoit que les séquences d’ARNm sont plus petites que les séquences d’ARN-pré-messager. Il y a donc eu
perte de parties de l’ARN pré-messager au cours de la maturation.
D’autre part, les zones de l’ARN pré-messager que l’on retrouve dans les ARNm sont différentes entre l’ARNm1 et
l’ARNm2 : 4 zones pour l’ARNm2 et 5 zones pour l’ARNm1.
On peut donc penser que l’une des modifications subies par l’ARN pré-messager est un découpage de la séquence
de nucléotides qui est différent dans les cellules de la thyroïde et dans les neurones.
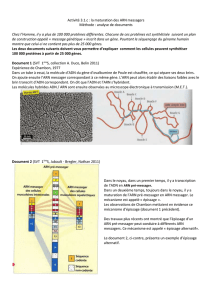
On peut schématiser les modifications subies dans le cas étudié :

Ce découpage de l’ARN pré-messager s’appelle l’épissage. Comme il peut être différent entre les types cellulaires
pour un même gène, on le qualifie d’alternatif. Les parties conservées pour former de l’ARNm s’appellent des exons.
Les parties qui ne sont jamais conservées dans l’ARNm s’appellent des introns.
1
/
3
100%