Utilisation des marqueurs pour la gestion de la variabilité

E. VERRIER1,2 , X. ROGNON1,3
1INA Paris-Grignon, Département des Sciences
Animales, 16 rue Claude Bernard, 75231 Paris
cedex 05
2 INRA, Station de Génétique Quantitative et
Appliquée, 78352 Jouy-en-Josas cedex
3 INRA, Laboratoire de Génétique des Poissons,
78352 Jouy-en-Josas cedex
e-mail : [email protected]
Utilisation
des marqueurs pour
la gestion de la
variabilité génétique
des populations
7 - Utilisation des marqueurs
génétiques
Résumé. Les méthodes usuelles de gestion de la variabilité génétique des populations reposent
sur la connaissance des généalogies des animaux et sur un contrôle de la structure familiale de
ces populations. Les marqueurs permettent d’affilier les animaux à des familles dans les situa-
tions où il est malaisé ou impossible d’enregistrer un état civil, donnant ainsi accès aux
méthodes classiques de gestion de la variabilité. Ils procurent des outils de suivi de la variabi-
lité intra-population, fondés sur les fréquences alléliques. Enfin, on peut choisir les reproduc-
teurs présentant le plus de variabilité pour un ensemble de marqueurs. Les études effectuées
dans ce sens montrent qu’une telle sélection est très efficace pour les régions du génome proches
des marqueurs employés, mais qu’elle doit être réalisée en complément des méthodes usuelles si
l’on s’intéresse à l’ensemble du génome.
INRA Prod. Anim., 2000, numéro hors série « Génétique moléculaire : principes et application aux populations animales », 253-257.
La gestion à plus ou moins long terme des popu-
lations implique que l’on se préoccupe du devenir
de leur variabilité génétique (variabilité intra-popu-
lation). Cela vaut pour les races en conservation
dont les effectifs de reproducteurs sont, par nature,
limités, ainsi que pour les souches expérimentales
qui peuvent également être dans ce cas. L’objectif
est alors généralement de préserver une variabilité
«patrimoine» et de limiter les inconvénients zoo-
techniques liés à une érosion de celle-ci. Cela vaut
aussi pour les races ou lignées sélectionnées à une
échelle commerciale : des études montrent en effet
que de telles populations ont des bases génétiques
fort étroites du fait de la concentration des efforts
de sélection sur un nombre de reproducteurs de
plus en plus restreint. L’objectif est alors de préser-
ver la variabilité afin d’autoriser la poursuite du pro-
grès génétique sur des caractères d’intérêt écono-
mique déjà sélectionnés ou la reconversion des
objectifs de sélection vers des caractères peu ou
pas pris en compte aujourd’hui. Après avoir rappelé
les principaux facteurs d’érosion de la variabilité
génétique et les méthodes disponibles pour limiter
ce type d’évolution, nous verrons en quoi les mar-
queurs peuvent constituer une aide à la gestion de la
variabilité génétique des populations.
1 / Evolution et contrôle de la
variabilité génétique dans les
populations d’effectif limité
Lorsque les effectifs d’une population sont limi-
tés, le passage d’une génération à une autre consti-
tue un échantillonnage des gènes, ce qui provoque
en un locus quelconque des fluctuations des fré-
quences alléliques pouvant conduire, du seul fait du
hasard, à la perte de certains allèles et, à terme, à la
fixation d’un seul allèle (dérive génétique). De façon
concomitante, l’apparentement entre les reproduc-
teurs, et donc la consanguinité de leurs descen-
dants, s’accroissent au fil du temps, ce qui induit
une augmentation de l’homozygotie sur l’ensemble
du génome. Le facteur-clef de cette évolution est la
taille efficace de la population, ou son effectif géné-
tique. On montre que dans une population panmic-
tique, fermée et de taille limitée, le coefficient de
consanguinité moyen (F, qui varie de 0 à 1) s’accroît
au même rythme que décroît le taux d’hétérozygotie
(H) à un locus neutre sans mutation, rythme d’au-
tant plus élevé que l’effectif génétique (Ne) est petit.
Soit, en mettant en indice le numéro de génération
(t), et en considérant une population non consan-
guine au départ, l’équation décrivant ce phénomène
et qui est illustrée à la figure 1 :
Ht1 t
–– = 1- Ft =
(
1- –––
)
(1)
H02Ne
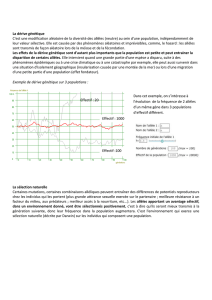
Figure 1. Evolution de l’hétérozygotie résiduelle en un
locus neutre (Ht/ Ho, en %) sous l’effet de la dérive, selon
l’effectif génétique (Ne) de la population (cf équation 1).

254 / E.VERRIER, X. ROGNON
INRA Productions Animales, 2000, hors série Génétique moléculaire
L’effectif génétique d’une population dépend des
effectifs de reproducteurs, mâles et femelles, et
croît avec ceux-ci. Il dépend aussi du déséquilibre
des tailles de descendance. Lorsque les descen-
dances sont de tailles uniformes, chaque reproduc-
teur contribue de la même façon à l’évolution du
polymorphisme. A contrario, en cas de déséqui-
libre, les allèles portés par les reproducteurs les
plus féconds sont favorisés au détriment des autres,
et l’effectif génétique est plus faible.
A nombre égal de reproducteurs, en comparaison
de la dérive pure, la sélection modifie la structure
familiale et renforce le déséquilibre des tailles de
descendance. En effet, deux individus plus appa-
rentés entre eux que la moyenne auront des valeurs
respectives pour le critère de sélection se ressem-
blant plus que celles de deux individus pris au
hasard : ils auront donc plus de chances d’être
simultanément sélectionnés ou rejetés. La sélection
renforce ainsi l’apparentement entre les reproduc-
teurs sélectionnés, de même qu’elle renforce le
déséquilibre des tailles de descendance de leurs
parents : les «bons» parents ont des descendances
plus nombreuses, les «mauvais» parents des des-
cendances moins nombreuses. La sélection joue par
ailleurs sur la variabilité aux locus gouvernant le(s)
caractère(s) sélectionné(s), par l’induction d’un
déséquilibre d’association entre gènes, d’une part,
et par une tendance à la fixation des allèles «favo-
rables», d’autre part. Ces deux phénomènes vont
dans le sens d’une diminution de la variabilité, à
court terme pour le premier, à long terme pour le
second.
Les méthodes permettant de limiter la diminu-
tion de variabilité génétique sont bien connues
(voir, par exemple, Verrier 1992). La première
d’entre elles, de loin la plus efficace, est d’équili-
brer les tailles de descendance. La solution idéale
de ce point de vue consiste à retenir de chaque
père un nouveau mâle reproducteur et un seul et le
même nombre de nouvelles reproductrices, et de
chaque mère une nouvelle reproductrice et une
seule (choix intra-famille). D’autres méthodes plus
ou moins élaborées existent. Pour les races en
conservation, des schémas d’accouplement fondés
sur une subdivision en groupes de reproduction
ont été mis en œuvre et fonctionnent efficacement.
En cas de sélection, toute une série de méthodes
visant à trouver un compromis entre progrès géné-
tique immédiat et préservation de la variabilité ont
été proposées (voir Verrier et al 1994) : diminution
du poids de l’information familiale dans les indices
de sélection, restriction sur le nombre de pleins
frères-sœurs sélectionnés, sélection pour un critè-
re combinant valeur génétique et apparentement
moyen, etc.
2 / Les marqueurs comme outil
pour l’établissement et/ou le
contrôle de l’état civil
Comme cela vient d’être discuté, gérer la variabi-
lité des populations nécessite la connaissance de la
structure familiale et donc de la généalogie des indi-
vidus. Pour des raisons pratiques, dans certaines
situations, il n’est pas possible de contrôler les
unions et/ou d’établir l’état civil. Chez les mammi-
fères, c’est le cas, au moins pour les paternités, des
espèces sauvages et des espèces domestiques éle-
vées en système extensif avec plusieurs mâles de
monte naturelle dans un même lot de femelles. C’est
le cas des espèces aquacoles, pour lesquelles le seul
moyen d’établir les filiations est de maintenir dans
des bassins séparés les pontes des différents
couples, au moins jusqu’à l’âge où un marquage phy-
sique est possible, ce qui, outre le surcoût que cela
représente, a de fâcheuses conséquences pour
l’analyse génétique et la sélection (confusion des
effets «famille» et «bassin», absence de compétition
entre familles, etc).
L’efficacité des contrôles de filiation dépend du
nombre de marqueurs utilisés pour ces contrôles et
de leur polymorphisme. L’usage des microsatellites
permet de réduire le nombre de marqueurs pour
une même fiabilité (Amigues et al 2000, cet ouvra-
ge). Ces marqueurs permettent aussi d’établir les
filiations (assignation de parenté) lorsque, au lieu
d’avoir une quasi certitude quant à la maternité
(mammifères), on ne dispose que de la liste des
pères et des mères potentiels (espèces aquacoles).
Des études théoriques et par simulation (San
Cristobal et Chevalet 1997) ont montré l’efficacité
de la méthode, surtout lorsque l’on intègre dans les
calculs les probabilités de mutation et d’erreur de
typage. Sur cette base, des jeux de microsatellites sont
définis en vue de protocoles routiniers d’assignation
de parenté (Estoup et al 1998). De tels protocoles,
avec automatisation des typages par LABOGENA,
sont actuellement mis en œuvre chez le turbot (6
microsatellites) et sont en cours de développement
chez le bar et les salmonidés. L’objectif est de dis-
poser d’informations fiables quant aux généalogies,
afin d’appliquer au sein de populations sélection-
nées certaines des méthodes «classiques» de ges-
tion de la variabilité, l’aide des marqueurs étant
donc ici indirecte.
3 / Les marqueurs comme outil
de suivi de la variabilité
génétique
La disponibilité de marqueurs permet d’avoir une
mesure de la diversité des allèles et des combinai-
sons d’allèles, donc de la variabilité génétique aux
locus observés. Un premier indicateur est le nombre
total d’allèles pour un locus donné (richesse allé-
lique). Ce paramètre peut toutefois être sous-estimé
si le nombre d’individus typés est faible et le mar-
queur très polymorphe, les allèles rares ayant alors
très peu de chances d’être échantillonnés. A l’inverse,
il présente peu d’intérêt si le nombre maximal d’al-
lèles est par nature faible, comme pour les mar-
queurs de type RAPD (deux allèles). De plus, cet indi-
cateur ne tenant pas compte des fréquences, il donne
une vision incomplète des choses. Ainsi, plusieurs
indices fondés sur les fréquences alléliques ont été
proposés (pour une synthèse récente, voir Kremer
1995). Le plus utilisé a été généralisé par Nei sous le
nom d’indice de diversité. Il correspond à la probabi-
lité pour que deux gènes pris au hasard représentent
des allèles différents, et il est égal au taux d’hétéro-
zygotes (H) attendu sous les hypothèses de Hardy-
Weinberg. Selon la même approche, on définit le
nombre efficace d’allèles (Ae) comme l’inverse de la
probabilité pour que deux gènes pris au hasard repré-
sentent le même allèle. En un locus donné, compor-

Utilisation des marqueurs pour la gestion de la variabilité génétique des populations / 255
INRA Productions Animales, 2000, hors série Génétique moléculaire
tant plusieurs allèles indicés i et de fréquences res-
pectives pi(Spi = 1), ces deux paramètres s’expri-
ment comme suit :
H= 1 - SPi2(2)
i
Ae= 1= 1 (3)
––––––– ––––––
SPi2(1 - H)
i
Si les fréquences sont toutes égales entre elles, le
nombre efficace d’allèles est égal au nombre total.
Dans les autres cas, le nombre efficace est inférieur
au nombre total : il est d’autant plus faible que les fré-
quences sont déséquilibrées. Les formules (2) et (3)
s’appliquent à tout type de marqueur, y compris au
niveau d’un site nucléotidique, les allèles étant alors
les 4 bases de l’ADN. L’estimation de ces deux para-
mètres est moins sensible aux aléas d’échantillonna-
ge, les allèles les plus fréquents étant ceux qui
«pèsent» le plus dans la valeur calculée. Avec plu-
sieurs locus, on calcule un taux d’hétérozygotie
moyen en faisant simplement la moyenne arithmé-
tique des taux observés pour chacun des locus.
Les indices de variabilité fondés sur les marqueurs
sont complémentaires de ceux fondés sur les généa-
logies et permettent de comparer différentes popula-
tions à un instant donné (tableau 1). Les valeurs pré-
sentées ici correspondent à des nombres efficaces
d’allèles de l’ordre de 1,5 à 1,6, c’est-à-dire que la
variabilité aux 16 marqueurs considérés est inférieure
à celle que l’on obtiendrait avec des locus à deux
allèles équiprobables.
Il est utile d’établir l’évolution dans le temps des
indices de variabilité génétique. Ainsi, l’évolution
observée du taux d’hétérozygotie (2) permet d’esti-
mer l’effectif génétique réalisé d’une population (1).
Des méthodes plus élaborées, fondées sur les varia-
tions de fréquences alléliques ou l’instauration de
déséquilibres d’association entre locus, ont également
été proposées dans ce but. Une difficulté est de dis-
poser de suffisamment de recul. Dans l’étude de
Moureaux et al (1996) évoquée plus haut, fondée sur
des marqueurs modérément polymorphes, l’informa-
tion disponible représentait à peine deux générations
équines, ce qui était nettement insuffisant pour détec-
ter des tendances significatives. L’emploi de mar-
queurs beaucoup plus polymorphes (microsatellites)
et l’analyse de populations où des goulots d’étrangle-
ment bien plus sévères peuvent être observés
(espèces aquacoles, par exemple) permettraient sans
doute de détecter des évolutions significatives sur des
pas de temps raisonnablement courts. Une question
encore ouverte concerne le type de marqueurs qu’il
convient d’employer pour ce type d’analyse (neutres,
liés à ou étant eux-mêmes des locus gouvernant des
caractères d’intérêt), ainsi que leur nombre.
4 / Les marqueurs comme outil
de gestion de la variabilité
génétique
L’idée est d’utiliser directement l’information à
des marqueurs pour retenir des reproducteurs per-
mettant d’espérer une variabilité génétique maxima-
le à la génération suivante, indépendamment ou en
complément des méthodes fondées sur la connais-
sance des généalogies, dans le cadre de populations
sélectionnées pour des caractères quantitatifs ou
non (la même approche peut être retenue afin de
choisir les individus donneurs de semence ou d’em-
bryons pour une cryobanque).
4.1/ Situations sans sélection
Nous considérons ici le cas de populations pour
lesquelles l’objectif principal est le maintien de la
variabilité, notamment celui des races en conserva-
tion. Après les premières études de Chevalet et
Rochambeau (1986) et Chevalet (1992), des travaux
plus systématiques ont été récemment entrepris par
Toro et al (1998 et 1999). Les méthodes proposées
ont été testées par simulation, les critères de juge-
ment de leur efficacité étant, d’une part, l’évolution
des probabilités d’identité des gènes et du taux d’hé-
térozygotie à des locus simulés répartis sur l’en-
semble du génome ainsi qu’aux marqueurs eux-
mêmes et, d’autre part, l’évolution du coefficient de
consanguinité moyen calculé à partir des pedigrees
(rappelons que ce dernier paramètre permet de
décrire l’évolution probable du polymorphisme en
un locus neutre quelconque). Nous tentons de déga-
ger ci-après les principaux résultats de ces études.
Nous nous plaçons toujours dans une situation où
le nombre de reproducteurs est fixe et où les repro-
ducteurs de renouvellement sont à choisir parmi un
plus grand nombre de candidats. Plusieurs
méthodes sont alors envisageables pour valoriser
l’information disponible à des marqueurs :
- retenir comme reproducteurs les individus qui
ont le plus grand nombre de marqueurs à l’état hété-
rozygote (méthode notée H sur la figure 2) ;
- retenir comme reproducteurs les individus por-
teurs, aux locus marqueurs, des allèles les plus
rares (méthode notée R). Pour cela, on peut calcu-
ler un critère de choix inversement proportionnel
au produit des fréquences des allèles portés, ou pro-
portionnel au produit des compléments à 1 de ces
fréquences ;
- retenir le groupe d’individus qui présente le taux
d’hétérozygotie moyen maximal (méthode notée G).
Le taux d’hétérozygotie est établi à partir de l’équa-
tion (2), en faisant la moyenne sur l’ensemble des
marqueurs et en considérant les fréquences allé-
liques à l’intérieur du groupe de reproducteurs rete-
nus.
Les deux premières méthodes sont simples à
mettre en œuvre. Elles autorisent un choix des
reproducteurs parmi l’ensemble des candidats, sans
prise en considération de leur origine familiale, ou à
l’intérieur de chacune des familles (contrainte notée
W), ce qui suppose une connaissance de la structu-
re familiale. La dernière méthode est plus lourde car
il faut comparer toutes les combinaisons possibles
de mâles et de femelles parmi l’ensemble des candi-
dats disponibles (il existe des procédures itératives
permettant d’obtenir des solutions approchées si le
Tableau 1. Taux d’hétérozygotie moyen à 16 marqueurs
sanguins au sein de cinq races françaises de chevaux
(animaux nés en 1992, la taille des échantillons varie de
500 à 5 000 environ). D’après Moureaux et al (1996).
Pur-Sang 0,33
Trotteur Français 0,39
Arabe 0,35
Anglo-Arabe 0,32
Selle Français 0,38

256 / E.VERRIER, X. ROGNON
INRA Productions Animales, 2000, hors série Génétique moléculaire
temps de calcul est un facteur limitant). Cette troi-
sième méthode peut, au prix de complications sup-
plémentaires, être appliquée en combinant l’infor-
mation aux marqueurs et l’information généalo-
gique quand celle-ci est connue. Il convient alors de
calculer, au sein de chaque groupe considéré, la
parenté moyenne conditionnée par les génotypes
aux marqueurs et les généalogies, et de retenir le
groupe avec la plus faible parenté moyenne.
L’ensemble des travaux effectués permet de déga-
ger quelques tendances générales, présentées ci-
dessous et en partie illustrées à la figure 2.
Parmi les deux méthodes simples, favoriser les
porteurs d’allèles rares est plus efficace que favori-
ser les hétérozygotes. Ceci est d’autant plus vrai que
le nombre de marqueurs est élevé, au moins dans la
gamme de variation considérée ici.
L’effet est encore plus important et l’avantage de
la méthode «allèles rares» est encore plus net si, au
lieu de considérer l’ensemble du génome, on s’inté-
resse aux marqueurs eux-mêmes et/ou aux zones
proches de ces marqueurs. Ce point est important à
considérer si l’on souhaite préserver la variabilité
sur une zone spécifique du génome ou en des locus
bien particuliers.
Ces deux méthodes contribuent cependant à
modifier la structure familiale, surtout si la pression
de sélection est forte. En effet, en un locus donné,
choisir des hétérozygotes augmente la probabilité
de retenir des reproducteurs issus de la même
famille (par exemple une famille dont le père et la
mère sont homozygotes pour des allèles différents).
Cet impact est encore plus marqué lorsque l’on
favorise les allèles rares. Ainsi, si l’on n’adopte pas
de contrainte sur la structure familiale (voir plus
loin), il est préférable de ne pas exercer une pres-
sion de sélection trop forte sur les critères liés aux
marqueurs, c’est-à-dire qu’il peut y avoir avantage à
ne pas forcément typer tous les jeunes mais seule-
ment certains choisis au hasard : l’intérêt génétique
rejoint alors l’intérêt économique.
L’imposition de contraintes sur la structure fami-
liale (ici, cas extrême du choix intra-famille) accroît
très nettement les possibilités de préservation de la
variabilité génétique, car elle permet d’éviter l’effet
de co-sélection souligné plus haut (cf figure 2). Ceci
est d’ailleurs vrai, on le savait déjà, en absence de
marqueur (méthode R+W, points d’abscisse zéro).
La méthode consistant à maximiser l’hétérozygo-
tie du groupe de reproducteurs retenus se révèle
très efficace aussi mais, à nombre d’allèles par mar-
queur constant, légèrement moins que la méthode
«allèle rares» appliquée à un choix intra-famille (cf
figure 2). Il est en fait nécessaire de combiner les
informations de généalogies et aux marqueurs pour
que l’optimisation de la variabilité du groupe
devienne la méthode la plus efficace de toutes
(résultats non montrés).
L’adoption des plans d’accouplement visant à
minimiser la consanguinité future permet de légers
gains d’efficacité par rapport à la panmixie (résul-
tats non montrés ici).
L’efficacité de toutes ces méthodes dépend de la
richesse de l’information représentée par les mar-
queurs. De ce point de vue, c’est une combinaison
du nombre de marqueurs et du nombre d’allèles par
marqueur qui semble jouer : par exemple, avec la
méthode «groupe à hétérozygotie maximale», 2
marqueurs à 10 allèles sont plus efficaces que 6
marqueurs à 4 allèles (cf figure 2).
En définitive, la solution la plus simple, optimale
ou sub-optimale sur le plan génétique, consiste à
choisir des reproducteurs porteurs d’allèles rares en
un nombre raisonnable de marqueurs polymorphes
répartis sur tous les chromosomes, tout en impo-
sant une contrainte sur la structure familiale.
4.2/ Situations avec sélection
Nous considérons ici le cas de populations
sélectionnées sur un ou plusieurs caractères.
L’existence de marqueurs pose deux types de
question. Tout d’abord, on peut envisager d’utili-
200
150
100 010
Nombre de marqueurs par chromosome
R10 + W
R4 + W
R4
H4
225
175
125 010
Nombre de marqueurs par chromosome
R10 + W
G10
R4 + W
G4
%
%
Taux d'hétérozygotie résiduelle
Effectif génétique réalisé
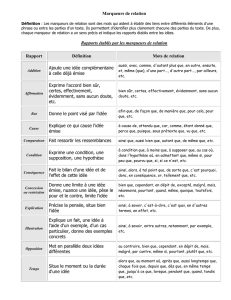
Figure 2. Effet du nombre de marqueurs par chromo-
some sur l’efficacité relative de méthodes visant à
préserver la variabilité génétique dans une population de
8 mâles et 24 femelles choisis parmi 72 candidats dans
les deux sexes (d’après Toro et al 1998 et 1999).
La variabilité est appréciée à 1900 locus répartis sur le
génome par le taux d’hétérozygotie résiduelle après 15
générations (figure du haut) et l’effectif génétique réalisé
calculé d’après le taux moyen d’élévation des probabil-
ités d’identité des gènes aux 1900 locus entre les
générations 6 et 15.
Mode de choix des reproducteurs (voir texte) : H =
hétérozygotes favorisés, R = porteurs d’allèles rares
favorisés, G = groupe avec hétérozygotie maximale ; le
chiffre à côté des initiales désigne le nombre d’allèles
par marqueur ; W = choix intra-famille.
Base 100 = dérive pure (choix des reproducteurs totale-
ment au hasard).

Utilisation des marqueurs pour la gestion de la variabilité génétique des populations / 257
INRA Productions Animales, 2000, hors série Génétique moléculaire
ser des marqueurs indépendants des caractères
considérés pour tempérer les effets de la sélec-
tion, en combinant index de valeur génétique et
critères de variabilité aux marqueurs. Il faudrait
déterminer les pondérations optimales à accorder
aux deux types d’information et établir le gain
d’efficacité espéré par rapport aux méthodes sans
marqueurs. Toutefois, les préoccupations
actuelles concernent surtout l’utilisation des mar-
queurs proches de QTL identifiés grâce à ces
mêmes marqueurs (Sélection Assistée par
Marqueurs ou SAM). Un des problèmes est d’opti-
miser la gestion de la variabilité au(x) QTL, d’une
part, et aux gènes non détectés (polygènes),
d’autre part (Manfredi 2000, cet ouvrage). Il s’agit
là d’un champ de recherche largement ouvert.
Signalons que la SAM peut être efficace pour évi-
ter la perte d’allèles «favorables» à des QTL jouant
sur des caractères secondaires. Des simulations
(Verrier 2000) concernant deux caractères généti-
quement antagonistes, l’un principal dans l’objec-
tif de sélection et de nature polygénique, l’autre
secondaire et gouverné par un QTL et des poly-
gènes, ont en effet montré qu’avec un marqueur
proche du QTL (taux de recombinaison de 2 %) et
quand la fréquence initiale de l’allèle «favorable»
est faible (0,1), la SAM permet, sans restreindre le
progrès génétique sur l’objectif global, de réduire
de près du tiers le risque de perte de cet allèle au
bout de 15 générations.
Conclusion
Les marqueurs peuvent être utiles pour préserver
la variabilité génétique des populations d’effectif
limité, que l’on s’intéresse à certaines zones du
génome ou à son ensemble. Les méthodes propo-
sées gagnent en efficacité si elles sont combinées à
des méthodes «classiques» jouant sur la structure
familiale. L’utilisation en pratique des marqueurs à
des fins de conservation demeure cependant encore
conditionnée à un abaissement des coûts de typage.
Des recherches sont encore nécessaires pour ce qui
est de leur utilisation en vue de combiner préserva-
tion de la variabilité et progrès génétique.
Remerciements
Les auteurs remercient M.Y. Boscher, J.C. Mériaux (LABOGENA),
C. Vauchez (SYSAAF), P. Le Roy, P. Sellier et H. de Rochambeau
(INRA) pour leurs informations et commentaires.
Amigues Y., Mériaux J.C., Boscher M.Y., 2000. Utilisation de mar-
queurs génétiques en sélection : les activités de LABOGENA.
INRA Productions Animales, numéro hors série « Génétique
moléculaire : principes et application aux populations ani-
males », 203-210.
Chevalet C., 1992. Utilisation de marqueurs pour la sauvegarde
de la variabilité génétique des populations. INRA Productions
Animales, Numéro hors série «Eléments de génétique quantitati-
ve et application aux populations animales», 295-297.
Chevalet C., Rochambeau H. de, 1986. Variabilité génétique et
contrôle des souches consanguines. Sciences et Techniques des
Animaux de Laboratoire, 11, 251-257.
Estoup A., Gharbi K., San Cristobal M., Chevalet C., Haffray P.,
Guyomard R., 1998. Parentage assignment using microsatellites
in turbot (Scophthalmus maximus) and rainbow trout
(Oncorhynchus mykiss) hatchery populations. Canadian
Journal of Fisheries and Aquatic Science, 55, 715-725.
Kremer A., 1995. Les marqueurs moléculaires en génétique des
populations. In : D. de Vienne (ed), Les marqueurs moléculaires
en génétique et biotechnologies végétales, 2ème édition, 119-138.
INRA, Paris.
Manfredi E., 2000. Intérêt et limites de la sélection intra-race
assistée par marqueurs. INRA Productions Animales, numéro
hors série « Génétique moléculaire : principes et application aux
populations animales », 239-242.
Moureaux S., Verrier E., Ricard A., Mériaux JC., 1996. Genetic
variability within French race and riding horse breeds from
genealogical data and blood marker polymorphisms. Genetics
Selection Evolution, 28, 83-102.
San Cristobal M., Chevalet C., 1997. Error tolerant parent identi-
fication from a finite set of individuals. Genetical Research, 70,
53-62.
Toro M., Silió L., Rodrigáñez J., Rodriguez C., 1998. The use of
molecular markers in conservation programmes of live animals.
Genetics Selection Evolution, 30, 585-600.
Toro M., Silió L., Rodrigáñez J., Rodriguez C., Fernández J., 1999.
Optimal use of molecular markers in conservation programmes.
Genetics Selection Evolution, 31, 255-261.
Verrier E., 1992. La gestion génétique des petites populations.
INRA Productions Animales, Numéro hors série «Eléments de
génétique quantitative et application aux populations animales»,
265-271.
Verrier E., 2000. Marker assisted selection for the improvement
of two antagonistic traits under mixed inheritence. Genetics
Selection Evolution (soumis).
Verrier E., Colleau J.J., Foulley J.L., 1994. Le modèle animal est-
il optimal à moyen terme ? In : J.L. Foulley, M. Molénat (eds),
Séminaire modèle animal, 26-29 septembre 1994, La-Colle-sur-
Loup, France, 57-66.
Références
 6
6
1
/
6
100%