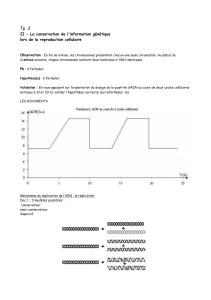
3 - AMPCfusion

UEx – Parcours Génétique
Cours n°3
29/10/2015
Caroline Nava
caroline.nava@aphp.fr
RT : SABRI Sophia
RL :
Maladies génétiques liées à des réarrangements
génomiques
Plan :
I. Classification des anomalies chromosomiques
II. Mécanismes de survenue des anomalies chromosomiques
A. NAHR non allelic homologuous recombination
B. NHEJ (non homologous end joining)
C. FoSTeS (Fork Stalling and Template Switching)
III. Méthodes d’exploration du génome
A. Le caryotype
B. La FISH
C. Les puces à ADN
IV. Réarrangements génomiques et pathologies humaines
A. CNV récurrents
B. CNV non récurrents
C. Pathologies A/R
D. Applications en recherche
V. Séquençage de nouvelle génération appliqué à la recherche des
réarrangements chromosomiques

I. Classification des anomalies K
La classification se fait selon :
1. le lieu et le temps de survenue : on peut avoir soit des anomalies constitutionnelles
(présentes dans toutes les Cl de l’organisme) qui mènent à l’existence d’une « maladie
génétique » qui sera transmissible à la descendance SOIT des anomalies acquises (au cours
des mitoses) appelées anomalies somatiques (pas présentes dans toutes les Cl de l’individu).
2. le mécanisme : cela peut être des anomalies de nombre (trisomie, monosomie), des
anomalies de structure (délétion, duplication, inversion, translocation, isochromosomes, K
en anneaux avec perte des télomères et fusion des K). Les délétions et duplications sont
appelés des CNV ou Copy Number Variant (à retenir).
3. la conséquence : anomalies déséquilibrées (trisomie, monosomie, délétion, duplication), ou
anomalies équilibrées (pas de perte ou de gain du matériel génétique) avec les translocations
ou les inversions.
Ces réarrangements chromosomiques peuvent avoir plusieurs conséquences :
1. Délétion d’un ou plusieurs gènes entraînant une haploinsuffisance
2. Duplication d’un ou plusieurs gènes entraînant souvent une surexpression de celui-ci
3. Délétion causant l’apparition d’un gène de fusion (cancer)
4. Un souci au niveau du point de cassure entraînant pathologie et haploinsuffisance
Des conséquences phénotypiques communes peuvent être retrouvés entre les différentes grandes
anomalies déséquilibrées des autosomes (tous les K sont concernés sauf les K sexuels) => atteinte
cognitive, syndrome malformatif, anomalie de la croissance. Pour les anomalies touchant les
gonosomes (les K sexuels), on peut observer une stérilité, une insuffisance gonadique (exemple dans
le syndrome de Turner) si anomalie de nombre. Par contre, on observera les mêmes conséquences
phénotypiques chez les garçons présentant une anomalie de structure sur ses K sexuels que chez les
personnes présentant une anomalie déséquilibrée des autosomes.
Pour ce qui est des grandes anomalies équilibrées cette fois, le PT sera généralement normal
mais il y aura un risque de déséquilibre chez la descendance.
II. Mécanisme de formation de ces réarrangements génomiques
A. NAHR (non allelic homologuous recombination)
Il s’agit d’une recombinaison entre régions répétées. On a ici des hots spots de réarrangements
principalement au niveau des LCR ou Low Copy Repeat, qui sont des séquences présentant plus de
95% de ressemblance. Ces réarrangements au niveau des LCR vont donner lieu à des anomalies
récurrentes, c’est-à-dire des anomalies retrouvées chez de nombreux patients pour une pathologie
donnée, il y a donc récurrence, ce qui s’oppose à des cas isolés. (Savoir distinguer anomalie récurrente
d’une anomalie non récurrente).
Exemple de pathologies où l’on peut voir ce genre de mécanisme : neurofibromatose de type 1
B. NHEJ (non homologous end joining)
Il s’agit ici d’un mécanisme de réparation de l’ADN lors d’une cassure double brin entre deux
séquences non homologues. Le problème retrouvé lors de cette réparation est une perte de matériel car
il y a fusion des deux extrémités avec modification éventuelle et très souvent une délétion. C’est un
mécanisme de réparation non conservatif car la séquence initiale de l’ADN ne sera pas conservée. On
retrouvera dans le cas de NHEJ des anomalies non récurrentes.
C. FoSTeS (Fork Stalling and Template Switching)

On retrouve ce mécanisme pendant la réplication de l’ADN. Il fait intervenir des sites de
microhomologie. Le mécanisme de FoSTeS est différent de la recombinaison homologue non-
allélique (NAHR) et de la jonction d’extrémités non homologues (NHEJ) parce qu’il s’agit d’une voie
basée sur la réplication qui ne nécessite pas forcément la survenue d’une cassure double brin.
(Mécanisme peu détaillé).
III. Méthodes d’exploration du génome
Pour étudier la séquence de l’ADN on va plutôt parler de génétique moléculaire, tandis que pour
étudier le chromosome, on parlera de génétique chromosomique ou cytogénétique.
Pour explorer le génome, on bénéficie de plusieurs techniques : le caryotype, la FISH, les puces à
ADN ainsi que le séquençage de nouvelle génération.
A. Le caryotype
Son intérêt: pangénomique (tout le génome est observé en une seule fois), observable au
microscope mais PEU précis (résolution 5-10 MB)
Les micro réarrangements ne sont PAS visibles avec un caryotype car ils font moins de 5 MB.
B. La FISH
Le problème de la FISH est qu’il faut que ce soit ciblé. C’est une méthode non pangénomique, il
faut être orienté et savoir « où chercher ».
Son principe : on dispose d’une sonde ADN complémentaire d’une séquence ADN marquée à la
fluorescence, puis dénaturée avec séparation du double brin, puis hybridation des sondes
complémentaires à l’ADN (sondes spécifiques à locus donné).
Son intérêt : BIEN pour des réarrangements récurrents. La résolution est d’environ 200 KB
C. Puces à ADN
Leur intérêt : pangénomique ET bonne résolution (la résolution dépend du nombre de sondes,
souvent autour d’une dizaine de KB).
Leur principe : on procède à une analyse K sur puces à ADN (ACPA). Miniaturisation ++. Le but
sera de comparer les différences entre de l’ADN du patient et de l’ADN témoin.
Il existe deux méthodes: CGH Array et puces à ADN de type SNP.
CGH Array: l’ADN témoin et l’ADN patient sont marqués à l’aide de deux couleurs
différentes. Exemple : l’ADN patient est rouge et l’ADN témoin est vert. On hybride l’ADN
avec des oligonucléotides présents sur la puce.
L’analyse des données consiste à regarder les couleurs pour chaque point de la puce.
*Si c’est normal, on aura un point de couleur jaune car il y a autant d’ADN patient que
d’ADN témoin (deux copies).
*Si on a qu’une seule copie, on obtient un point de couleur verte, car délétion et donc moins
d’ADN patient.
*Si on a plusieurs copies sur l’ADN patient, avec des duplications, la sonde du patient
s’hybridera préférentiellement aux oligonucléotides, on obtiendra un point de couleur rouge.
Puces à ADN de type SNP : On a ici seulement le patient, pas d’ADN témoin. L’ADN est
dénaturé, amplifié, fragmenté, redénaturé puis hybridé sur la puce. Sur la puce:
oligonucléotide qui va s’arrêter pile au moment du polymorphisme avec ajout d’une seule
base à chaque fois. Permet de savoir si l’individu est homozygote ou hétérozygote pour un
allèle donné. C’est un principe différent de la CGH Array car on a dans l’exploitation des
résultats, la présence du B allèle fréquency. Si on a une délétion, on observera une baisse de

l’intensité de ce signal avec perte du signal hétérozygote. Inversement, s’il y a duplication, on
aura une augmentation du signal et apparition de 4 GT possibles. Le défaut des puces à ADNP
de type SNP: ne détecte PAS les anomalies équilibrées (exemple translocation équilibrée,
toutes les anomalies où il n’y a pas de perte de matériel génétique).
IV. Réarrangements génomiques et pathologies humaines
On peut dénombrer deux grands types de microarrangements :
Les arrangements récurrents avec syndromes connus
Les arrangements non récurrents (cas isolés)
A. CNV récurrents
Exemples: Syndromes de Di George, Charcot Marie Tooth, Angelman
Maladie de Charcot Marie Tooth ou neuropathie sensitivomotrice héréditaire :
La duplication de cette région donne une pathologie et la délétion en regard (exactement dans la même
région que la duplication de la maladie de Charcot) donne d’autres symptômes.
Signes cliniques : Faiblesse progressive des Mb inférieurs, scoliose, pieds creux, perte de
sensibilité distale, réflexes diminués ou absents.
Un certain type de cette maladie (parmi cinq grands types trouvés): CMT1A (présentant une
démyélinisation de l’axone des neurones).
Duplication du gène PMP22: transmission autosomique dominante à pénétrance
Incomplète (certains individus peuvent porter la duplication sans être forcément malade). Le
gène PMP22 code pour une protéine de structure de la myéline des nerfs périphériques.
70% des patients ont une duplication de 1,5 MB en 17p112.
Neuropathie héréditaire avec hypersensibilité à la pression: pathologie présentant une délétion
dans la même région que la maladie de Charcot (où on a duplication).
Signes cliniques : épisodes transitoires, fréquents après traumatisme léger ou compression.
Récupération en plusieurs jours/semaines. Déficit moteur résiduel dans 10% des cas avec
vitesse de conduction anormale à l’EMG.
Epaississement de la myéline (contrairement à la maladie de Charcot où on observe une
démyélinisation).
Transmission autosomique dominante à pénétrance incomplète avec délétion de la même
région que celle de la maladie de Charcot.
Dans les deux cas, la délétion/duplication de PMP22 est due à une recombinaison inégale entre
des régions de haute homologie.
Syndrome de Di George (microdel 22q11)
1 enfant sur 4000
de novo dans 90% des cas
Variabilité du PT. Les signes cliniques sont : dysmorphie faciale discrète (alignement avec
extrémité de l’oeil, narine, coin de la lèvre), fente palatine, troubles de l’apprentissage,
troubles psychiatriques, malformations viscérales (au niveau du thymus, des glandes
parathyroïdes, cardiopathie cono troncale).
Situé sur gène TDX1. Plusieurs régions d’homologie, délétions récurrentes (petites ou
grandes)
Syndrome de Smith Magenis (del17p11)
Signes cliniques : dysmorphie, petite taille, doigts et orteils courts, difficultés d’apprentissage,
déficit intellectuel, troubles du comportement, hyperactivité, agressivité, troubles du sommeil.

Délétion vue à la FISH.
Gène RAI1 responsable de la majorité des signes cliniques.
Syndrome de William (7q11)
1 enfant sur 10 000 atteint
dysmorphie en visage d’elfe (car il y a plus d’élastine, la peau est donc plus souple) et
cardiopathie caractéristiques (sténose aortique), retard du développement intellectuel, retard
de langage, hypersensibilité émotive, anxiété, fascination pour les jeux de lumières,
comportement social et amical, hypercalcémie.
Délétion 7q11 vue à la FISH.
Syndromes dûs à des régions soumises à empreinte :
15q11-q13 région soumise à empreinte. Cela signifie que les gènes au niveau de cette région
sont exprimés soit à partir du K du père soit à partir du K de la mère, et pas les deux K des
deux parents à la fois.
Syndrome d’Angelman : avec comme signes cliniques un déficit mental, une microcéphalie,
un aspect joyeux, une ataxie, des crises d’épilepsie. Ce syndrome est lié à une délétion de
gènes du K maternel.
Inversement, le syndrome de Prader Willi, avec comme signes cliniques une hypotonie, un
retard de langage, des difficultés alimentaires dans la petite enfance, un retard d’apprentissage,
un hypogonadisme, une hyperphagie entraînant un surpoids, avec délétion de cette même
région 15q11-q13, mais transmise par le père.
Question : Pourquoi observe-t-on d’avantage de microdélétions que de microduplications ?
Hypothèses
1. Les microduplications ne sont pas viables, létales donc non vues?
2. Les microduplicatins ne sont pas pathologiques, donc on observe pas de PT, donc non vues
non plus?
3. Le PT est très variable, donc pas de caractéristiques communes entre les différents patients,
donc PT plus difficile à reconnaître?
Une réponse possible serait que les conséquences des microduplications sont plus modestes
que les microdélétions. La pénétrance serait souvent incomplète et le Pt d’expression variable.
Les microduplications sont beaucoup plus souvent héritées et moins « de novo » que les
délétions (70% des microduplications de 1 à 10 MB sont héritées). Il faut être prudent
cependant dans l’interprétation des résultats car l’expressivité est certainement variable.
Syndrome de Potocki Lupski (duplication de 17p11.2 comme Syndrome Smith
Magenis dont il est le syndrome réciproque)
Signes cliniques : une hypotonie, un retard de développement, des difficultés alimentaires, une
déficience intellectuelle, un retard de langage
Duplication au niveau de la région 7q11.23, syndrome réciproque de Willliams. Grande variabilité
clinique décrite dans des articles.
Duplication 15q11-q13: le PT est variable, il dépend de l’origine parentale (duplication paternelle
héritée moins sévère que si la duplication vient du côté maternel). On observe tout de même quelques
signes cliniques constants :déficience intellectuelle, troubles autistiques, épilepsie.
Les duplications sont plus difficiles à distinguer dans la clinique, que certaines délétions causant des
syndromes connus avec des signes cliniques caractéristiques. Souvent, dans les duplicatins, les
éléments dysmorphiques ou malformation sont mineurs voire absents.
C’est pour cela que les puces à ADN sont très importantes car pangénomiques.
 6
6
 7
7
 8
8
 9
9
 10
10
1
/
10
100%