Cours3 - Université Nice Sophia Antipolis

Polytech UEF1
Outils moléculaires
pour l’analyse des génomes
BONCOMPAGNI Éric
MCU - Univ. Nice Sophia Antipolis
Site WEB : sites.unice.fr/EB

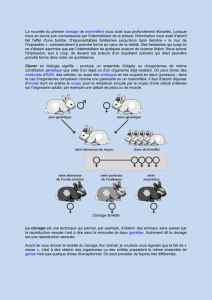
Le clonage d’une séquence
But : Disposer à volonté et en quantité d’une séquence d’ADN d’intérêt
En la multipliant dans un système bactérien (Escherichia coli)
Cloner des séquences d’intérêt
Exemple de vecteur de clonage
Nécessite un vecteur de clonage qui comporte:
1- Une origine de réplication
2- Un gène de sélection
4- Un site multiple de clonage
MCS du pBluescript SK-

Cloner des séquences d’intérêt
1- Digérer l’ADN à cloner par une enzyme de
restriction adéquate
2- Digérer le vecteur par la même enzyme
4- Mettre en présence le vecteur et fragment
à cloner: appariement par
complémentarité des bases
5- Ajouter une DNA ligase (referme la
molécule d’ADN)
6- Transformer une souche d’E. coli
7- Sélectionner les bactéries transformées
et les isoler
Fragment d’ADN obtenu
facilement par extraction d’ADN
plasmidique à partir des bactéries
transformée
Le clonage d’une séquence: Les étapes

Le clonage d’une séquence: la technologie GATEWAY
Issue des connaissances acquises sur le processus d’intégration du phage
Lambda dans le chromosome d’E. coli
Cloner des séquences d’intérêt

Cloner des séquences d’intérêt
Le clonage d’une séquence: la technologie GATEWAY
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
1
/
19
100%