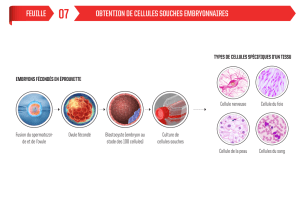
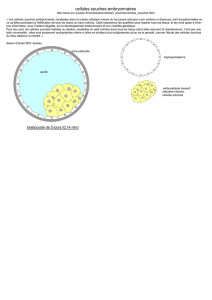
Master Ecosciences, Microbiologie PROPOSITION SUJET de

MasterEcosciences,Microbiologie
ParcoursRecherche«EcologieMicrobienne»
BâtimentDubois–2èmeétage
UniversitéClaudeBernardLyon1
69622–VILLEURBANNECEDEX
Tel:0472431377
E‐mail:master2.ecomi@univ‐lyon1.fr
http://spiral.univ‐lyon1.fr/Files_m/M5298/WEB/EcologieMicrobienne.htm
PROPOSITIONSUJETdeMASTER2014‐2015
TITRE:Diversité spécifique et infra-spécifique du genre Agrobacterium spp. : apport de la
technologie MALDI-TOF à l'analyse des agrobactéries isolées de cas cliniques.
Nom,PrénomduMaitredeStage: AnneDOLEANS‐JORDHEIMetXavierNESME
Qualité:Maître de Conférences-Praticien Hospitalier Attaché
Téléphone: + 33 (0)4 78 77 75 15 (secrétariat : + 33 (0)4 78 78 56 89)
E‐mail: anne.doleans‐jordheim@univ‐lyon1.fr
Laboratoired’accueil,
UMRCNRS5557EcologieMicrobienne–Y.MOENNE‐LOCCOZ
Responsable(s)etéquipe(s):
1‐Equipe"Bactériespathogènesopportunistesetenvironnement"(A.Doléans)
LaboratoiredeMycologie‐Microbiologie(PavillonNétien3èmeétage)
FacultédePharmacie‐ISPB
8,avenueRockefeller,69373LyonCedex08
2‐Equipe"Diversitéetadaptationdesbactériesphytopathogènes"(X.Nesme)
Bat.G.Mendel,16rueR.Dubois
69622VilleurbanneCedex
Nomducandidatéventuellementproposé:
S'iln'estpasretenu,acceptez‐vousunautrecandidat? Oui‐Non
Descriptiondusujetauverso

Sujet(objectif,démarcheettechnique,collaboration(s),...):
Agrobacteriumspp.estungenrebactérientrèsdiversifié,largementretrouvédansl’environnement
telluriqueetlarhizosphèredesplantes.Lesagrobactériessontbienconnuespourleurimplicationdans
lamaladiedelaGalle‐du‐Colletdesvégétauxetpourleurutilisationenbiotechnologiepourconstruire
desplantestransgéniques.Ellessontégalementsignaléesencliniquehumaine,notammententant
qu’espècescolonisatricesvoirepathogènesdespoumonsdespatientsatteintsdemucoviscidose.En
prospectantdenombreuxbiotopes(sol,racines,galleducollet,cascliniques...),nousavonstrouvé19
espèceschezAgrobacteriumspp.Cesespècesontétédéfiniessurdescritèresgénomiqueset
phylogénétiquesetsontde"bonnesespèces"ausenssystématique.Cependant,dufaitdeleurs
découvertesrécentes,nombred'entreellesn'ontpasencoreétéofficiellementvalidéesalorsque
certainesdeces"bonnesespècesnon‐validées"ontétéisoléesdecascliniques.Afind'améliorerle
diagnosticdesinfectionsopportunistesàAgrobacteriumetdanslebutdedévelopperdesétudes
épidémiologiquesperformantes,ilapparaitdèslorsnécessairedeprendreencomptelesconnaissances
lesplusrécentessurladiversitédecetaxon,endevançantl'évolutionattenduedesanomenclature.
Desméthodesdetypagemoléculairebaséessurleséquençagemulti‐locusontétédéveloppées.Elles
permettentdedistinguerlesespècesmaiségalementlessouchesàl'intérieurdesespèces.C'estdoncun
outilépidémiologiquetrèsefficacequirequiertcependantuneétapedeséquençagequiralentitle
diagnostic.LatechnologieMALDI‐TOF,baséesurdesanalysesdeprotéines(essentiellementdes
protéinesribosomiques)parspectrométriedemasseestpoursapartbeaucoupplusrapideetsouple
d'emploi.C'estdecefaitunedestechnologieslesplusutiliséespourl’identificationbactérienneen
cliniquehumaine.Laportéedesonintérêtenépidémiologierestecependantàdémontrercarelle
dépenddesacapacitéàdistinguerdessouches,c'est‐à‐direlesdifférentscomplexesclonauxexistantà
l'intérieurdesespèces,voirededistinguerdesformespathogènesdesformesnon‐pathogènes.
L'hypothèsequenousvoulonstesterestquelatechniqueMALDI‐TOFpermetnonseulementde
distinguerlesespècestrèsapparentéesdugenreAgrobacteriummaiségalementdessouches
notablementdifférentesàl'intérieurdesespèces(i.e.appartenantàdescomplexesclonauxdifférents)
voirelessouchesphytopathogènes(i.e.hébergeantleplasmideTisupportdelavirulence).
Démarcheexpérimentales:
Nousdisposonsd'unecollectionde42souchesappartenantaux19espèces/espècesgénomiques
actuellementconnueschezAgrobacteriumspp..Nousdisposonségalementdelaséquencedesgénomes
decessouches.
Dansunpremiertemps,lessouchesserontanalyséesàl'aided'unappareilMALDI‐TOFVITEKMS
(bioMérieux®)entestantlesconditionsdepré‐culture(milieuxettempératuredecroissance)donnant
lesrésultatslesplusreproductibles.Lesspectresémisservirontàlacréationd’unebasededonnées
exhaustivesurAgrobacteriumspp.quipermettrad'évaluerl'aptitudedelaméthodeàdistinguer:(i)les
espèces;(ii)lescomplexesclonauxdanslesespèces(complexesquiserontmisenévidenceparanalyses
multi‐locusdessouchesàpartirdel'analysedeleursgénomes);(iii)dessouchesquasiisogéniquesavec
etsansplasmideTi.L'étudeseracomplétéeenvérifiantlacapacitédelaméthodeàidentifierdes
souchescliniquesrécemmentisolées.
Dansunsecondtemps,lesdonnéesgénomiquesserontutiliséespouridentifierlesprotéines
ribosomiquescomposantlespectreMALDI‐TOFdesespècesanalyséesafindeproposerunmodèle
permettantdeprédirelespectreMALDI‐TOFattendudenouvellesespèces.
Collaborations:
Equipe«Diversitéetadaptationdesbactériesphytopathogènes»,UMR5557(Resp.X.Nesme)
Equipe«Bactériespathogènesopportunistesetenvironnement»,UMR5557(Resp.B.Cournoyer)
LaboratoiredebactériologieduCentredeBiologieEst,HCL(Resp.F.Vandenesch)
SociétébioMérieux,siteLa‐Balme‐LesGrottes
1
/
2
100%