Master Ecosciences, Microbiologie PROPOSITION SUJET de

MasterEcosciences,Microbiologie
ParcoursRecherche«EcologieMicrobienne»
BâtimentDubois–2èmeétage
UniversitéClaudeBernardLyon1
69622–VILLEURBANNECEDEX
Tel:0472431377
E‐mail:master2.ecomi@univ‐lyon1.fr
http://spiral.univ‐lyon1.fr/Files_m/M5298/WEB/EcologieMicrobienne.htm
PROPOSITIONSUJETdeMASTER2014‐2015
TITRE:Recombinaison et conversion génique biaisée: une nouvelle force évolutive à l'œuvre
dans les génomes bactériens?
Nom,PrénomduMaitredeStage:BERTOLLAFranck(co‐encadrementL.DURET,LBBE‐UMR5558)
Qualité: MaîtredeConférences
Téléphone:0472438289 E‐mail:bertolla@univ‐lyon1.fr
Laurent.Duret@univ‐lyon1.fr
Laboratoired’accueil,Responsableetéquipe:
UMRCNRS5557EcologieMicrobienne–Y.MOENNE‐LOCCOZ
EquipeDiversitéetadaptationdesbactériesphytopathogènes
Adresse: Bat.G.Mendel,16rueR.Dubois
69622VilleurbanneCedex
Nomducandidatéventuellementproposé:
S'iln'estpasretenu,acceptez‐vousunautrecandidat? Oui‐Non
Descriptiondusujetauverso

Sujet (objectif, démarche et technique, collaboration(s),...) :
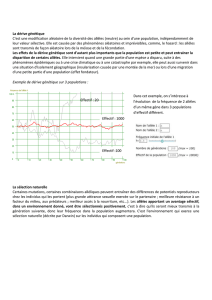
Classiquement, trois processus sont pris en considération pour interpréter l'évolution des séquences dans
les génomes: la mutation, la sélection et la dérive. Cependant, depuis quelques années, nos travaux (LD)
ont permis de montrer qu'un quatrième processus jouait un rôle majeur dans l'évolution des génomes: la
conversion génique biaisée (gBGC). Chez les eucaryotes, ce processus est intrinsèquement lié à la
recombinaison méiotique: la présence de sites polymorphes au sein des hétéroduplex formés entre les
deux chromosomes parentaux conduit à l'apparition de mésappariements, dont la réparation aboutit à la
conversion d'un des deux allèles par l'autre. Il a été montré que chez la levure, les mésappariements
AT|GC sont préférentiellement réparés en paire de base G:C plutôt que A:T [1,2]. De ce fait, les allèles
G:C ont une probabilité plus forte d'être transmis aux gamètes que les allèles A:T. Ce processus augmente
donc la probabilité de fixation des allèles G:C, et peut même contrer les effets de la sélection et conduire à
la fixation de mutations délétères [3].
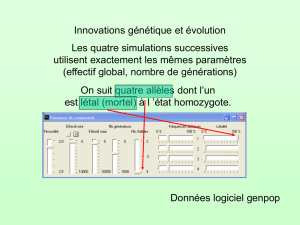
Chez les organismes asexués, contrairement à la plupart des eucaryotes sexués, la reproduction
n'implique pas de recombinaison obligatoire. De ce fait, jusqu'à présent, le gBGC n'était pas envisagé
comme un processus potentiellement important chez les bactéries et les archées. Récemment deux
équipes ont démontré que chez les bactéries, les allèles G:C tendent à se fixer dans les populations avec
une probabilité plus grande que les allèles A:T [5,6]. Cette observation a été interprétée comme résultant
d'une pression de sélection en faveur d'un taux de G+C génomique plus élevé [6]. Cependant la raison
pour laquelle le taux de G+C d'un génome pourrait affecter la fitness d'un organisme reste inconnue [6].
L'objectif de ce sujet de recherche est de tester directement l'existence du gBGC chez des bactéries.
Démarche expérimentale :
Des expériences de transformation seront menées pour intégrer de l'ADN exogène dans un génome
bactérien par recombinaison homologue à un locus cible. Cette partie implique la construction des
matrices d'ADN exogènes présentant des mésappariemments avec divers teneurs G+C et les expériences
de transformation. Les produits de recombinaison feront ensuite l'objet de séquençage massif afin de
quantifier la transmission des allèles dans les zones de conversion génique par des outils d'analyse
biostatistique.
Le stage M2 sera menée chez la bactérie Acinetobacter baylyi, chez qui la recombinaison est très efficace (de
l'ordre de 1% de clones recombinants) permettant ainsi d'obtenir facilement le nombre de recombinants
nécessaire pour les analyses. Par la suite, cette expérience pourra être répétée chez différentes espèces de
bactéries et d'archées pour ainsi tester si le gBGC est – ou non – un processus universel et pour explorer
quels sont les facteurs qui influent sur l'intensité du gBGC chez différents organismes.
Collaborations :
Ce sujet sera mené en collaboration entre deux équipes, l'une du laboratoire de Biométrie et Biologie
Evolutive, l'autre du laboratoire d'Ecologie Microbienne. Laurent Duret est spécialiste de génomique
évolutive, et est notamment pionnier dans l'étude du gBGC. Ses travaux sont principalement basés sur
des approches bioinformatiques et statistiques d'analyse de séquences. Franck Bertolla est spécialiste de la
biodiversité bactérienne et s'intéresse en particulier au processus de transformation, qui joue un rôle
majeur dans l'évolution des répertoires géniques chez les bactéries.
Références :
[1] Mancera et al. (2008). Nature, 454:479–85.
[2] Lesecque et al. (2013). Mol. Biol. Evol. 30:1409–19.
[3] Duret &Galtier (2009). Ann. Rev. Genom. Hum. Genet. 10:285–311.
[4] Pessia et al. (2012). Genome Biol. Evol, 4:675–82.
[5] Hershberg & Petrov (2010) PLoS Genet. 6:e1001115.
[6] Hildebrand et al. (2010) PLoS Genet. 6:e1001107
1
/
2
100%