d`Escherichia cou - DSpace à Université abou Bekr Belkaid Tlemcen

République Algérienne Démocratique et Populaire
-
Ministère de l'Enseignement Supérieur et de la Recherche Scientifique
Université Abou Bekr Belkaid -Tlemcen
Faculté des Sciences de la Nature et de la Vie et
-
Sciences de la Terre et de l'Univers
Département de Biologie
Laboratoire
-
Antifongiques, Antibiotiques: Physico-chimie, Synthèse et activité biologique
Ï
<
L
,
3 p S c3b
-
Mémoire en vue de l'obtention du Diplôme de Master Académique
En Biologie
Option Contrôle du développement microbien
-
Intitulé
-
Profils de résistance aux quinolones de souches communautaires
L
d'Escherichia cou
Présèntée par:
.
BENLAZAR Nabila
Soutenu devant le jury:
•• Mme Bouchrit Z.
Professeur
Présidente
+
Mme Hassaine H.
Professeur
Examinatrice
+
Mme Kazi Tani Z.
Maître Assistant A
Promotrice
Année Universitaire: 2013-2014

Remerciements
Je tiens à remercier plus particulièrement
Mrne
KAZI TANI
Z,
pour avoir accepté
de diriger ce mémoire, pour ses encouragement, sa disponibilité et sa patience, je la remercie
d'avoir conseillé dans la réalisation de ce projet et du soutien qu'elle ma toujours manifeste.
Je tiens à remercier profondément
Mme
BOUCHERIT Z, qui m'a fait l'honneur de
présider le jury.
Mes remerciements les plus sincères à
Mme
HASSAINE H, pour avoir accepté
d'examiner ce mémoire, pour toute son aide, sa gentillesse et ses conseils. Je saisis cette
occasion pour vous exprimer mes sentiments de respect et de gratitude.
Je remercie chaleureusement tous les membres du laboratoire de recherche « Antibiotiques,
Antifongiques, Physico-chimiques, Synthèse et Activité biologique ».
Enfin, je tiens à exprimer mes remerciements à toutes les personnes qui m'ont permis de
réaliser ce travail.

DEDICACE S
Je dédie ce modeste travail
Mes très chers parents,
Pour leur amour, leurs encouragements et leurs sacrifices
Mes frères et ma soeur et toute ma famille.
Mes amies Samia, Fouzia, Asma, Ikram et Ammaria.

Liste des abréviations
°C:
degrée celcus.
gramme
ADH : Arginine DiHydrolase.
ADN: Acide désoxyribonucléique.
CA-SFM : comité d'antibiogramme-société française de microbiologie.
CMI : Concentration Minimale Inhibitrice
DO: Densité Optique
ONPG
: Orthonitrophényl-B-D-galactopyrannoside.
UFC: Unité Format Colonie
VP : Voges Proskauer.

Liste des figures
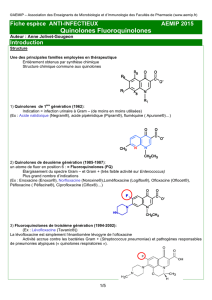
Figure 1.
Structure de base des quinolones
.
Figure 2. Mécanisme de diffusion intra-bactérienne des quinolones...............................4
Figure 3. Mécanisme de bactéricidie des quinolones.................................................5
Figure 4. Schématisation
des différents mécanismes de résistance aux quinolones .............. ô
Figure
5.Identification par galerie API 20E .........................................................11
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
1
/
37
100%