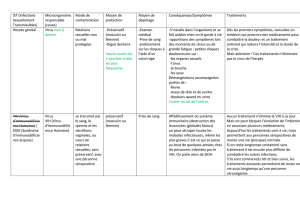
Les bases de la résistance du VIH aux antirétroviraux

Les bases de la r
é
sistance du VIH aux
antir
é
troviraux
Dr Bernard
Masquelier
Laboratoire
de
Virologie
, CHU de Bordeaux
et EA2968
Universit
é
Victor
Segalen
Bordeaux 2

RT
Provirus
Proteins
RNA
RNA
RT
RNA
RNA
DNA
DNA
DNA
Cibles
des
antir
é
troviraux
Inhibiteurs
d
’
entr
é
e
Inhibiteurs
de
la TI
Inhibiteurs
de
Prot
é
ase
Inhibiteurs
du
bourgeonnement
Inhibiteurs
de
l
’
int
é
grase

Taux
d
’
erreur
de la TI : 1/10.000
nucl
é
otides
Production
virale
: 10
9
-
10
10
particules
par jour
Taux
de
recombinaison
: 5
à
10
é
v
è
nements
par cycle
Evolution
constante
de la
quasiesp
è
ce
Toutes
les mutations
pr
é
-
existent
avant
traitement
Demie
-
vie d
’
un virus
plasmatique
= 0.3
jours
Demie
-
vie des cellules
infect
é
es
= 2.2
jours

Definition de la R
é
sistance aux
Antiviraux
•
La r
é
sistance
est
due
à
la
pr
é
sence
de mutations
sur
le
g
é
nome
viral qui
r
é
duisent
la
sensibilit
é
du virus par
rapport
à
celle
observ
é
e
chez un virus
sauvage
–
Modifications au
niveau
de la
cible
mol
é
culaire
de
l
’
ARV
(Ex : RT,
prot
é
ase
, Int
é
grase)
–
Modifications au
niveau
d
’
autres
prot
é
ines
virales
qui
interf
è
rent
indirectement
avec
l
’
activit
é
de la
mol
é
cule
antir
é
trovirale
(ex :
g
è
ne
gag/ R
é
sistance aux IP)

Pression
de
s
é
lection
antir
é
trovirale
S
é
lection
de variants
r
é
sistants
Suppression
incompl
è
te
de la
r
é
plication
virale
•
D
é
faut
de puissance
•
Taux
plasmatiques
insuffisants
•
D
é
faut
d
’
observance
•
Pr
é
-
existence de r
é
sistance
Charge
virale
Temps
Variants
sensibles
Variants
r
é
sistants
D
é
but du
traitement
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
1
/
36
100%