Code génétique

Alain Bruyère
Paris 12-13/05/2006 1
exemple avec la protéine
hémoglobine
• les globules rouges acheminent l’oxygène depuis
les poumons jusqu’aux tissus qui en ont besoin
•les globules rouges ne possèdent pas de noyau et
donc ont tout le loisir de se remplir
d’hémoglobine, molécule/protéine responsible du
prélèvement et du dépôt de l’oxygène
•les globules rouges sont fabriqués dans la moelle
osseuse par des cellules
•En résumé :
–globule rouge
• contient l’hémoglobine
– qui contient l’oxygène

Alain Bruyère
Paris 12-13/05/2006 2
gène
• lorsque le besoin de produire d’avantage
d’hémoglobine se fait sentir, le segment d’ADN
correspondant de la moelle osseuse, le gène de
l’hémoglobine, s’ouvre en deux (comme lors de la
réplication de l’ADN) mais cette fois un seul des
deux brins est copié ou transcrit
• gène : sorte de programme situé dans l’ADN =>
unité d’information permettant de coder, c’est
donc une partie, un segment porteur de sens de
l’ADN, destiné à être transcrit en ARN
•chez les eucaryotes, l’ADN est généralement sous
forme de plusieurs chromosomes linéaires

Alain Bruyère
Paris 12-13/05/2006 3
chromosome
•les chromosomes portent les gènes
•un chromosome est constitué
•d’une molécule d’ADN
•et de protéines
•l’espèce humaine conte 46
•23 paires
•dont 22 sont des
chromosomes
homologues
•la dernière paire
correspond aux 2
chromosomes sexuels X et Y
•le génome humain est réparti sur ces 24 chromosomes;
les gènes ne constituent qu’une partie du génome
la plus grande partie du
génome est contenue dans
le noyau
le cytoplasme contient
également une petite
partie du génome
cytoplasme

Alain Bruyère
Paris 12-13/05/2006 4
ARNr - ARNt
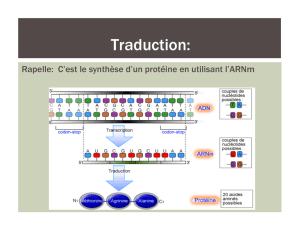
• la molécule d’ARN ainsi produite peut
ensuite
– soit être traduite en protéine (on l’appelle dans
ce cas, ARNmessager)
–soit être directement fonctionnelle
•ARN ribosomique ou ARNr ou
•ARN de transfert ou ARNt

Alain Bruyère
Paris 12-13/05/2006 5
ARNp-ARNm-ribosome
• grâce à l’enzyme l’ARN polymérase, ARNp,
plutôt qu’un nouveau brin d’ADN, c’est un brin,
d’ARNmessager ou ARNm qui est créé et qui
correspond donc au gène de l’hémoglobine
• l’ADN peut alors se refermer
• l’ARNm est transporté en dehors du noyau vers un
ribosome (se trouve dans le cytoplasme, matériel cellulaire contenu par la
membrane, excepté le noyau)
–molécule ribonucléoprotéique contenant
• des fragments d’ARNr, doté de propriétés catalytiques
•des protéines ribosomiques
•l'existence de l'ARNm a été demontrée par Jacques Monod et ses
collaborateurs, ce qui lui valut le prix Nobel de Médecine en 1965
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
1
/
27
100%