09 Polymorphisme

UE Génétique médicale Typeur : LEGRAND Renaud– ARTUS Arnaud
Cours n° 9 – Pr Rochette – 29.01.2014 Correcteur : JAAFAR Ali
1
LE POLYMORPHISME
I. Généralités
A. Définition
Polymorphisme plusieurs formes
Toute variation de séquence de l’ADN, qu’elle soit ponctuelle ou qu’elle intéresse une répétition de
séquences (mini-microsatellites), est un polymorphisme si sa séquence a une fréquence ≥ 1% dans
une population donnée.
L’ADN n’est pas un élément statique :
- Il est sujet à des modifications (variations transmissibles) intra ou interindividuelles.
- Les variations de séquences sont appelées polymorphisme lorsqu’elles surviennent à une
fréquence > 0,01.
- Si une séquence a une fréquence < 1% : on parle de mutation (privée).
- En fait, les polymorphismes sont arrivés par mutation !
Les polymorphismes sont soit : neutres, avantageux ou désavantageux.
L’hétérozygotie moyenne de l’ADN humain est environ 0,004 : 1 base différente pour 250 à 300
bases entre 2 allèles ou 2 séquences entre 2 individus (vient des recombinaisons).
Le polymorphisme affecte toutes les régions de l’ADN :
- séquences codantes
- introns
- ADN répété (mini – microsatellites)
Les polymorphismes de l’ADN servent à son analyse : La séquence d’un locus peut varier d’un
individu à un autre.
Le polymorphisme (de restriction ou de répétition) n’est pas nécessairement la cause de la maladie.
- Il peut s’agir d’un polymorphisme marqueur d’une maladie sans qu’il en soit la cause
- Le fait qu’un polymorphisme soit près d’un gène muté (par exemple) est utilisé comme
marqueur de la maladie.
Domaine du diagnostic indirect
Le polymorphisme peut modifier :
- Un site de restriction Polymorphisme de restriction
- La longueur d’un motif répété Polymorphisme de répétition

2
B. Types de polymorphismes
Donc deux types de polymorphismes :
- Polymorphisme SNP / de restriction
o Single Nucleotide Polymorphism
o polymorphisme de taille (de longueur)
o Sur un profil analysé par la méthode de Southern
o Peut induire une différence de longueur détectable par une enzyme de restriction
o Ce sont des outils qui vont servir au diagnostic des maladies, et à reconnaître les
individus
- Polymorphisme de répétition : variation de taille
o Microsatellite (2 ou 3 nucléotides répétés, ex : CACACACACACA…)
o Minisatellite (15 nucléotides répétés)
C. Nomenclature :
La séquence d’un locus peut varier d’un individu à l’autre.
- RFPL : Restriction Fragment Lenght Polymorphism
o Variation ponctuelle de séquence affectant un site de restriction
o utile pour faire les diagnostiques de maladies (détection de présence ou absence
d’un acide a un endroit donné)
o mis en évidence par : Méthode de Southern, PCR
- VNTR : Variable Number Tandem Repeat
o Répétition en tandem pour des séquences assez longues
o = Minisatellite (nombre de copies différentes d’une séquence répétée > 10 pb X n)
o polymorphisme de répétition
- STR : Simple Tandem Repeat
o Microsatellite (1 à 5 pb X n)
Exemple : pilule et mutation homozygote pour certains gènes : du risque de thrombose.
II. RFLP
A. Définition
Un RFLP est défini:
- par un couple Sonde / Enzyme (Southern)
- ou par un couple taille / enzyme (PCR).
Ils sont mis en évidence par la méthode de Southern (Sonde / Enzyme) ou PCR (taille / enzyme).
Sa localisation précise, sa variabilité (présence ou absence du site de restriction) et sa transmission
mendélienne lui confèrent un caractère de marqueur génétique codominant (un chromosome peut
être porteur du site de restriction et pas l’autre)
Le couple sonde/enzyme :
- se caractérise par : +/- (coupe : + / ne coupe pas : -)
- correspond à des allèles (Bi-allélisme).

3
Pour être informatif, le RFLP (=polymorphisme de taille) doit être reconnu par une sonde unique.
Le nombre de sites de restriction pour une enzyme donnée reconnaissant un motif
hexa-nucléotidique: 46 = 4096 séquences possibles.
- Mais seules 64 combinaisons ont la symétrie palindromique réclamée par les enzymes de
restriction (Une séquence palindromique se lie de la même façon de 5’ vers 3’ et de
3’ vers 5’ - ex : RADAR).
- et 41 combinaisons sont reconnues seulement par les enzymes déjà caractérisées.
Donc 1% seulement des polymorphismes par mutation ponctuelle sont détectés par les enzymes de
restriction et constituent des RFLP.
B. Exemple:
L’enzyme EcoR1 reconnaît puis hydrolyse :
5’ GAATTC 3’
3’ CTTAAG 5’
Si cette séquence est mutée, l’enzyme EcoR1 ne peut plus hydrolyser l’ADN à cet endroit.
Si par exemple l’enzyme doit couper 3 sites, elle libèrera 2 morceaux A et B.
Si le site EcoR1 est muté, l’enzyme ne coupera pas il y aura alors 1 morceau AB non coupé.
Dans une maladie, où il y a une mutation qui « annule » un site de restriction : cette méthode permet
d’inventer une méthode de diagnostic.
Mutation

4
C. Sites de restriction
On distingue :
- des sites de restriction obligatoires (toujours
présents chez tous le monde, ex : MST2 coupe
en 1, 2, 3, 4, 5)
- des sites de restriction variables : Chez un
individu, sur 100, il y a un site de restriction en
plus = Polymorphisme
Ce polymorphisme peut être la cause d’une
maladie : on peut faire le diagnostic de la maladie.
N = n’importe quel nucléotide
D. Application indirecte pour faire un diagnostic : marqueur (à savoir +++)
Le site de restriction peut être utilisé comme un marqueur
La mutation sur le chromosome est du au hasard dans une région où il y a un site de restriction aboli.
Explication :
- Pour cette mutation on en déduira que : si le
morceau d’ADN est de 5kb au lieu de 4kb
il y a alors une abolition du site de restriction.
- C’est un diagnostic indirect, qui se fait de moins
en moins aujourd’hui au profit du séquençage.
Lorsqu’il y a une mutation dans le gène qui génère une
maladie génétique, on s’aperçoit que l’enzyme EcoR1 n’a
pas de site Le morceau sera plus grand.
Ceci est valable dans une famille donnée : une
particularité génétique d’une famille ne se retrouve pas
forcément chez une autre.
Plus un polymorphisme est loin du gène muté, moins c’est
utilisable, notamment à cause des recombinaisons.
PS : Plus un polymorphisme est loin du gène muté, moins c’est utilisable, notamment à cause des
recombinaisons.

5
III. Les polymorphismes de variation de longueur
A. Les minisatellites Hypervariables (VNTR)
- Ce sont des séquences répétitives, dispersées et très polymorphes.
- Il s’ajoute à cette variabilité un taux élevé de mutation.
- Ce sont donc des marqueurs polyalléliques sur tous les chromosomes sauf X et Y.
- La séquence est de type : GGAGGTGGGCAGGA(A/G)G, et varie de 11 à 16pb, répétée et en
tandem.
- Ils permettent d’étudier la ségrégation de nombreux loci.
- La probabilité de rencontrer deux individus non apparentés avec un profil identique de VNTR
est < 10-20
Deux individus n’auront jamais les mêmes séquences répétées excepté les jumeaux
homozygotes : c’est la signature génétique (intérêt en médecine légale).
B. Les minisatellites
- 20-100 pb répétées en tandem (c’est la séquence de 16 qui répétée plusieurs fois)
o L’inverse de tandem = dispersé : plusieurs nucléotides entre les répétitions
o tandem = répétitions à la suite
- Polymorphisme de variation de longueur.
- Longueur des répétitions peut varier d’un individu à l’autre (test de paternité et maternité,
un individu aura la moitié similaire à sa mère et l’autre à son père).
C. Exemple
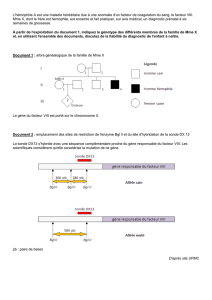
A et B sont deux marqueurs polymorphes encadrant le gène d’une maladie liée à l’X.
Hypothèse :
- le gène, dont on veut suivre la ségrégation, est « lié » à deux marqueurs.
- Si on sait repérer A et B, on en déduit la transmission du gène puisqu’à chaque fois qu’on
hérite du gène, on hérite des marqueurs dans une famille donnée.
Le polymorphisme de restriction ou de répétition n’est pas nécessairement la cause de la
maladie.
Il peut s’agir d’un polymorphisme marqueur dune maladie sans qu’il en soit la cause.
Le fait qu’un polymorphisme soit près d’un gène muté par exemple est utilisé comme marqueur
de la maladie.
Difficile dans la génétique médicale sans étudier la famille (pour les maladies rares +++)
 6
6
 7
7
1
/
7
100%