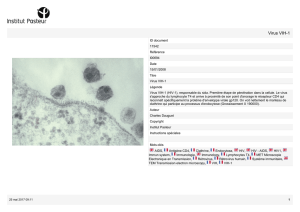
thèse - Université François Rabelais

UNIVERSITÉ FRANÇOIS - RABELAIS
DE TOURS
ÉCOLE DOCTORALE «Santé, Sciences, Technologies»
INSERM U966 : « Morphogénèse et antigénicicté du VIH et des virus des hépatites »
THÈSE
présentée par :
Elodie BEAUMONT
soutenue le : 15 décembre 2009
pour obtenir le grade de : Docteur de l’université François - Rabelais
Discipline/ Spécialité : Sciences de la vie / virologie
Etude de l’émergence in vivo d’un variant du virus de l’immunodéficience
humaine de type 1 portant des glycoprotéines d’enveloppe naturellement
tronquées dans leur domaine cytoplasmique
THÈSE dirigée par :
Mr Denys BRAND Professeur des Universités, Université François Rabelais -Tours
RAPPORTEURS :
Mme Delphine MURIAUX Chargée de recherche, INSERM - Lyon
Mr Serge BENICHOU Directeur de recherche, INSERM - Paris
JURY :
Mr Yves GAUDIN Directeur de recherche, CNRS - Gif sur Yvette
Mr Fabrizio MAMMANO Directeur de recherche, INSERM - Paris
Mme Delphine MURIAUX Chargée de recherche, INSERM - Lyon
Mr Serge BENICHOU Directeur de recherche, INSERM – Paris
Mr Denys BRAND Professeur des Universités, Université François Rabelais -Tours

REMERCIEMENTS

Comme il se doit, je voudrais remercier l’ensemble des personnes ayant contribué de
près ou de loin à ce travail et toutes les personnes qui m’ont soutenue pendant toutes
ces années.
Tout d’abord, je voudrais exprimer ma plus profonde reconnaissance à mon
directeur de thèse, le Professeur Denys Brand, pour la confiance qu’il m’a accordée
durant toutes ces années. Un grand merci pour votre sympathie, votre écoute, votre
sens de l’humour, votre savoir-faire et votre disponibilité de tous les instants. Je vous
remercie pour l’attention que vous avez porté à la direction de ce travail.
Un grand merci également au Docteur Fabrizio Mammano pour la collaboration
fructueuse que nous avons établie et pour l’intérêt porté à mon travail. Merci pour
ton soutien tout au long de ce projet et ta grande disponibilité. Tes précieux conseils
et ta participation dans la rédaction de ma publication ont largement contribué à
ma réussite. Je tiens aussi à te remercier pour avoir si gentiment accepté d’être
examinateur de mon travail.
Par la même occasion, je tiens aussi à remercier Daniela Vendrame pour sa
participation dans la réalisation de certaines manips de cette étude. Merci pour ton
savoir-faire et pour la qualité de ton travail.
Un grand merci au Professeur Bernard Verrier et au Docteur François Biron qui
nous ont mis a disposition tous le matériel sanguin nécessaire à la réalisation de
cette étude, et pour nous avoir donné tous les renseignements cliniques et biologiques
concernant ce fameux patient.
J’adresse mes plus sincères remerciements au Docteur Delphine Muriaux, chargée de
recherche INSERM à Lyon, et au Docteur Serge Benichou, directeur de recherche
INSERM à Paris, pour m’avoir fait l’honneur d’accepter d’évaluer ce travail de
thèse en participant à mon jury en tant que rapporteurs.
Je tiens à remercier chaleureusement le Docteur Yves Gaudin, directeur de
recherche CNRS à Gif sur Yvette, de me faire l’honneur de juger ce travail et de
présider le jury de cette thèse.

Je tiens également à remercier l’ensemble des membres de l’ U966 pour tous les bons
moments que j’ai partagé en votre compagnie au cours de ces années de thèse :
A Philippe, notre responsable du laboratoire, pour m’avoir permis de réaliser ce
travail de thèse au sein de ton équipe, pour ta disponibilité et ton attention de
tous les instants. Un grand merci pour la confiance que tu m’accordes.
A Francis, Alain G. et Jean-christophe, pour leur soutien tout au long de cette
thèse et pour leurs précieux conseils.
A Manu B. et Christophe, le duo de choc, pour votre disponibilité, votre aide au
quotidien et votre sympathie.
A ma petite Manue R., ma voisine de paillasse, pour ton aide technique tout au
long de ce projet. Merci mille fois pour ta disponibilité, ta bonne humeur
quotidienne et ton écoute de tous les instants ! Ce fut un vrai plaisir que de
travailler à tes cotés !
A Eric, mon sauveur, pour ta disponibilité et tes précieux conseils
informatiques qui m’ont été d’une grande aide dans la rédaction de mon article
et de ce manuscrit
A Alain, mon voisin de paillasse, pour ton aide technique et ton savoir-faire en
séquençage. Nos petits moments de rire m’ont beaucoup manquée ces derniers
mois, j’ai vraiment hâte de reprendre du service! (Attention n’oublies jamais
qu’il n’y a que le mercredi que tout est permis !)
A Martine, pour ton aide tout au long de cette thèse et pour ton oreille
attentive. Merci pour tes relectures.
A Nadine, pour ta joie de vivre, pour ton attention et ton écoute de tous les
instants. Ma championne de l’orthographe, merci pour tes relectures.
Au clan des filles (Laura, Marion, Pauline, Suzie, Anne, Manue R. et Virginie
(Les filles en force !)) pour votre soutien au quotidien et votre joie de vivre.
Merci pour toutes les petites soirées organisées par vos soins qui m’ont permis
de faire des "breaks" bien sympathiques au cours de ces derniers mois très
intenses ! J’en avais bien besoin !
A Vincent, le roi du QPUC, pour ton dynamisme et ta sympathie.
A Catherine, pour ta sympathie et tes précieux conseils. Merci aussi pour tous
ces bons moments passés à tes côtés lors de nos escapades parisiennes (En
souvenir de notre petit moment détente enneigé à la tour Eiffel ! Trop bien !)
A christine, pour tes précieux conseils, ta gentillesse et ta bonne cuisine.
A Tanawan et Woottichai, mes deux compagnons thaïlandais du soir, pour
votre joie de vivre et votre sympathie.
A Marie et Romu, pour votre savoir-faire et vos bons conseils
Sans oublier Valentina, Christiane et Loïc

Un grand merci à toute l’équipe du Laboratoire de Biologie Cellulaire et de
Microscopie Electronique pour son accueil chaleureux des derniers mois et surtout à
Pierre-Yves pour sa formation en microscopie confocale.
Je tiens également à remercier l’ensemble du personnel du laboratoire hospitalier de
virologie pour leur accueil chaleureux, et plus particulièrement à Sylvie Brunet pour
son aide technique et ses précieux conseils.
Merci bien évidemment au Ministère de l’Education Nationale, de la Recherche et de
la Technologie ainsi qu’au Sidaction pour leurs financements qui m’ont permis de
réaliser cette thèse.
Un grand merci aussi à tous mes amis qui m’ont soutenue tout au long de cette thèse
et qui m’ont surtout permis tous les week-ends d’oublier tous les soucis de la semaine
et de m’évader un peu, le temps d’une soirée. Merci à Sophie, Valou, Matthieu, Juju,
Aurélien, Sandrine, Jesse, Nini, Alex, Lolo, Mao, Tony, Pat, Domi et Charlène pour
tous ces bons moments de rire et détente passés en votre compagnie, merci à Sylvie
pour toutes ces "IBIZA nights" endiablées, pour ton sens de l’humour et ta joie de
vivre (Ne changes rien !), et enfin merci à mon amie fidèle, Souheila, pour ton
soutien et ton écoute de tous les instants.
Je dédie ce manuscrit à ma famille, et tout particulièrement à ma petite Maman et
ma grande sœur, Lauréna, qui ont toujours cru en moi. Merci pour votre soutien
permanent, votre confiance et votre écoute. Vous avez toujours trouvé les bons mots
pour me réconforter dans les moments difficiles et pour me relancer dans cette quête
de réussite. Merci aussi à mon petit rayon de soleil, Dylan, qui a toujours été très
attentionné envers sa tante (fleurs, chansonnettes, petits dessins…) et qui s’est
toujours chargé de me divertir pendant ces années de thèse.
Merci aussi à Pat, mon père, et à tous mes oncle et tantes, Michel, Jeannine, Ginette,
Jacques, Monique, Pierrot et mon parrain Henri, qui m’ont toujours soutenue dans
cette poursuite d’étude et qui ont toujours été à mon écoute.
Enfin, j’aimerais prendre le temps de remercier mon petit homme qui a toujours été
présent quelque-soient les épreuves traversées. Merci pour ton soutien dans les
périodes sombres, tes encouragements permanent et ta grande patience. Merci de
m’avoir soutenue, supportée et attendue tout ce temps.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
 59
59
 60
60
 61
61
 62
62
 63
63
 64
64
 65
65
 66
66
 67
67
 68
68
 69
69
 70
70
 71
71
 72
72
 73
73
 74
74
 75
75
 76
76
 77
77
 78
78
 79
79
 80
80
 81
81
 82
82
 83
83
 84
84
 85
85
 86
86
 87
87
 88
88
 89
89
 90
90
 91
91
 92
92
 93
93
 94
94
 95
95
 96
96
 97
97
 98
98
 99
99
 100
100
 101
101
 102
102
 103
103
 104
104
 105
105
 106
106
 107
107
 108
108
 109
109
 110
110
 111
111
 112
112
 113
113
 114
114
 115
115
 116
116
 117
117
 118
118
 119
119
 120
120
 121
121
 122
122
 123
123
 124
124
 125
125
 126
126
 127
127
 128
128
 129
129
 130
130
 131
131
 132
132
 133
133
 134
134
 135
135
 136
136
 137
137
 138
138
 139
139
 140
140
 141
141
 142
142
 143
143
 144
144
 145
145
 146
146
 147
147
 148
148
 149
149
 150
150
 151
151
 152
152
 153
153
 154
154
 155
155
 156
156
 157
157
 158
158
 159
159
 160
160
 161
161
 162
162
 163
163
 164
164
 165
165
 166
166
 167
167
 168
168
 169
169
 170
170
 171
171
 172
172
 173
173
 174
174
 175
175
 176
176
 177
177
1
/
177
100%