partie i. les systèmes génétiques bactériens et viraux

Université Paris 13. UFR SMBH. L2. Sciences du Vivant.
UE S3F1. Génétique 2. Partie I.
PARTIE I. LES SYSTÈMES GÉNÉTIQUES BACTÉRIENS ET VIRAUX
COURS
V. LES VIRUS SONT DES SYSTÈMES GÉNÉTIQUES ACCESSIBLES À L'ANALYSE GÉNÉTIQUE
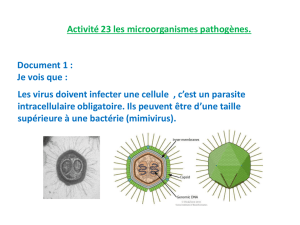
Les virus, entités acellulaires, sont des agents infectieux, des parasites intracellulaires qui ont besoin d'une cellule pour se multiplier.
Les virus sont des éléments génétiques qui se répliquent indépendamment du ou des chromosome(s) de la cellule mais non
indépendamment de la cellule elle-même. Cependant, à la différence des éléments génétiques comme les plasmides, les virus ont
une forme extracellulaire qui leur permet d'exister à l'extérieur de l'hôte pour de longues périodes et qui facilite la transmission d'un
hôte à un autre. Pour se multiplier, les virus doivent pénétrer dans une cellule dans laquelle ils peuvent se répliquer ; ce processus est
appelé infection. Les virus, comme les plasmides et d'autres éléments génétiques, exploitent la machinerie métabolique de la cellule.
Comme ces autres éléments, les virus peuvent conférer à leur hôte d'importantes et nouvelles propriétés. Ces changements sont
généralement inoffensifs et peuvent même être bénéfiques. Les virus peuvent aussi se répliquer d'une manière destructive pour la
cellule-hôte ; c'est la raison pour laquelle certains virus sont des agents pathogènes.
De façon résumée, les virus présentent les caractéristiques suivantes :
- ils ne possèdent qu’un seul type d’acide nucléique (ADN ou ARN), alors que les cellules contiennent les deux.
- ils n’ont pas de machinerie propre pour la synthèse des protéines ; ils n’ont pas de système de conversion d’énergie qui leur soit
propre.
- ils ne sont pas sensibles aux antibiotiques. Par contre, leurs cellules hôtes peuvent l’être.
- ils ne peuvent pas se mouvoir autrement que par diffusion et ne contiennent pas de cytosquelette.
- ils ne croissent pas dans le sens classique du terme (par augmentation de taille et de masse) ; c’est-à-dire qu'une fois le virus formé,
il ne grandit plus.
A. PROPRIÉTÉS GÉNÉRALES DES VIRUS
Au cours de l'émergence de la virologie comme discipline autonome, la classification s'est fondée initialement sur leur pouvoir
pathogène, leur tropisme pour certains organes et leurs caractéristiques épidémiologiques. Puis, au milieu des années 60, a été
construite une classification fondée sur les propriétés physico-chimiques et moléculaires des virus, qui a été associée à une
nomenclature spécifique. Le résultat en a été la taxonomie virale actuelle. Elle permet de considérer de façon unique un même virus,
que l'approche soit clinique, épidémiologique ou moléculaire.
Les virus peuvent exister sous une forme extracellulaire ou intracellulaire. Dans la forme extracellulaire, un virus est une particule de
nature principalement protéique, qui contient de l'acide nucléique. La particule virale appelée virion est métaboliquement inerte et
n'accomplit pas de fonctions respiratoires ou biosynthétiques.
Le virion est la structure par laquelle le génome viral passe d'une cellule, dans laquelle il a été produit, à une autre. Une fois dans la
nouvelle cellule, l'état intracellulaire est initié. La réplication virale se produit alors avec synthèse de nouvelles copies du génome viral
et des composants nécessaires à l'encapsidation de celui-ci.
Les virus ne peuvent pas être observés au microscope optique, des microscopes électroniques sont nécessaires. Ils sont beaucoup
plus petits que les cellules pro- et eucaryotes (à l'exception de Mimivirus). Les particules virales ont une dimension et une forme
variables. Quelle que soit leur taille, elles comportent spécifiquement trois éléments : l'acide nucléique génomique, la capside et
l'enveloppe, cette dernière étant présente seulement pour les virus enveloppés et absentes des virus nus.
L'acide nucléique qui porte l'information génétique spécifiquement virale est d'un seul type, ADN ou ARN, définissant les deux grands
ensembles de virus correspondants. L'ADN est constitué de deux brins complémentaires, excepté ceux des Parvovirus et des
Circovirus qui comportent un seul brin. Sauf exception, l'ARN est constitué d'un seul brin.
Les tailles des génomes viraux sont plus réduites que celles des génomes cellulaires ; le plus petit est d’environ 500 kb, le plus long
d’à peu près 5000kb. Les génomes viraux ne contiennent donc qu’un nombre réduit de gènes. Typiquement ils renferment des gènes
indispensables à l’infection, à la réplication du génome, à la production de virions, à la destruction de la cellule-hôte. La plupart des
virus ne contiennent qu’une seule molécule d’acide nucléique, mais certains en possèdent plus d’une. La variabilité génétique des
virus à ARN est habituellement plus grande que celle des virus à ADN. Cette variabilité leur confère une grande adaptabilité, mais
s'oppose à l'existence de génomes très longs, plus exposés à la survenue de dysfonctionnements suite à l'accumulation de mutations
potentiellement létales. Cette contrainte a été en partie contournée par l'existence de génomes à ARN comportant plusieurs fragments
indépendants qui peuvent compenser, par complémentation et réassortiment, l'impact de ces mutations létales. Un virus réduit à son
acide nucléique inséré dans le génome de la cellule-hôte est nommé provirus.
La capside est un ensemble de protéines associées à l'acide nucléique, formant avec lui la nucléocapside pour laquelle sont décrits
deux modèles. Dans les capsides à symétrie hélicoïdale, les protéines engainent l'acide nucléique enroulé en hélice, l'ensemble ayant
une structure tubulaire. Dans les capsides à symétrie cubique, les protéines forment un polyèdre régulier ayant les propriétés de
symétrie d'un icosaèdre et contenant en son sein le génome viral. Certaines capsides virales ont une structure complexe qui n'entre
dans aucune de ces deux catégories. Les sous-unités protéiques constitutives de la capside sont appelées les capsomères.
L'enveloppe, qui dérive des membranes cellulaires, est constituée principalement de phospholipides au sein desquels s'ancrent des
protéines virales et cellulaires. L'infectiosité des particules virales dépend de l'intégrité de leur structure externe qui interagit avec des
récepteurs cellulaires spécifiques : capside protéique pour les virus nus ou enveloppe lipidique pour les virus enveloppés. L'enveloppe
est plus sensible que la capside à l'action délétère des facteurs physico-chimiques de l'environnement (dessication, chaleur, pH
extrêmes, radiations ionisantes, détergents..) ou de certains compartiments de l'organisme-hôte tels que le tube digestif, d'où une plus
grande fragilité des virus enveloppés et leur moindre transmissibilité dans les compartiments ou milieux concernés.
Pour se multiplier, les virus doivent infecter une cellule hôte. Cette multiplication opère à partir de leur seul matériel génétique par un
processus de réplication et non pas de croissance et division comme chez les bactéries ou les cellules. Le génome viral, libéré dans
la cellule, pilote la fabrication de protéines virales et sa propre réplication en détournant les voies de synthèse de l'hôte, les
composants viraux s'associent ensuite entre eux par auto-assemblage. La transcription des ARN messagers et la réplication du
génome viral mettent en jeu des enzymes, qui souvent, n'existent pas dans la cellule ou dans le compartiment sub-cellulaire où

s'effectue la multiplication virale. Cette restriction impose l'apport de ces enzymes par les particules virales ou leur synthèse de novo
dans la cellule infectée. Cette spécificité de la multiplication virale conduit souvent à l'individualisation au sein de la cellule d'une entité
topographique observable, véritable « usine à virus » assimilée à un corps d'inclusion et désignée sous le vocable de viroplasme.
B. DIVERSITÉ DU MONDE VIRAL
La division traditionnelle du monde viral entre bactériophages infectant les bactéries et virus infectant les cellules animales ou
végétales reposait sur la division binaire du monde vivant élaborée dans les années 1960 entre les procaryotes, dont les cellules sont
dépourvues de noyau, et les Eucaryotes, dont les cellules possèdent un noyau. Cette dichotomie, fondée sur des critères
cytologiques, a été remise en cause dans les années 1970 par les travaux du biologiste moléculaire américain Carl Woese qui, en
comparant les séquences des ARN des petites sous-unités des ribosomes (16S et 18S), ont mis en évidence l'existence de trois
lignées évolutives distinctes de ribosomes. L'existence de ces trois lignées a ensuite été corroborée par les données plus récentes de
la génomique comparée. On a donc proposé de diviser le monde vivant cellulaire en trois domaines : deux domaines comprenant des
êtres vivants procaryotes, celui des archées (Archaea) et celui des bactéries (Bacteria), et un domaine d'êtres vivants composés de
cellules eucaryotes (Eukarya). De façon tout à fait remarquable, l'étude des virus d'archées a confirmé cette classification en trois
domaines du monde vivant. En effet, les virus d'archées forment un ensemble unique de virus aux caractéristiques très originales,
distinct à la fois des virus infectant les bactéries (à quelques exceptions près) et de ceux infectant les eucaryotes.
1. Les bactériovirus (bactériophages)
Les bactériovirus produisent dans leur très grande majorité (environ 90%) des virions de type tête-queue (Figure 38), qui forment
l'ordre des Caudovirales. Leur génome est formé d'ADN double-brin linéaire ou circulaire. Les prototypes en sont les bactériophages
T4 ou λ qui, pendant des années, ont servi de modèles aux pionniers de la biologie moléculaire. L'étude des bactériophages a ainsi
permis de démontrer le rôle de l'ADN en tant que porteur de l'information génétique, de décrypter les mécanismes de la
recombinaison génétique et elle a contribué à démontrer que le code génétique est lu par groupe de trois lettres (triplets).
Certains auteurs pensent aujourd'hui qu'il serait bon d'abandonner l'usage du terme bactériophage qui reflète l'ancienne dichotomie
eucaryote/procaryote, ce qui conduit à confondre virus de bactérie et virus d'archée, et divise artificiellement le monde des virologistes qui étudient les
bactériophages (assimilés aux microbiologistes) et ceux qui étudient les virus, infectant l'Homme, les animaux domestiques ou encore les plantes.
Dans les faits, le terme bactériophage est conservé pour décrire les bactériovirus modèles.
2. Les eucaryovirus
Les eucaryovirus sont beaucoup plus divers que les bactériovirus. Des dizaines de familles ont été décrites, ce qui traduit l'intense
activité de recherche menée sur ces virus chez l'Homme, les animaux domestiques, les plantes et certains insectes. Un très grand
nombre sont des virus à ARN, simple ou double brin, possédant un ou plusierus petits chromosomes linéaires. Les virus eucaryotes à
ADN sont également très variés, leur génome étant composé d'ADN simple ou double-brin et leurs chromosomes pouvant être
circulaires ou linéaires.
Ces virus ont des capsides icosaédriques, pléomorphiques ou filamenteuses. Certains se répliquent dans le cytoplasme des cellules
infectées, d'autres dans leurs noyaux. De nombreux virus infectant des cellules eucaryotes possèdent une enveloppe formée à partir
de la membrane plasmique de la cellule hôte. Certains eucaryovirus à ADN ont des capsides complexes, plusieurs enveloppes et de
grands génomes pouvant coder des centaines de gènes (jusqu'à 1 millier dans le cas de Mimivirus).
3. Les archéovirus
Tous les archéovirus connus possèdent des génomes à ADN (linéaires ou circulaires) de taille relativement réduite (entre 8 et 75 kb).
La plupart de ces génomes sont formés d'ADN double-brin, mais deux archéovirus à ADN simple-brin ont été récemment découverts.
L'un d'entre eux, possède le plus gros génome à ADN simple-brin connu (25 kb). La plupart des archéovirus infectent des archées
hyperthermophiles, présentes dans les biotopes très chauds (sources chaudes terrestres acides en particulier), ou des archées
halophiles, que l'on trouve dans les milieux hypersalés.
C. DIVERSITÉ DES CYCLES : CYCLE LYTIQUE (T4 / E. COLI) ET CYCLE LYSOGÈNE (Λ / E. COLI)
La plupart des phages (comme le phage T4 qui infecte E.coli) n’ont qu’un cycle de vie lytique au cours de laquelle ils tuent leur cellule
hôte en produisant leur descendance. De tels phages sont dits virulents. Quelques phages (comme le phage λ qui infecte E.coli) ont
un cycle de vie lysogénique au cours duquel ils peuvent soit se camoufler temporairement et agir ainsi en phage tempéré (non
virulent) soit entrer en cycle lytique.
1. Les cycles lytiques : exemple du phage T4 chez E. coli
La première étape dans le cycle de vie du phage T4 consiste en l’adsorption d’un virion sur un récepteur membranaire spécifique de
la cellule hôte (Figure 39 et 40). Toute cellule dépourvue de ce récepteur serait protégée d’une infection par T4. Généralement, un
type de phage ne peut infecter qu’une seule espèce de bactéries, et, dans certains cas, uniquement une ou plusieurs souches de
cette espèce. Le nombre de types bactériens dans lesquels un phage peut réaliser son cycle lytique est appelé son spectre d’hôtes.
Après adsorption, T4 injecte son ADN dans la cellule hôte à travers sa queue. La capside phagique vide reste à l’extérieur de la
bactérie, telle un fantôme.
Une fois l’ADN phagique nu présent dans la cellule, les phages adoptent différentes stratégies pour produire les particules filles
(Figure 41). Généralement, l’ADN phagique est transcrit par l’ARN polymérase de l’hôte en ARNm précoces. Plus tard, les ARNm
seraient synthétisés par une ARN polymérase phagique traduite à partir d’un ARNm précoce ; alternativement, l’ARN polymérase
bactérienne serait modifiée afin de transcrire préférentiellement voire exclusivement les gènes du phage. Ces ARNm sont alors
traduits en protéines enzymatiques, régulatrices et structurales. Les protéines régulatrices du phage contrôlent la cinétique
d’expression de plusieurs gènes du phage. Les protéines structurales forment les tête, queue et autres modules protéiques requis
pour l’obtention de particules phagiques complètes. Les enzymes du phage assurent la production d’un grand nombre de génomes
phagiques par réplication, certaines transcriptions, et même parfois la destruction de l’ADN de l’hôte.
2. Les cycles lysogéniques (phage lambda λ d’E.coli)
Il existe deux types de cycles lysogéniques (Figure 42). Dans le cas le plus fréquent, dont l’exemple-type est le phage lambda (λ)
d’E.coli, l’ADN phagique est intégré dans le chromosome de l’hôte. Dans l’autre cas, représenté par le phage P1 d’E.coli, l’ADN
phagique ne s’insère pas dans le chromosome de l’hôte mais se réplique de manière synchrone avec lui, tel un plasmide. Lorsqu’il est
intégré, l’ADN intégré porte le nom de prophage. La production d’un prophage λ intégré se réalise en 4 grandes étapes (exemple chez

E. coli K12) :
- liaison du virus à la perméase au maltose de la bactérie ; injection de l’ADN phagique linéaire dans la bactérie-hôte puis circularisé
par appariement des bases des extrémités redondantes ;
- transcription et traduction de certains gènes phagiques précoces en quelques molécules d’un répresseur protéique et d’une
intégrase (le répresseur inactive alors la transcription de gènes phagiques) ;
- intégration de l’ADN phagique au niveau d’un site spécifique du chromosome de l’hôte à l’aide de l’intégrase, donnant ainsi un
prophage ;
- survie et multiplication de la bactérie (pendant toute cette phase, le prophage est répliqué avec le chromosome de l’hôte).
A près infection par le phage λ, deux protéines sont synthétisées : le répresseur λ et la protéine cro. Le répresseur λ inhibe la
transcription de la protéine cro et inhibe la transcription des autres protéines nécessaires à l’établissement du cycle lytique. La
protéine cro bloque la transcription du gène codant le répresseur λ. L’évolution du système est donc tributaire du rapport de
concentrations entre λ et cro : quand λ est fortement concentré, le cycle lysobène est maintenu ; si la concentration de λ diminue et
que celle de cro augmente, la bactérie entre en cycle lytique.
Le mécanisme par lequel une cellule infectée s’oriente vers un cycle lytique ou lysogénique n’est pas bien compris. De nombreux
détails du cycle lysogénique du phage λ sont connus. Cependant deux conditions semblent favoriser l’établissement du cycle
lysogénique d’un phage tempéré : l’élimination des substances nutritives du milieu et une forte multiplicité d’infection – c’est-à-dire, de
nombreux phages adsorbés par une bactérie. Les phages ne peuvent réaliser un cycle lytique qu’au sein de bactéries ayant un
métabolisme actif. Lorsque les substances nutritives sont éliminées, les bactéries dégradent leurs propres ARNm et protéines avant
d’entrer en dormance. Lorsque les substances nutritives sont à nouveau disponibles, une bactérie dormante non infectée peut
reprendre sa croissance. L’entrée en dormance d’une cellule infectée par un phage interrompt le cycle lytique ; celle-ci perd
habituellement sa capacité à produire des phages et meurt. Mais, si la cellule peut être lysogénisée, la bactérie et le phage peuvent
survivre durant une période de dormance, et le potentiel de production de phages par induction persiste. Si une bactérie lysogénique
subit des altérations au niveau de son ADN, il est avantageux pour le prophage de s’exciser du chromosome bactérien, entrer en
cycle lytique, produire une descendance phagique puis quitter la cellule. Ce processus est nommé induction du prophage.
D. ANALYSE GÉNÉTIQUE CHEZ LES BACTÉRIOVIRUS
1. Titration de virus
Il est possible de compter les virus d'une suspension avec un microscope électronique. Cependant, durant l’infection des bactéries,
d’énormes quantités de bactériophages peuvent être obtenues et étudiées. Souvent, plus de 1010 virus par millilitre de milieu de
culture sont produits.
Il est donc en général plus facile de les quantifier en mesurant leurs effets sur l'hôte. Dans ce contexte une unité infectieuse virale est
la plus petite unité qui cause un effet détectable sur un hôte sensible. En déterminant le nombre d'unités infectieuses par volume de
liquide, une mesure de la quantité de virus, appelée titre, peut être obtenue.
Plusieurs études génétiques, dépendent de notre capacité à déterminer le nombre de phages produits après infection dans des
conditions de culture données grâce à une technique appelée test de plages de lyse (Figure 43). Dans cette technique, une série de
dilutions est effectuée à partir d’une culture pure de bactériophages dérivés d’une culture de bactéries infectées (Figure 44). Un
échantillon ou inoculum de 0,1 mL (appelé une aliquote) d’une dilution est ajouté à un petit volume de milieu nutritif contenant de
l’agar fondu dans lequel quelques gouttes d’une culture bactérienne ont été mélangées. L’ensemble est ensuite transféré sur une
boîte de Pétri contenant une culture bactérienne confluente et sensible au virus.
Quand un virus initie une infection sur une couche ou un tapis de cellules hôtes, une zone de lyse, c'est-à-dire une zone claire voire
translucide peut se former. Cette zone claire est appelée une plage, et il est admis que chaque plage provient des événements de
réplication qui ont débuté à partir d'une seule particule virale. Les plages sont essentiellement des fenêtres dans le tapis de
croissance cellulaire confluente.
Aux quelques endroits où une particule phagique sera déposée, une plage de lyse se formera. En comptant le nombre d’unités
formant des plages (pfu), on a la possibilité de calculer le nombre d’unités virales infectieuses présentes dans l’échantillon initial (titre
phagique). Ce procédé permet aussi de purifier les virus, en effet si une plage de lyse provient d’un seul virus, tous les virus de cette
plage sont en principe génétiquement identiques. On peut donc obtenir une lignée virale pure. De manière générale et comme pour
les cultures bactériennes, le facteur de dilution des cultures virales est important.
2. La transduction
La transduction est par définition, le transfert d’ADN bactérien entre deux bactéries par un virus. On distingue deux types de
transduction : spécialisée et généralisée. Dans les deux cas, de l’ADN bactérien est intégré dans le génome d’un virus mature qui
infecte alors un second hôte bactérien. Généralement les phages transducteurs sont défectifs en raison de l’intégration d’ADN
exogène dans leur génome. Cependant ils sont toujours capables d’infecter une bactérie.
a. La transduction spécialisée
Elle se produit lorsqu’une région spécifique du chromosome bactérien est intégrée dans une particule virale mature. Quatre aspects la
caractérisent :
- les seuls gènes bactériens transduits sont ceux situés à forte proximité du site d’intégration du prophage
- ce phénomène résulte d’une excision imparfaite du prophage du chromosome bactérien
- il y a une possibilité de co-transduction
- les bactéries filles peuvent être des diploïdes partielles
La découverte d’une telle transduction s’est faite chez les phages tempérés au cours du cycle lysogène mais on sait aujourd’hui que
c’est également possible au cours d’un cycle lytique. L’excision anormale d’un propage est un événement rare si bien que la
transduction spécialisée est rare dans la nature. On peut cependant obtenir une transduction à haute fréquence en laboratoire.
b. La transduction généralisée
Dans d’autres cas, l’ADN du phage est complètement exclu et seul de l’ADN bactérien est empaqueté dans le phage. On peut
empaqueter jusqu’à 1% du chromosome bactérien en longueur. Comme dans un transduction spécialisée, les phages sont toujours
capables d’infecter une bactérie. Dans ce cas, lors d’une infection :
- de l’ADN bactérien est injecté dans une cellule bactérienne

- il peut soit rester dans le cytoplasme, soit recombiner avec région homologue du chromosome hôte.
Si l’ADN reste dans le cytoplasme, il ne se réplique pas et peut être transmis à l’une des cellules filles s’il y a une division. Dans ce
cas, si une seule cellule fille hérite des nouveaux gènes, on parle de transduction abortive. Si l’ADN est recombiné, toutes les cellules
filles en hériteront dans leur génome, il s’agit d’une transduction complète.
3. Cartographie détaillée chez les bactériovirus (exemple de T4 / E. coli)
Dans les années 50, Benzer entreprit de faire une cartographie détaillée d'une zone du génome du phage T4 (appelée locus rII).
Grâce à des expériences, il put récupérer quelques recombinants génétiques. Il conçut alors avec succès des expériences permettant
de récupérer les très rares recombinants génétiques résultant d'un échange intragénique. Une telle recombinaison est l'équivalent du
crossing-over eucrayote, mais dans ce cas, à l'intérieur d'un gène plutôt qu'entre deux gènes. Benzer démontra qu'une telle
recombinaison avait lieu entre les ADN de deux phages individuels lors d'une infection simultanée de la bactérie hôte E. coli.
Le résultat final du travail de Benzer fut la carte détaillée du locus rII. Les informations extrêmement détaillées provenant de ces
analyses et le fait que ces résultats aient été obtenus des décennies avant le développement des techniques de séquençage de
l'ADN, soulignent sa remarquable perspicacité sur la structure interne du gène.
a. Le locus rII du phage T4
La première étape d'une analyse génétique est l'isolement d'un grand nombre de mutations dans le gène étudié. Les mutants du
locus rII produisent des plages de lyse repérables après un étalement sur la souche B d'E. coli, ce qui permet de les identifier
facilement. L'approche de Benzer était d'isoler plusieurs mutants rII indépendants – il en obtint au total 20000 – et de réaliser des
expériences de recombinaison afin d'établir une carte du locus (Figure 45). Il supposait que la plupart des mutations, parce qu'elles
étaient obtenues de manière indépendante, étaient localisées au niveau de sites différents du locus rII et pourraient donc servir de
base pour la cartographie. La clé de l'analyse de Benzer est que les phages mutants rII, bien que capables d'infecter et de lyser E.
coli B, ne peuvent lyser une autre souche apparentée, E. coli K12( λ). Les phages sauvages au contraire, peuvent lyser à la fois les
souches B et K12. Le raisonnement de Benzer était que ces conditions fournissaient un système de sélection hautement sensible. Si
les phages des deux souches mutantes infectaient simultanément E. coli B, les échanges entre les sites mutants dans le locus
produiraient de rares recombinants sauvages. Si la population de phages, contenant plus de 99,9 % de phages rII mutés et moins de
0,1 % de phages sauvages, infecte ensuite une souche K12, les recombinants sauvages se reproduiront et produiront des plages
sauvages. Ceci est une étape clé de l'obtention et de la quantification des rares recombinants. En utilisant la technique des dilutions
en série, Benzer put calculer le nombre total de phages rII produits sur E. coli B et le nombre total de recombinants sauvages
capables de lyser E. coli K12. Ces données furent la base du calcul de la fréquence de recombinaison, valeur proportionnelle à la
distance entre les sites mutationnels étudiés à l'intérieur du gène.
b. La complémentation entre mutations rII
Au cours des manipulations précédentes, Benzer s’aperçut que parfois, au cours d’une infection simultanée de K12 par des phages
mutants, certains couples lysaient les bactéries K12. Ce fait était très intriguant puisque seuls les phages sauvages étaient supposés
capables de lyser ces bactéries. Benzer supposa que, lors de l'infection simultanée, chaque souche mutante produisait quelque chose
que l'autre n'avait pas, restaurant ainsi une fonction sauvage. Ce phénomène s'appelle la complémentation (Figure 46). Quand
plusieurs couples de mutations sont testés, chacune des mutations tombe dans l'un ou l'autre des deux groupes de complémentation,
A ou B. Celles qui ne complémentent pas sont placées dans le même groupe de complémentation, alors que celles qui
complémentent sont assignées chacune à un groupe de complémentation différent. Benzer inventa le mot cistron, qu'il définit comme
la plus petite unité génétique fonctionnelle, pour décrire un groupe de complémentation. Dans la terminologie moderne, nous savons
qu'un cistron représente un gène. Nous savons maintenant que les cistrons A et B de Benzer représentent deux gènes distincts dans
ce que nous avons appelé le locus rII. Il y a complémentation quand les bactéries K12 sont infectées avec deux mutants rII, l'un avec
une mutation dans le gène A et l'autre avec une mutation dans le gène B. Il y a donc production des deux produits sauvages puisque
le mutant A fabrique le produit B sauvage et le mutant B fabrique le produit A sauvage. Nous pouvons aussi expliquer pourquoi deux
souches ayant des mutations dans le même gène, disons le cistron A, ne complémentent pas. Dans ce cas, si deux mutants du
cistron A sont combinés, il y aura bien production du produit B sauvage mais aucune production du produit A sauvage. Une fois que
Benzer a pu placer toutes les mutations rII soit dans le cistron A soit dans le cistron B, il a pu reprendre ses études de recombinaison
intragénique, en testant les mutations du cistron A entre elles et les mutations B entre elles.
c. L'analyse de la recombinaison
Sur les 20000 mutations rII, environ la moitié tombait dans chaque cistron. Benzer entreprit de faire la carte de chaque cistron. Par
exemple, en se focalisant d'abord sur le cistron A, si deux mutants rII infectent une culture liquide de bactéries E. coli B et si un
événement de recombinaison se produit entre les sites mutationnels du cistron A, des virus sauvages apparaîtront dans la
descendance avec une faible fréquence. Si des échantillons de la descendance virale sont ensuite étalés sur E. coli K12, seuls les
recombinants sauvages lyseront les bactéries et produiront des plages de lyse. Le nombre total de descendants non-recombinants
sera déterminé en étalant des échantillons sur E. coli B. Le pourcentage de recombinants peut être calculé en comptant les plages à
la dilution appropriée dans chaque cas. Comme dans les expériences de cartographie chez les eucaryotes, la fréquence de
recombinaison est une estimation de la distance entre deux mutations dans le même cistron. Par exemple (Figure 47), si le nombre
de recombinants est égal à 4.103 mL-1 et si le nombre total de descendants est 8.109 mL-1, la fréquence de recombinaison est : 2
(4.103) / 8.109 = 10-6 = 0,000001. Il est nécessaire de multiplier par deux puisque chaque événement de recombinaison donne deux
produits réciproques, et que seul l'un d'eux – le sauvage – est détecté. En menant ces recherches, Benzer s'est aperçu que les
mutations des cistrons n'étaient pas toujours ponctuelles, mais pouvaient également être des délétions.
1
/
4
100%