Protéines ribosomales

1
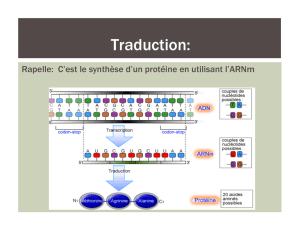
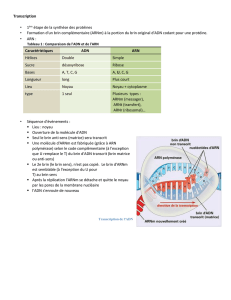
La traduction
1 triplet de nucléotide sur l’ARN
m
(codon) →1 acide aminé
Le mécanisme et les éléments :
polypeptide ribosome
ARN de transfert
acide aminé
Synthèse des protéines:
La machinerie
• Composition des ribosomes procaryotes
– Protéines ribosomales
– ARNs ribosomiques
• ARNs de transfert
• L’aminoacyl-ARNtsynthétase
• Le code génétique
Composition des ribosomes procaryotes
→les plus grosses structures de la cellule
→formés de protéines et d’ARN (ribosomiques)
Ribosome
Sous unité 50S
(grosse sous unité) Sous unité 30S
(petite sous unité)
Modern Genetic Analysis, Griffiths et al, 1999
Procaryotes
Eucaryotes
Les ribosomes : procaryotes versus eucaryotes
Les protéines ribosomales
Plusieurs gènes codant pour les protéines ribosomales
→organisation en opérons
Protéines indispensables à la structuration du ribosome

2
Les ARNs ribosomiques
Synthèse d’un large ARN précurseur contenant les 3 espèces d’ARN ribosomiques
et souvent également 1 ou plusieurs ARNde transfert
Clivage de l’ARN précurseur (Pre-ARNr)→séparation des ARNret des ARNt
Bactéries : soit 1 seule copie soit plusieurs copies (dispersées sur le chromosome)
de cet ADNr
Pre-ARN
r
Maturation du Pre-ARNr
Lafontaine and Tollervey (2006) Encyclopedia of Life Sciences
Escherichia coli
Eucaryotes
Formation de structures tige-boucle
Clivage par ribonucléase (RNaseIII)
Clivage par plusieurs ribonucléases
(Processus plus complexe)
Les principales fonctions des ribosomes sont attribuées à des domaines
spécifiques des ARNr
Les sites A P, E du ribosome Les ARNs de transfert
→Représentent 10-15% des ARNs totaux chez E. coli
→Longueur : 75 à 95 nucléotides
Structure secondaire (en trèfle) :
Structure primaire :

3
Les ARNs de transfert
→Contiennent un triplet de nucléotides (anticodon) complémentaire
du codon sur l’ARNm
ARN
m
ARN
t
→Fixation de l’acide aminé correspondant au niveau du ribose du résidu
adenine situé en 3’ (modification post-transcriptionnelle)
→20 familles d’ARNt(1 famille contenant tous les ARNtcorrespondant un
acide aminé donné = famille d’isoaccepteur)
→Extrémité 3’ se termine avec une séquence CCA (appelée « bras accepteur »)
→Assurent la correspondance entre ARNmet acides aminés
Transcrits en un ARN plus long que la molécule finale
Présence de nucléotides inhabituels
Les ARNs de transfert
→maturation par clivage par des ribonucléases
Couplage entre l’ARNtet l’acide aminé
Les Aminoacyl-(aa-) ARNtsynthétases
ARNtnon chargé ARNtchargé
Processus en 2 étapes :
Activation : aa +ATP ↔aa~AMP + PPi
Transfert : aa~AMP + ARNt↔aa~ARNt+ AMP Reconnaissance très spécifique →important pour la fidélité de la traduction
Reconnaissance de l’ARNt (en vert) par l’ARNt synthétase (en bleu)
Le code génétique
Il est……
→Correspondance entre les différents triplets de nucléotides (codons) et
les 20 acides aminés

4
Universel →le même dans tous les organismes
Ex : La seule exception connue chez les bactérie est : UGA
UGA: codon stop dans la plupart des cas
UGA: tryptophane chez les Mycobactéries
Redondant:
→quelques exceptions :
→souvent plus d’un codon pour 1 acide aminé
Le « Wobble »
Appariement d’un ARN
t
avec plusieurs codons
sur l’ARN
m
Appariement entre la 3ème base du codon et la
1ère base de l’anticodon →pas très strict
Appariement non aléatoire →règle du Wobble
L’usage des codons
→Un même acide aminé peut être préférentiellement codé par un codon
différent en fonction des organismes
Exemples de codons rares chez E. coli
Arginine: AGG, AGA
Isoleucine: AUA
Leucine: CUA
Phenyalanine: CCC
Glycine:GGA
→Tous les codons correspondants à un même acide aminé ne sont pas
utilisés avec la même fréquence d’un organisme à l’autre
→Relation avec la composition en bases de l’ADN de l’organisme ?
(ex : Organisme avec fort % GC →utilisation pour chaque acide aminé des
codons ayant le plus de C et de G)
Ribosome
Région d’initiation Codon stop
initiation
élongation
terminaison
ARN
m
séquence codante
5’P 3’ OH
La traduction
N C Protéine
Traduction: le mécanisme
L’initiation de la traduction
- L’ARNtinitiateur
- Les facteurs d’initiation
L’élongation de la traduction
- La liaison peptidique
- Les facteurs d’élongation
Terminaison de la traduction
Les polysomes
Traduction d’un ARNmcassé
Couplage transcription/traduction
Inhibiteurs de la traduction
L’initiation de la traduction
(chez les procaryotes)
La séquence de fixation des ribosomes (RBS)
Le codon initiateur :
→2 éléments :
- Le codon initiateur
- Le Shine Dalgarno (SD)
AUG : 83% →le plus courant
GUG : 14%
UUG : 3%
chez E. coli
5’ NNNNNAGGAGGU-N5-10-AUGNNNNNN 3’
Codon
initiateur
Shine
Dalgarno

5
Le Shine Dalgarno
Présent chez les procaryotes
(Absent chez les eucaryotes)
Complémentaire de courtes séquences présentes dans l’ARN 16S de la
petite sous unité du ribosome
Localisé en 5’ du codon initiateur (« upstream » ; en amont)
N N N N N A G G A G G U - N5-10-A U G N N N N N N 3’
U C C U C C A
5’
AU
3’ 5’
ARNm
ARN 16S
Codon
initiateur
Shine
Dalgarno
L’ARNtinitiateur :
* fMet-ARNt: un groupement formyl sur la méthionine
* Se fixe sur le site P de la petite sous unité
* Structure différente des ARNtde l’élongation
L’initiation de la traduction
1ère étape : Fixation de la sous-unité 30S sur le site RBS de l’ARNm
2ème étape : Fixation de l’ARNtinitiateur sur le site P de la sous unité 30S
3ème étape : Fixation de la sous unité 50S →formation du complexe 70S
NH
C COOHH
(CH2)2
S
CH3
C
OH
formyl
ARN
t
initiateur
ARN
t
élongation
Les facteurs d’initiation (IFs)
*Accélèrent la formation du complexe initiateur 30S
* Influencent l’interaction entre les codon et anticodon initiateurs
L’initiation de la traduction Initiation de la traduction d’un messager polycistronique
(chez les procaryotes)
 6
6
 7
7
 8
8
1
/
8
100%