[De nombreux processus cellulaires, développementaux et

1 / 8
Concours B ENSA
B-0112B
BIOLOGIE
Durée : 4 heures
L’usage de la calculatrice, d’abaques et de tables est interdit pour cette épreuve.
Si, au cours de l’épreuve, un candidat repère ce qui lui semble être une erreur d’énoncé, il le
signale sur sa copie et poursuit sa composition en expliquant les raisons des initiatives qu’il a
été amené à prendre.
Chaque candidat est responsable de la vérification de son sujet d'épreuve : pagination et
impression de chaque page. Ce contrôle doit être fait en début d'épreuve.
En cas de doute, il doit alerter au plus tôt le chef de centre qui contrôlera et éventuellement
remplacera son sujet.
L’épreuve comprend deux parties indépendantes comptant chacune pour la moitié de
la note.
Première partie : question de synthèse
Le cœur des Mammifères
Les candidats présenteront successivement la révolution cardiaque, puis les activités
mécaniques et électriques cardiaques de l’échelle de l’organe à l’échelle de la cellule.
Les mécanismes de contrôle de ces phénomènes ne sont pas à traiter.
N.B. Il est rappelé aux candidats que la notation prendra en compte non seulement la
clarté, la précision et la concision de l’exposé, mais également la qualité de l’expression, de
l’illustration et de la présentation.

2 / 8
Deuxième partie : étude de documents
Les microARNs (miRNAs) constituent une classe d’ARN qui régulent l’expression génétique
au niveau post-transcriptionnel. Les précurseurs des miRNAs (PremiR) sont transcrits dans
le noyau, puis transportés dans le cytosol où ils subissent des maturations successives,
notamment par l’enzyme Dicer, pour donner finalement les miRNAs matures longs d’une
vingtaine de nucléotides. Ces miRNAs matures se lient en général à leurs ARN messagers
(ARNm) cibles par appariement partiel de leur séquence après avoir été incorporés dans le
complexe RISC (RNA-induced silencing complex). Ceci conduit soit à la dégradation des
transcrits ARNm, soit à l’inhibition de leur traduction.
Les expériences qui suivent présentent quelques conséquences des délétions ciblées de
l’endonucléase Dicer et du miRNA miR-1 sur le développement et le fonctionnement du
cœur de souris.
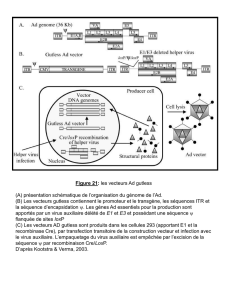
1 - Le système Cre/loxP est appliqué pour invalider spécifiquement le gène Dicer
(cf. Annexe). Pour ce faire, des souris homozygotes pour les allèles Dicer floxés
(Dicer flox/flox) sont croisées avec une souris transgénique dans laquelle la recombinase Cre
est exprimée sous le contrôle du promoteur de la chaîne lourde alpha de myosine (α-MHC)
(MHC Cre). Ainsi, chez cette souris transgénique, la recombinase Cre est exprimée
uniquement dans les cellules musculaires cardiaques. Ces cardiomyocytes comportent par
ailleurs les allèles sauvages du gène Dicer (Dicer +/+). Les descendants d’intérêt (MHC Cre ;
Dicer flox/+) sont identifiés et accouplés avec des souris (Dicer flox/flox).
Les résultats du génotypage de la descendance de ce dernier accouplement sont présentés
en Figure 1.
Justifier la répartition statistique de ces résultats.
Quelles souris vont permettre d’étudier le rôle fonctionnel joué par l’endonucléase
Dicer dans les cardiomyocytes ? Expliquer.
Dans la suite du sujet ces souris seront par convention appelées (-/-).
Toutes les figures qui suivent correspondent à des résultats obtenus chez des souris
nouvelles-nées.
2 - L’expression de deux des miRNAs les plus abondants exprimés dans le cœur, miR-1 et
miR-133, est analysée par Northern blot chez des individus sauvages (+/+) et mutants (-/-)
(Figure 2).
Rappeler en quoi consiste un Northern blot, puis commenter les résultats obtenus.
Que mettent-ils en évidence ?
3 - La fonction cardiaque des souris mutantes (-/-) est analysée par électrocardiogaphie et
comparée à celle d’individus sauvages (+/+) qui servent de contrôles (Figure 3).
Définir les termes diastole et systole, puis rappeler le rôle du ventricule gauche dans
le cycle cardiaque.
Analyser les données présentées. Quelles hypothèses peuvent être émises quant aux
causes des modifications cardiaques observées chez les souris mutantes ?
4 - Des études morphologiques et histologiques sont conduites sur les cœurs de souris
sauvages (+/+) et mutantes (-/-) (Figure 4).
Commenter les documents présentés en soulignant de façon synthétique les
différences observées au niveau de l’organe entier, des tissus et des cellules.
En quoi l’étude de l’ultrastructure des muscles cardiaques et de l’expression de la
connexine conforte-t-elle les hypothèses émises à la question précédente ?

3 / 8
5 – Une nouvelle lignée de souris mutantes est créée, chez lesquelles le miRNA miR-1 a été
éliminé de façon ciblée dans les cardiomyocytes par recombinaison homologue grâce au
système Cre/loxP.
La fonction cardiaque de telles souris mutantes (miR-1-/-) est analysée par
électrocardiographie parallèlement à celle de souris sauvages (wt) (Figure 5).
Rappeler à l’aide d’un schéma explicatif à quoi correspondent les différents intervalles
et ondes visibles sur l’électrocardiogramme des individus sauvages.
Commenter les différences observées. A quels types de défauts cardiaques
pourraient-elles être attribuées ?
6 - Des analyses bioinformatiques de séquences ont permis d’identifier la région non traduite
en 3’ (3’-UTR : 3’-UnTranslated Region) de l’ARNm codant la protéine Irx5 comme étant la
cible du microRNA miR-1. Irx5 est un facteur de transcription qui régule notamment
l’expression du canal potassique Kcnd2. Kcnd2 est un canal voltage dépendant qui contribue
au courant potassique transitoire sortant Ito.
La région 3’-UTR de l’ARNm codant Irx5 est clonée, dans un vecteur d’expression, dans la
région non traduite en 3’ du gène rapporteur codant la luciférase. Ce vecteur d’expression
comporte un promoteur constitutivement actif qui contrôle l’expression de la luciférase.
L’activité luciférase est suivie en fonction de l’ajout de miRNAs après transfection de ce
plasmide rapporteur dans des cellules en culture (Figure 6a). Ensuite, l’expression du gène
Irx5 est évaluée par RT-PCR semiquantitative et Western blot sur des cœurs sauvages et
mutants (Figures 6b & c). Enfin, l’expression du gène Kcnd2 est également étudiée sur des
cœurs sauvages et mutants (Figure 6d).
N.B.1 - La luciférase est une enzyme contrôlant l'oxydation de la luciférine en oxyluciférine
provoquant une émission lumineuse.
N.B.2 - La RT-PCR semiquantitative (qRT-PCR) est une technique destinée à quantifier un
type d’ARN initialement présent dans un échantillon. Ce terme désigne l'utilisation de deux
techniques employées successivement : une transcription inverse suivie d’une PCR en
temps réel.
Commenter les résultats obtenus en Figure 6a. Que démontrent-ils ?
Rappeler sous forme d’un schéma légendé le principe de la réaction de polymérisation
en chaîne (PCR).
Rappeler le principe d’un Western blot en précisant ses différentes étapes et le rôle de
chacune d’elles. Quel est le rôle de l’α-tubuline ?
Interpréter ces différents documents et conclure quant aux fonctions de miR-1 et Irx5.
Quel lien est-il possible de faire entre ces résultats et les hypothèses émises à la
question 5 ?

4 / 8
Figure 1 – Génotypage des produits du croisement de souris (MHC Cre ; Dicer flox/+) et
(Dicer flox/flox). Le tableau rassemble le nombre et le pourcentage des différentes souris issues
du croisement.
Total
288
100%
Dicer flox/+
68
24%
Dicer flox/flox
73
25%
MHC Cre ; Dicer flox/+
72
25%
MHC Cre ; Dicer flox/flox
75
26%
Figure 2 – Analyse par Northern blot des miRNAs miR-1 et miR-133. PremiR : précurseur de
miRNA ; tRNA : ARN de transfert.
Figure 3 - Analyses électrocardiographiques de souris sauvages (+/+) et mutantes (-/-). Les
valeurs proviennent de 5 mesures indépendantes. n, nombre d’individus ; EPVGd, épaisseur
de la paroi ventriculaire gauche en fin de diastole ; EPVGs, épaisseur de la paroi
ventriculaire gauche en fin de systole. bpm : battement par minute ; mm : millimètre.
Génotypes
Fréquence cardiaque
bpm
EPVGd
mm
EPVGs
mm
+/+ (n = 10)
556 ± 42
0,36 ± 0,07
0,62 ± 0,13
-/- (n = 7)
279 ± 64
0,57 ± 0,10
0,71 ± 0,10

5 / 8
Figure 4 (1ère partie, suite page suivante) – Etudes morphologiques, histologiques et
cytologiques de cœurs de souris sauvages (+/+) et mutantes (-/-). (a) Cœurs vus en face
ventrale. (b) Rapport poids du cœur/ poids du corps. Les barres horizontales indiquent les
moyennes. (c) Coupes sagittales de cœurs entiers après coloration hématoxyline – éosine.
Ces réactifs permettent de colorer le noyau en bleu/violet et le cytoplasme en rose.
(d) Myocarde ventriculaire gauche à plus fort grossissement. (e) Micrographies électroniques
à transmission de myocarde ventriculaire.
Pour toutes ces études, les clichés des cœurs sauvage et mutant sont au même
grandissement.
a
c
b
d
+/+ -/-
Poids cœur / Poids corps (mg/g)
a
c
b
d
+/+ -/-
Poids cœur / Poids corps (mg/g)
+/+ -/-
Poids cœur / Poids corps (mg/g)
ee
 6
6
 7
7
 8
8
1
/
8
100%