L`expression du patrimoine génétique

Thème 1 : La Terre dans l’Univers, la Vie et l’évolution du vivant
Partie 1 : Expression, stabilité et variation du patrimoine génétique
Chapitre 3 : L’expression du patrimoine génétique
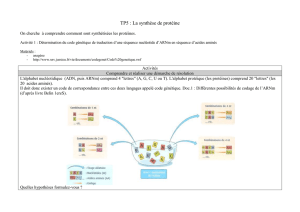
TP 6 : Du gène à la protéine (2)
On utilisera le logiciel Anagène.
1) Nous commencerons par afficher les séquences des deux brins de l’ADN, de l’ARNm codant et du
polypeptide qui nous intéressent. Pour cela, sélectionner « fichier », « thèmes d’étude », choisir
« expression de l’information génétique » puis « globine beta », « gène et ARNm codant ». Refaire
l’opération, mais sélectionner « séquence peptidique ».
2) Convertir la séquence d’ARNm codant en polypeptide. Pour cela, sélectionner la ligne « béta ARNm
codant » dans la fenêtre d’affichage. Cliquer ensuite sur « traiter », « convertir les séquences ».
Choisir « peptidique », « traduction simple » et cocher « résultat dans la fenêtre
d’affichage/édition ».
Noter la longueur de la séquence d’ARNm et celle du polypeptide qui résulte de la traduction du
message génétique : pour cela, sélectionnez la séquence qui vous intéresse et cliquer sur l’icône (i)
bleue.
Formuler une première hypothèse sur le nombre de nucléotides qui codent pour un acide aminé.
3) Comparer le polypeptide résultant de cette traduction (« Pro-Bêta ARNm ») avec le polypeptide de
la banque de données (Polypeptide Bêta) et effectuer et effectuer une comparaison simple. Que
constatez-vous ?
4) Fermer la fenêtre de comparaison, dupliquer la séquence d’ARNm : pour cela, sélectionner la
séquence « beta ARNm codant », cliquer sur « édition », « dupliquer la séquence ». Nous allons
modifier cette seconde séquence d’ARNm. Pour cela, il faut la déprotéger (« options » puis
décocher « protéger les données »)
Inverser la séquence : sélectionner la copie « Beta ARNm codant », « édition », « inverser la
séquence ». Elle s’affiche en « i-Beta ARNm codant »
Traduire la séquence « i-Beta ARNm codant » : sélectionner la séquence puis « traiter », « convertir
les séquences », sélectionner « ARN messager » et « peptidique » et désélectionner « résultat dans
la fenêtre d’affichage/édition ». Faire une traduction simple.
Comparer visuellement le polypeptide résultant de cette traduction (« Pro-i-Beta ARNm codant »
obtenu à partir de l’ARNm inversé) à celui de référence de la banque de données (polypeptide beta).
Qu’est-ce que cela montre quant à la lecture du message de l’ARNm ?
Fermer toutes les fenêtres sans quitter le logiciel.
Il existe quatre types de nucléotides différents, qui codent pour 20 acides aminés.
1 nucléotide pour un acide aminé : … combinaisons
2 nucléotides pour un acide aminé : … combinaisons
3 nucléotides pour un acide aminé : … combinaisons
Etant donné qu’il n’y a que 20 acides aminés, quelle remarque pouvez-vous faire ?
Pour répondre à cette remarque, vous allez synthétiser 4 molécules d’ARNm particulières de trois
nucléotides chacune : « fichier », « créer » choisir « ARN ». Entrer les séquences GUU, GUC, GUA,
GUG, puis avec AAU, AAC, AAA, AAG.
Sélectionner les 8 séquences et convertissez les séquences en séquences protéiques : « traiter »,
« convertir les séquences », cocher « ARN messager » et « peptidique », « traduction simple »,
désélectionner « résultat dans la fenêtre affichage/édition ». Quelles conclusions en tirez-vous ?
L’hypothèse d’un codage par triplets est-elle validée ?

Fermer toutes les fenêtres et ouvrir à nouveau les séquences d’ADN, d’ARNm et peptidique de la
chaine beta de l’hémoglobine : choisir « expression de l’information génétique », « globine
beta », « gène et ARNm codant » puis refaire l’opération en sélectionnant « séquence peptidique ».
Dirigez-vous, pour la séquence d’ARNm, à l’aide de l’onglet déroulant, au niveau des nucléotides
442 à 444. Que constatez-vous au niveau de la protéine ?
Dupliquer comme précédemment la molécule d’ARNm et inversez la séquence.
Faire ensuite une traduction simple de la séquence inversée (« traiter », « convertir les séquences »,
sélectionner « ARN messager » et « peptidique » et désélectionner « résultat dans la fenêtre
d’affichage/édition ». Faire une traduction simple) et constater l’effet du triplet constitué par les
nucléotides n°25-26-27 de l’ARNm inversé. Il existe trois triplets de ce type (on appelle codon tout
triplet de nucléotides) : ce sont des codons-stop.
Vous pouvez observer la totalité du code génétique en cliquant sur l’icône AUG.
Envisager la conséquence sur la synthèse d’une protéine d’une mutation entraînant l’apparition
d’un codon ATC dans le brin transcrit de l’ADN codant pour cette protéine.
Il est possible de faire traduire un ARNm de globine humaine dans une cellule de germe de blé.
Quelle information supplémentaire cela vous apporte-t-il sur le code génétique ?
Réponses attendues :
ARNm : 444 nucléotides / Peptide : 147 acides aminés
444/147 ≈ 3. On en déduit que 3 nucléotides codent pour un acide aminé
Ce sont les mêmes
« Pro-i-bêta » est beaucoup plus courte que « bêta »
Il existe un sens de lecture du message
4/16/64 combinaisons possibles
Il existe plus de combinaisons que nécessaire
Certains acides aminés doivent être codés par plusieurs séquences
GUU/GUC/GUA/GUG : VAL
AAU/AAC : ASN
AAA/AAG : LYS
L’hypothèse du codage par triplet est validée
441 444 : plus d’acides aminés (arrêt de la séquence protéique)
25-26-27 : arrêt de la séquence protéique
ATC UAG (ARN m) : codon stop : arrêt
La séquence protéique est plus courte
La protéine est certainement non fonctionnelle
Le code génétique est universel : il est le même pour tous les êtres vivants
COURS :
I- La relation gènes-protéines
L’expérience de Beadle et Tatum (1941) : le champignon Neurospora crassa réalise la synthèse du
tryptophane grâce à trois réactions successives qui impliquent chacune l’intervention d’une enzyme.
A la suite d’une mutation chez une souche du champignon, la souche mutante devient incapable de
synthétiser le tryptophane sauf si on lui ajoute un des trois enzymes. Beadle et Tatum montrent ainsi
que le gène muté a déterminé la synthèse d’une enzyme différente qui est inactive.
D’autres expériences ont également prouvé par la suite qu’un gène détermine la synthèse d’une
protéine, c’est-à-dire un enchainement ordonné d’acides aminés : on dit qu’un gène code une
protéine. La séquence en acides aminés d’une protéine dépend de la séquence nucléotidique du gène
qui la code.

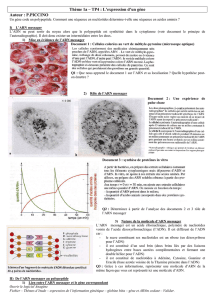
II- La transcription : première étape de l’expression d’un gène
Algue acétabulaire (unicellulaire)
Les protéines du « chapeau »
d’une algue acétabulaire
peuvent être synthétisés dans
un fragment d’algue énuclée
(noyau enlevé). On peut en
déduire que la synthèse des
protéines a donc lieu dans le
cytoplasme. Or les gènes qui
codent ces protéines sont
localisés dans le noyau (les
chromosomes ne sortent
jamais du noyau). On peut
donc penser qu’il existe un
intermédiaire entre l’ADN et
les protéines. Si on détruit les
ARN dans un fragment d’algue
énuclée, cela entraîne l’arrêt
de la synthèse protéique
(arrêt de la fabrication du
chapeau). L’ARN est donc bien
l’intermédiaire entre l’ADN.
D’autres expériences le
montrent :

L’ARN est un acide nucléique proche de l’ADN, synthétisé dans le noyau puis exporté dans le
cytoplasme.
Caractéristiques :
Sucre : Ribose (Acide-Ribo-Nucléique)
Un seul brin
Bases azotées AUCG
Dans le noyau, le gène qui s’exprime sert de modèle pour la fabrication d’une molécule d’ARN : c’est
la transcription. Un ARN pré messager, complémentaire du brin codant de l’ADN est d’abord produit.
Après une maturation, il devient un ARN messager qui sera traduit en protéines dans le cytoplasme.
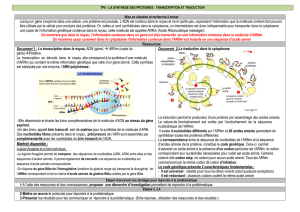
III- La traduction, seconde étape de l’expression d’un gène
Dans le cytoplasme, l’ARN messager sert de modèle pour la synthèse de la protéine, codée par un gène
qui s’exprime. A chaque codon (triplet de nucléotides) correspond à un acide aminé dans la protéine
synthétisé : c’est le code génétique (document ci-dessus).
Il est universel (commun à tous les êtres vivants), redondant (plusieurs codons possible pour un acide
aminé) et univoque (un codon correspond à un seul acide aminé). Dans le cytoplasme, la traduction de
l’ARN messager en protéine est effectuée au niveau des ribosomes.
Ces petits organites lisent la séquence et assurent la liaison des acides aminés les uns aux autres.
Exemple :
ATGAACCCGGTTAAATGTTAA -> Brin non codant
TACTTGGGCCAATTTACAATT -> Brin codant
AUGAACCCGGUUAAAUGUUAA -> ARN messager
Met-Asp-Pro-Val-Lys-Cys-STOP -> Protéine (acides aminés)

IV- Les différentes échelles de phénotypes
Exemple de phénotype : la drépanocytose.
A l’échelle de l’organisme (macroscopique) : anémie chronique, crises articulaires…
A l’échelle cellulaire : hématies déformées en forme de faucille, rigides et cassantes
A l’échelle moléculaire : le 6ème acide aminé de la β-globine est une valine à la place de d’un
acide glutamique. Les molécules de HBS s’agglutinent pour former des fibres. Les différentes
échelles de phénotypes sont dépendantes les unes des autres.
Le phénotype d’un malade dépend de son génotype (mutations) mais aussi de son environnement
(certaines situations favorisent les crises).
La changement d’un acide aminé dans la séquence polypeptidique de l’hémoglobine entraîne
Modification de la solubilité des HBS qui s’agglutinent les unes aux autres pour former
des fibres rigides.
Déformation des hématies, qui prennent alors une forme de faucille et qui deviennent
rigides.
Les hématies, du fait de leur manque de souplesse, circulent mal dans les capillaires.
Les capillaires sanguins peuvent alors être obstrués.
L’oxygénation des tissus se fait mal, le sujet présente des difficultés respiratoires qui
entrainent une anémie grave
1
/
5
100%