version PDF - Flash informatique

FI 2 – 24 février 2009 – page 1
So m m a i r e
Fi 2/2009
1 Cuda
Francis Lapique
2 DIT-info
3 FlashiPhone
François Roulet
9 Comment extraire des don-
nées depuis Internet!
David Portabella &
Charles Rey
12 Nokia – Students’ Call for
Proposals
Aurore Amaudruz
13 Spécial été 2009 – appel aux
articles & concours de la
meilleure nouvelle
14 Nouvelle autorité de
certification
Martin Ouwehand
14 Projets AAA de Switch
Pierre Mellier
14 38e Workshop en HPC
Vittoria Rezzonico
15 FileMaker Pro
Anne-Cécile Follonier
16 Programme des cours
21 Le cluster du pauvre
Martin Rentschle &
Bob de Graffenried
22 Départ d’un homme réseau
Jacqueline Dousson
23 Vers une informatique
axiologique?
René Berger
S u i t e e n
p a g e 4
éCOLE POLYTECHNIQUE
FéDéRALE DE LAUSANNE
p/a EPFL - Domaine IT - CP 121 - CH 1015 Lausanne 15 - tél. +41 21 69 322 11 - Web: http://dit.epfl.ch
Les GPU ne sont pas
uniquement faits pour les
consoles de jeux
Francis.Lapique@epfl.ch, Domaine IT
gpu v s Cpu: t o b e , o r
n o t t o b e
La figure 1 présente un comparatif,
en performance par Watt, entre Nvidia
et Intel qui s’affrontent par déclarations
interposées quant à l’avenir respectif du
GPU et du CPU.
Nvidia a ainsi récemment dé-
claré que les processeurs n’avaient plus
aucun avenir et qu’ils seraient très
prochainement supplantés par les puces
graphiques!
Intel, répond en déclarant que
Cuda ne sera qu’une intéressante note
de bas de page dans les annales de
l’histoire informatique et d’expliquer
que malgré les gains exceptionnels
NV38(03)
FX5950 Ultra
NV45(04)
6800 Ultra
G80(07)
8800 Ultra
G92(08)
9800 GTX
G71(06)
7900 GTX
GT200(08)
GTX280
Kentsfield
QX9770
Yorkfield
QX68500
Bloomfield
i7965XE
Presler
EE965
Smithfield
D840
Prescott
P4570
Northwood
P4HT3.4
0 10 20 30 40 50 60 70
Months since January 2003
8
7
6
5
4
3
2
1
0
–1
Computational Efficiency [GFlopsW]
Computational Efficiency: GPU vs. CPU (Source: heise.de, tomshardware.com. wikipedia.org)
Nvidia Consumer Graphics Card
Intel Desktop CPU
fig. 1
promis par la technologie Cuda, un
problème de taille subsiste. Les gains de
performances sont bien présents, mais
peu de développeurs sont en mesure de
l’exploiter pleinement.
Sur ce dernier point, l’année der-
nière un cours pour les architectures
multi-core sur la base de la bibliothè-
que Intel reading Building Blocks
(TBB) était annoncé en posant la
question: que va-t-on en tirer au ni-
veau applicatif? Je dois avouer que les
résultats peu encourageants, de mon
point de vue, avec des double ou quadri
cœurs m’ont décidé à reporter ce cours.
Je ne sais pas si Cuda ne sera qu’une in-
téressante note de bas de page dans les
annales de l’histoire informatique, mais
aujourd’hui, nous
le verrons dans la
suite de cet article,
cette technologie
nous offre des gains
de performance
50x à 100x pour
de faibles coûts de
développement en
terme de temps et
de prix.

FI 2 – 24 février 2009 – page 4
ar C h i t e C t u r e
fig. 2 – la carte NVIDIA GeForce GTX 280
L’architecture des cartes GeForce compte un ensemble de
multiprocesseurs (ou SM pour Streaming Multiprocessor) au
nombre de 30 pour la carte GeForce GTX280 (voir fig. 2).
Chacun de ces SM est équipé de 8 ALUs (Arithmetic Logic
Unit).
Une unité SIMT (Single-Instruction Multiple-read)
prend en charge la création, l’ordonnancement et l’exécution
de warps (groupe de 32 threads, le terme vient des machines
à tisser, il désigne un ensemble de fils de coton).
Un thread CUDA n’a pas tout à fait le même sens qu’un
thread CPU, c’est un élément de base des données à traiter.
A l’inverse des threads CPU, les threads CUDA sont extrême-
ment légers ce qui signifie qu’un changement de contexte est
une opération peu coûteuse. Un warp est exécuté tous les 2
cycles; par exemple, pendant les deux premiers cycles, warp
8 exécute l’instruction 11, pendant les deux cycles suivants,
wrap 1 exécute l’instruction 42. Chaque multiprocesseur
peut prendre en compte 32 warps soit 1024 threads.
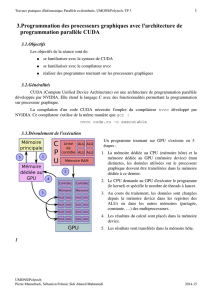
mo d è L e d e p r o g r a m m a t i o n
Au niveau programmation on ne manipule pas direc-
tement ces warps, on écrit des noyaux de programmation
ou kernels.
Ces kernels se subdivisent en blocs composés de warps.
Chaque SM dispose d’une mémoire partagée ou Shared
Memory de 16’384 bytes et de 16’384 registres de 32 bits
(fig. 3). La mémoire partagée n’est pas une mémoire cache,
c’est un espace ouvert à la programmation.
Shared Memory
SP
SP
SP
SP
SP
SP
SP
SP
SFU SFU
Instruction Fetch/Dispatch
Instruction L1 Data L1
Streaming Multiprocessor
fig. 3 – architecture SM
Les GPU ne sont pas uniquement faits pour les consoles de jeux
S u i t e d e L a p r e m i è r e p a g e
L’environnement de développement CUDA compte
quelques extensions au langage C, des librairies, un compi-
lateur (nvcc) et un pilote (fig. 5). Sans trop rentrer dans le
détail de ces extensions on trouve des qualificateurs comme
__global__ pour désigner un kernel, __device__ pour une
fonction exécutée et appelée depuis le GPU, il désigne éga-
lement une variable dans l’espace Global memory à savoir la
DRAM du GPU (1Go pour la carte GTX280), __shared__
pour une variable Share memory.
Application
CPU
GPU
CUDA Libraries
CUDA Runtime
CUDA Driver
fig 5 – architrcture CUDA
Un kernel:
__global__ void Func(oat* parameter)
est appelé d’une façon un peu particulière;
Func<<< Dg, Db, Ns >>>(parameter);
où Dg représente la taille de la grille en nombre de blocs, Db la
taille du bloc en nombre de threads et Ns un espace mémoire
optionnel dynamiquement alloué par bloc dans la mémoire
partagée et entre parenthèses la liste des paramètres.
fig. 4 – Modèle de programmation

FI 2 – 24 février 2009 – page 5
Les GPU ne sont pas uniquement faits pour les consoles de jeux
Total amount of global memory 1'073'479'680 bytes
Number of multiprocessors 30
Number of cores 240
Total amount of constant memory 65'536 bytes
Total amount of shared memory per block 16'384 bytes
Total number of registers available per block 16384
Warp size 32
Maximum number of threads per block 512
Maximum sizes of each dimension of a block 512 x 512 x 64
Maximum sizes of each dimension of a grid 65'535 x 65'535 x 1
Maximum memory pitch 262'144 bytes
Un jeu de variables intégrées ou built-in permet
d’identifier un thread. La ligne de commande qui suit est un
exemple de calcul d’index de thread à travers ces variables
built-in:
int idx = blockIdx.x * blockDim.x +
threadIdx.x;
où blockIdx contient l’index du bloc dans la grille, threa-
dIdx celui du thread dans le bloc et blockDim le nombre de
thread par bloc.
La fonction __synchthreads() permet de synchroniser
les threads à l’intérieur d’un bloc.
Pour résumer les limitations matérielles de la carte
GTX280 qui nous sont données par le programme device
Query:
Le développement de votre application au niveau kernel
(grille et bloc) ne vous affranchit pas de penser aux niveaux
multiprocesseurs d’exécution de warps et au niveau ALU
(Unité Arithmétique et Logique, sous-partie d’un processeur
capable d’effectuer des opérations définies par un jeux d’ins-
tructions) d’identifiant de bloc et de thread. Deux points
très importants qui concernent l’optimisation: le conflit
de banques de la mémoire partagée et la coalescence de la
mémoire globale. Vous pouvez approfondir ces deux notions
dans le Programming Guide CUDA [7].
Sh a r e d m e m o r y e t C o n F L i t d e
b a n q u e S
fig. 6 – conflit de banques
L’espace de communication Shared memory, espace de
16 Ko, est organisé en 16 banques (fig. 7).
fig. 7
La figure 6.a présente un exemple de conflit: les thread 0
et 8 accèdent à la même case 0 de la banque. C’est quelque
chose qu’il faut éviter, car dans ce cas l’accès est sérialisé avec
une chute de la bande passante (fig. 6.b). En cas de non-
conflit, l’accès est presque aussi rapide que
les registres contrairement aux 400 cycles
d’horloge de la mémoire globale.
gL o b a L m e m o r y e t
C o a L e S C e n C e
La mémoire globale souffre d’une
latence importante (400 à 600 cycles
d’horloge pour un accès). Pour remédier à
ce problème, CUDA donne la possibilité d’accéder à un bloc
de plusieurs cases mémoires ou coalescence. La coalescence
est réalisée au niveau du demi-wrap (un cycle d’horloge du
SIMT soit 16 threads) si on accède à des régions mémoires
de 32, 64 ou 128 octets.
On obtient également des gains de vitesse si l’on accède
à des cases mémoires voisines et dans l’ordre des indices de
threads. Les figures 6a et 6c illustrent ces propos.
Il existe deux types d’accès non coalescents. Le premier
est dû au fait que les threads n’accèdent pas dans l’ordre à
des cases voisines (fig. 6b); le second est dû à un problème
d’alignement (fig. 6c). Le premier thread d’un warp doit
accéder à une case mémoire multiple de 64.
fig. 6.b-6c – coalescence
Une dernière remarque lorsque différents threads d’un
même wrap travaillent dans deux branches différentes d’un
même if, l’impact sur les performances peut être important,
car le GPU est obligé d’exécuter le warp sur plusieurs cycles
d’horloge.
0 2 4 6 8 10 12 14 16 18 20 22 24 26 28 30
0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15
0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15
offset=1 Address (ai) stride is 2 resulting in 2-way bank conflicts
Bank
ai
thid
int ai = offset * (2*thid+1)-1;
fig. 6.a

FI 2 – 24 février 2009 – page 6
Les GPU ne sont pas uniquement faits pour les consoles de jeux
fig. 8 - CUDA processing flow – auteur: Tosaka
__global__ void sum(oat* A, oat *B,
oat* C, int width)
{
....
3 exécution du noyau dans le GPU
.....
unsigned int idx = threadIdx.y * width + threadIdx.x;
C[idx] = A[idx] + B[idx];
}
int main()
{
...
Allocation des vecteurs de données à traiter par le GPU
....
unsigned int mem_size=10*10*sizeof(oat);
oat *a; cudaMalloc((void**)&a, mem_size);
oat *b; cudaMalloc((void**)&b, mem_size);
oat *c; cudaMalloc((void**)&c, mem_size);
oat input_a[100]; // size = 10*10
oat input_b[100];
oat output_c[100];
...
1- Envoi des données vers le GPU
...
cudaMemcpy(a,input_a, mem_size, cudaMemcpyHostToDevice);
cudaMemcpy(b,input_b, mem_size), cudaMemcpyHostToDevice);
dim3 dimBlock(10, 10);
...
2 - demande d’exécution du noyau dans le GPU
...
sum<<<1, dimBlock>>>(A, B, C);
...
4 - retour du résultat vers le CPU
....
cudaMemcpy(output_c, C, mem_size,cudaMemcpyDeviceToHost);
...
}
code 1 – Somme de deux vecteurs
Sm i t h -Wa t e r n a m a v e C Cuda
Revenons à notre titre Les GPU ne sont pas
uniquement faits pour les consoles de jeux. La
technologie NVIDIA CUDA veut montrer au
marché HPC (Calcul Haute Performance) qu’elle
peut jouer un rôle important dans les années qui
viennent. Il est vrai que son site présente une pa-
lette d’applications (fig. 9) qui vont bien au-delà
de son métier de niche [6, 8].
Pour nous faire notre propre opinion, nous
avons acquis deux cartes GTX280 et décidé de
porter un algorithme de recherche heuristique
utilisée en bio-informatique permettant de trou-
ver les régions similaires entre deux ou plusieurs
séquences de nucléotides ou d’acides aminés.
La recherche d’alignements entre séquences
génomiques est une des tâches fondamentales de
la bio-informatique. L’objectif est de localiser des
régions semblables dans des séquences d’ADN
ou des séquences protéiques. Une application
typique est l’interrogation d’une banque avec un
gène dont la fonction est inconnue. Les résultats
retournés correspondent à des segments similaires
présentant un indice de ressemblance élevé. Plus
exactement, l’information utile est un alignement,
c’est-à-dire deux portions de séquence où sont
précisément indiqués les appariements entre
nucléotides (pour l’ADN) ou les appariements
entre acides aminés (pour les protéines).
On doit parcourir systématiquement l’en-
semble des banques, de la première à la dernière
séquence. Il existe plusieurs algorithmes pour extraire des
alignements. Les premiers, comme celui de Smith-Waterman
élaboré en 1981 [1], utilisent des techniques de programma-
tion dynamique et possèdent une complexité quadratique.
Les seconds, apparus en 1990, comme le programme BLAST,
se basent sur une heuristique très efficace (recherche de points
d’ancrage) permettant de cibler directement de courtes zones
identiques potentiellement intéressantes.
Nous avons porté notre choix sur celui de Smith-Water-
man qui a fait l’objet ces derniers temps de plusieurs publi-
cations concernant son implémentation sur GPU [2-5].
fig. 9
he L L o Wo r L d !
Pour découvrir le monde CUDA voici un exemple de
somme de deux vecteurs (voir code 1). Les numéros indiqués
dans le code renvoient à ceux de la figure 8.

FI 2 – 24 février 2009 – page 7
Les GPU ne sont pas uniquement faits pour les consoles de jeux
#include "cuda.h"
#include "mex.h"
/* Kernel to square elements of the array on the GPU */
__global__ void square_elements(oat* in, oat* out, int N)
{
int idx = blockIdx.x*blockDim.x+threadIdx.x;
if (idx < N) out[idx]=in[idx]*in[idx];
}
/* Gateway function */
void mexFunction(int nlhs, mxArray *plhs[], int nrhs, const mxArray *prhs[])
{
/* Call function on GPU */
square_elements<<<dimGrid,dimBlock>>>(data1f_gpu, data2f_gpu, n*m);
}
code 2
L’a L g o r i t h m e d e Sm i t h -Wa t e r m a n
L’algorithme de Smith-Waterman évalue une ressem-
blance locale entre deux séquences A et B de taille m et n
respectivement. Les éléments de A et B sont notés a1, … am
et b1, … bn. La matrice de similitude entre éléments W(ai,
bj) et les pénalités d’ouverture et d’extension de gap Ginit et
Gext sont également données.
On calcule les n x m matrices suivantes:
Hi,j=max {0
Ei,j
Fi,j
Hi–1,j–1 – W(ai,bj)}
Ei,j=max
Fi,j=max
{
{
Ei,j–1 – Gext
Hi,j–1 – Ginit
Fi–1,j – Gext
Hi–1,j – Ginit
}
}
La procédure qui permet de trouver l’alignement à partir
de la matrice est la suivante: à partir de la cellule d’arrivée,
remonter vers la(les) cellule(s) voisine(s) de score maximal;
itérer pour arriver à la cellule initiale.
Pour illustrer le propos, la figure 10 donne un exemple
concret de la procédure de recherche locale entre deux sé-
quences ADN avec une expression simplifiée de la matrice:
H(i,j) = max{0, H(i,j-1)-a, H(i-1,j)-a, H(i-1,j-1) +
W(S1i ,S2j ) }, avec a=1, W(S1i ,S2j ) =+2 si les résidus i et j
sont identiques et W(S1i ,S2j )=-1 dans le cas contraire.
fig. 10 - Example of the Smith-
Waterman algorithm to compute
the local alignment between two
DNA sequences ATCTCGTATGAT
and GTCTATCAC. The matrix
H(i,j) is shown for the linear gap
cost a= 1, and a substitution
cost of +2 if the characters are
identical and –1 otherwise. From
the highest score (+10 in the
example), a traceback proce-
dure delivers the corresponding
alignment, the two subsequences
TCGTATGA and TCTATCA
Vous trouverez le pseudo-code d’implémentation à al
référence [2]. Une des particularités de ce code est de char-
ger dans la mémoire de texture une matrice acides aminés
-requête dite Query-profile.
En partant du travail de ces deux auteurs, cet algorithme
a été porté sur une machine de bureau (Intel 3.0 Ghz, cache
6MB, express Chipset. 4 GB DDR3, 2x Nvidia GTX 280,
system Linux kernel 2.6.27).
Vous pouvez faire des tests en-ligne depuis la page gpu.
epfl.ch/sw.html. Pour lancer la requête cliquer Query, car cette
page contient par défaut un jeu de paramètres de test. La règle
d’attribution du service est des plus simples premier arrivé,
premier servi. La base de données (Swiss-Prot) compte un peu
plus de 400 mille séquences et environ 145 millions de résidus.
La réponse doit parvenir au bout de quelques secondes.
fig. 11
À la lumière des résultats présentés à la figure 11, évitez
le mode CPU pour des requêtes plus grandes qu’une dizaine
de résidus, car n’oubliez pas que vous multipliez le temps par
un facteur d’environ 50.
matLab
Les figures 12.a et 12.b donnent une idée du facteur
d’accélération que l’on peut obtenir avec l’extension CUDA
pour MATLAB que l’on peut obtenir depuis le site Nvidia
[9]. Vous trouverez dans cette extension un script nvmex à
l’image du script mex pour compiler l’application (code 2.0)
 6
6
1
/
6
100%