TP 8 bis

TP 8 : Structure et composition de l’ADN de différents êtres vivants
Objectif : Trouver des points communs dans la structure et la composition de l’ADN de différents êtres
vivants.
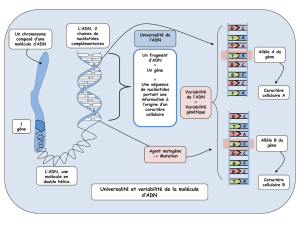
Historiquement, ce sont Watson et Crick qui ont élaboré le modèle de la molécule support de l’hérédité. Leur
travail a reposé sur des connaissances établies auparavant :
- la composition chimique de l'ADN ;
- les clichés de diffraction aux rayons X d'ADN cristallisé, clichés dus principalement à Rosalind Franklin
- les travaux de Erwin Chargaff;
- les analyses en microscopie électronique.
Il est possible de découvrir cette structure en 3D avec le logiciel Rastop.
A partir de l’exploitation de l’ensemble des documents fournis et de l’utilisation du logiciel Rastop,
comparez la structure et la composition des molécules d’ADN de deux êtres vivants ; vous communiquerez
vos résultats à l’aide d’un document numérique composés d’un texte et de schémas complétés.
Matériel :
- un ordinateur relié à une imprimante,
- logiciels : Rastop = logiciel de visualisation moléculaire + fiche technique Rastop.
Libre office, logiciel de dessin vectoriel
Capturino = logiciel de capture d’images
- documents numériques :
- fichiers rastop : dans le dossier moladn, ouvrir par ex. les fichiers « adn-hum1 » et « adn-lev »
- à compléter = fichiers de dessin vectoriel dans le dossier ADN.
Doc1.sxd : schématisation possible d’un nucléotide
doc2.sxd : schématisation possible de la complémentarité des bases
doc3.sxd : schématisation possible d’une molécule comme si elle était à plat (en forme d’échelle)
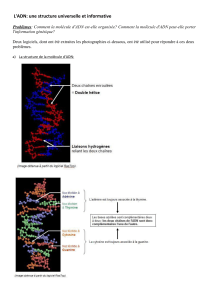
Document 1 : Un peu d’histoire des sciences : Le modèle établi par Watson et Crick en 1953.
« Nous souhaitons proposer une structure pour l’acide désoxyribonucléique (ADN). Cette structure a deux
chaînes, enroulées en hélice autour du même axe […] Les bases azotées sont à l’intérieur de l’hélice et les
phosphates à l’extérieur. Les sucres (1) sont perpendiculaires aux bases auxquels ils sont reliés. Chaque chaîne
consiste en une succession d’un motif [base azotée, sucre, phosphate] (2) qui se répète tous les 3,4 Angström
(3). Un tour d’hélice est réalisé tous les 10 motifs. »
D’après J.D. Watson et F.H. Crick, Nature, 25 avril 1953.
(1) sucre = désoxyribose, (2) motif = nucléotide, (3) un angström = 10 -10 m.
Document 2 : Les travaux de Rosalind Franklin et Maurice Wilkins.
Ces clichés montrent une figure en croix,
caractéristique des structures en hélice.
Rosalind Franklin dirigeait des rayons X sur une
fibre de l'épaisseur d'un cheveu qui contenait
des millions de brins d'ADN B (la forme
hydratée) extraits du thymus de veau.
Découverte par R. Franklin, cette forme B
hydratée est celle qui se trouve à l'état naturel
dans les cellules.

Document 3 : Résultats d’une hydrolyse de l’ADN
L’hydrolyse totale de la molécule d’ADN (coupure de l’ADN en molécules élémentaires par l’eau et l’acide
chlorhydrique à 120°C pendant 2 heures) libère trois constituants chimiques :
‐ des groupements phosphates notés P
‐ des sucres simples, les désoxyriboses notés D
‐ des bases azotées.
Un nucléotide est constitué par la liaison de trois éléments ; Un acide phosphorique = groupement phosphate
(PO4H3), un sucre (C5=désoxyribose) et d’une base azotée.
Document 4 : à compléter
Titre :
Document 5 : Les résultats d’Erwin Chargaff (1950) : Pourcentage de chaque nucléotide dans différentes
molécules support du matériel génétique.
Organismes
Nucléotide à
Adénine
Nucléotide à
Guanine
Nucléotide à
Cytosine
Nucléotide à
Thymine
Homme
30,9
19,9
19,8
29,4
Blé
27,3
22,7
22,8
27,1
Levure
31,3
18,7
17,1
32,9
Poule
28,8
20,5
21,5
29,2
Bactérie
24,7
26
25,7
23,6

Aides à la résolution :
1. Indiquez, d’après le modèle de Watson et Crick, la structure d’une molécule d’ADN et les constituants de
base.
2. Indiquez quelle information sur l’ADN donnait les travaux de R. Franklin.
3. Avec Rastop, indiquez le nombre de chaînes composant la molécule et comment elles sont associées.
Pour cela :
Ouvrir le logiciel Rastop en cliquant sur le raccourci du bureau.
Afficher la molécule « adn-hum1 » et la molécule « adn-lev » (aller à Fichier, ouvrir, choisir le
dossier « Sciences de la vie et de la terre », dossier Mme Van Belle, dossier seconde, dossier ADN,
dossier molécules ADN.
Afficher la molécule en mode « boules et bâtonnets » en cliquant sur
Manipuler la molécule : pour faire pivoter la molécule, cliquer dessus + déplacer la souris ;
pour agrandir la molécule : Majuscule + cliquer + déplacer la souris de haut en bas.
Dans le menu « Atomes », choisir « colorer » par « chaînes ».
Remarque :
Utilisez capturino pour capturer les images importantes.
4. La molécule est formée de chaînes et chaque chaîne est composée de plusieurs sous-unités appelées
nucléotides ; avec Rastop, indiquez de combien de types différents de nucléotides la molécule est formée et
leur nom.
Pour cela :
Dans le menu « Atomes », choisir « colorer » par « forme »
Passer le curseur sur la molécule et lire dans le cadre « Res » en bas de l’écran.
5. Représentez les nucléotides sur doc1.sxd
6. Comparez les teneurs en A et T pour chaque organisme et les teneurs en C et G de chaque organisme ;
Indiquez alors quels sont les nucléotides qui s’associent deux à deux permettant aux deux chaînes de s’associer.
7. Représentez les nucléotides complémentaires sur doc2.sxd.
8. Synthèse : A l’aide des informations collectées et de la structure observée à l’écran, schématisez sur
doc3.sxd une molécule comme si elle était à plat (en forme d’échelle) et faites un texte pour décrire sa
structure.
1
/
3
100%