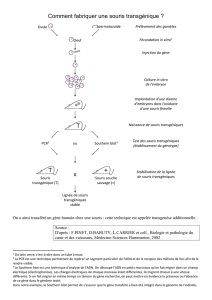
3.2. Les animaux transgéniques

http://bioinfo.unice.fr/enseignements/EPU_2005/EPU_1-
2/rapports_2nd_semestre/La_lymphangiogen%E8se-1.pdf
3.2. Les animaux transgéniques
Les animaux transgéniques, exprimant des gènes étrangers (knock-in) ou ne pouvant pas
exprimer l’un de leur propre gène (knock-out), sont étudiés en recherche fondamentale,
comme animaux modèles pour les maladies humaines, la sélection animale et la production
de protéines pharmaceutiques. Le modèle animal le plus important en recherche médicale
est la souris (Mus musculus), pour sa simplicité d’élevage et une physiologie proche de
l’homme.
Afin de minimiser les effets secondaires liés aux caractères héréditaires hétérozygotes, la
production d’animaux transgéniques s’effectue sur des lignées « inbreds ». Ces lignées de
souris par exemple sont quasiment homozygotes après 7 à 10 croisements entre frères et
soeurs.
La production d'un animal transgénique doté dans chaque cellule d'un nouveau morceau
identique d'ADN semble à première vue un processus fort laborieux. Les scientifiques
peuvent utiliser une caractéristique fondamentale du développement animal : il est possible
de déterminer de quelle cellule proviennent toutes les cellules d'un animal donné. En
d'autres termes, chaque animal est né d'une seule cellule, qui s'est divisée et redivisée
pour finalement produire les milliards de cellules qui le constituent (86).
En conséquence, pour s'assurer que chaque cellule de l'animal contient le même morceau
d'ADN, il est possible d’ajouter de l'ADN à une entité cellulée avant qu'elle ne commence à
se diviser. Le nouvel ADN s'intègre au génome de la cellule et, en théorie, sera présent
dans toutes les cellules qui constituent l'animal.
Pour créer des animaux transgéniques, il faut modifier les cellules germinales (comme
l'ovule et le spermatozoïde) qui sont les seules cellules animales capables de donner
naissance à une nouvelle progéniture (87,88).
3.2.1. La micro-injection
Pour produire un animal transgénique, on peut entre autre avoir recours à la microinjection.
Le nouvel ADN est injecté directement dans un ovule fécondé (zygote) avant qu'il
ne commence à se diviser. Le nouvel ADN s'intègre dans un chromosome dans le noyau et
sera dès lors présent dans chaque cellule de l'animal qui en résultera dont les cellules
germinales. Cela signifie que le nouvel ADN sera transmis aux descendants de cette lignée
(86). (figure 15)
27
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.2709
Figure 15 : Construction de souris transgénique par micro-injection nucléaire
Des pipettes en verres très fines sont utilisées spécialement pour ces manipulations ce qui permet de
percer l’oocyte et percer le pronucléus male qui est plus gros que la femelle. Une solution aqueuse
contenant des inserts est ensuite directement injectée dans le pronucléus. L’ADN des clones introduit
peut intégrer l’ADN endogène au sein des chromosomes, formant ainsi le transgène. Suite à cela, les
oocytes qui survivent sont réimplantés dans l’oviducte de femelle pseudo enceinte. Les souriceaux
naissants résultent du développement des embryons implantées et sont alors étudiés par PCR pour
observer la présence ou non de l’ADN d’intérêt.
En résumé, l’introduction stable de gènes étrangers au niveau embryonnaire consiste
en :
- l’injection l'ADN contenant le gène d'intérêt dans le noyau d'un oeuf récemment
fécondé.
- l’intégration du gène dans l'ADN génomique.
- l’implantation de l'embryon dans une mère porteuse.
- l’obtention d'un animal transgénique.
La micro-injection est un processus aléatoire. Il se peut que l'ADN injecté ne s'intègre pas
au chromosome du zygote. Aucune des cellules de l'organisme résultant ne possédera alors

le nouvel ADN.
Dans certains cas, le nouvel ADN ne s'intègre pas au chromosome avant la division de la
cellule. Il en résulte un animal mosaïque, qui contient le nouveau gène dans certaines de
ses cellules, mais pas dans l'ensemble (87).
28
En général, l'ADN injecté contient un gène qui code pour une protéine particulière destinée à
s'exprimer dans l'animal. De nombreuses régions d'un chromosome donné sont
inaccessibles aux enzymes responsables de l’initiation de l'expression génique.
L'emplacement de ces régions varie en fonction du type de cellule et permet de s'assurer
que seuls les gènes appropriés sont « actifs » dans toute cellule donnée. Si le transgène
s'intègre dans l'une de ces régions inaccessibles, la protéine associée ne sera pas produite
ou sera peut-être produite uniquement dans certains types de cellules (87).
En conséquence, les animaux produits à l'aide de cette méthode doivent faire l'objet de tests
approfondis visant à déterminer si :
- toutes les cellules de l'animal contiennent le transgène
- tous les types de cellules voulus produisent le produit protéique du gène
3.2.2. Les rétrovirus
A partir d’un stade embryonnaire précoce, les cellules peuvent être totipotentes ou au moins
pluripotentes et peuvent permettre l’entrée d’ADN étranger dans la lignée germinale. Une
des méthodes qui permet à l'ADN étranger d'intégrer les chromosomes des cellules hôte
emploie des rétrovirus. Ces virus à ARN ont la capacité de passer de la forme ARN à ADN
grâce à la transcriptase inverse avant d'intégrer des génomes cellulaires (89).
Les rétrovirus s’incorporent aléatoirement dans l'ADN de l’hôte. Cet ADN, appelé l'ADN
proviral,
peut être efficacement exprimée par la cellule hôte et répliqué avec l’ADN de l’hôte. Les
rétrovirus intègrent rarement et au hasard l’ADN les cellules accessibles et ne tuent
normalement pas leur hôte (90). (figure 16)
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.2357
Figure 16 : Le cycle de vie du rétrovirus
Les particules virales contiennent le génome sous forme d’ARN et une transcriptase inverse qui sont
entourés d’une enveloppe lipoprotéique puis d’une capside protéique. Une copie d’ADN double brin
du génome viral peut s’intégrer dans l’ADN de l’hôte. Ce dernier synthétise l’ARN et les protéines
virales, qui s’auto assemblent et bourgeonne à partir de la cellule même. L’hôte n’est pas tué.
29
Les vecteurs rétroviraux présentent de nombreux avantages par rapport à d'autres systèmes
de transfert de gène. En effet, ils ont la capacité d’infecter un large éventail de cellules
animales, ils expriment les gènes d’intérêt de manière élevée, et permettent également
l’absence de synthèse des protéines virales après infection de l’hôte (91).
Des gènes étrangers ont été efficacement insérés dans les cellules de mammifères en les
infectant avec des vecteurs dérivés du virus murin de leucémie de Moloney, qui peut
accepter des insertions jusqu’à 6 Kb.
Deux autres vecteurs viraux sont fréquemment employés :
- Virus de Vaccinia : il contient un ADN de grande taille, se réplique dans le cytoplasme des
cellules de mammifères, et arrête la synthèse des protéines de la cellule hôte.
- Baculovirus : il infecte les cellules d'insecte, qui peuvent être facilement cultivées. Les
larves d'insecte atteintes de ce virus permettrent la production massive de protéine.
Des vecteurs basés sur ces virus ont été utilisés pour exprimer des inserts d'ADN de
manière efficace (90)
30
3.3. Le Knock Out
Le Knock Out (KO) est une technique permettant d’inactiver sélectivement un gène en
remplaçant celui-ci par un allèle mutant provenant d’un autre organisme. Cette technique qui
détruit la fonction du gène est un outil puissant qui permet d’observer les processus
cellulaires et les mécanismes dans lesquels le gène est initialement impliqué.
Les souris Knock Out, permettent l’étude du développement, du comportement et de

l’activité physiologique d’un gène. Ils peuvent également servir de systèmes modèles
utiles afin d’étudier certaines maladies génétiques humaines et son implication dans le
cancer (92).La méthode de production des souris Knock Out implique les étapes suivantes :
(figure 17)
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=mcb.figgrp.1981
Figure 17 : Processus général de la formation de souris Knock Out :
Les cellules souches embryonnaires hétérozygotes exprimant une mutation pour un gène d’intérêt (X)
et les cellules homozygotes pour le marqueur d’un gène ( ici peau noire) sont injectées dans le
blastocyte embryonnaire de 4,5 jours qui est homozygote pour un autre marqueur ( peau blanche).
L’embryon obtenu est alors implanté chez une souris femelle enceinte. Les souriceaux obtenus sont
des chimères, ce qui est révélé par leur pelage noir et blanc. Les souris chimériques sont croisées
avec des souris blanches. Les souriceaux issus de ce croisement ont des cellules ES dérivées dans
leur lignées germinales. Par isolement de l’ADN de ces souris il est possible d’identifier l’allèle muté.
Un croisement entre ces souris noires produit des individus homozygotes pour l’allèle muté. Des
souris Ko sont alors obtenues.
31
1. L’allèle mutant est introduit par recombinaison homologue dans les cellules souches
embryonnaires (cellules ES). La recombinaison homologue est un échange de fragments
d’ADN entre deux molécules au niveau de séquences nucléotidiques homologues. (figure
18) Le KO est basé sur la recombinaison homologue entre un ADN exogène et un ADN
génomique. Ce mécanisme a été mis en évidence en 1980 par injection directe d’ADN dans
des cellules de mammifères (92).
Les facteurs qui favorisent la recombinaison homologue sont:
- La taille de la région homologue
- Le fort pourcentage d’homologie
- L’ADN sous forme linéaire (par rapport à forme circulaire)
- Le mode de transfection
http://www.genoway.com/commun/img/homologous2.jpg
Figure 18 : Insertion d’un gène par recombinaison homologue
Un transgène est inséré par recombinaison homologue au niveau d’un locus d’intérêt. Une fois
réarrangé, le transgène peut alors être exprimé.
2. Une fois que les clones positifs de cellules ES sont obtenus, ils sont micro injectés dans
les blastocytes de souris receveuses.
3. Les blastocytes sont alors réimplantés dans l’utérus de souris.
4. Les cellules ES injectées chez la souris femelle colonisent le blastocyte et après un
croisement avec des souris mâles, des souris chimères sont obtenues dans lesquelles
le transgène est exprimé en fonction des tissus. Ces cellules peuvent être exprimées
dans les lignées somatiques ou germinales, ces dernières étant très importantes afin
d’obtenir des gamètes transgéniques.
5. Des souris chimériques sont croisées avec des souris normales afin d’obtenir des
cellules germinales avec le matériel transgénique.
6. Seules les souris hétérozygotes sont croisées entre elles ce qui permet d’obtenir environ
25% de souris homozygotes pour l’allèle mutant. Il est important de savoir que les souris
homozygotes sont parfois non viables et meurent au stade embryonnaire. Afin de vérifier ce
point, il est nécessaire de sacrifier des souris hétérozygotes fécondées par les mâles
hétérozygotes et de génotyper les embryons à différents temps embryonnaires. Ceci
permettra de montrer l’importance de ce gène dans le développement embryonnaire.
32
La létalité embryonnaire due à l’invalidation d’un seul allèle du gène est observée dans
le cas du VEGF. Il est néanmoins possible d’observer l’effet de l’invalidation du gène à l’état
adulte en passant par des mutations silencieuses provisoires (système CRE/Lox P) (92).
Le système Cre/loxP a donc été utilisé pour générer des souris Knock Out pour le gène
du VEGF-C permettant ainsi d’observer les conséquences de son absence dans certains
tissus spécifiques chez la souris adulte (86).
La Cre est une recombinase (de la famille des intégrases) du bactériophage P1. Cre est
une protéine de 38 kDa qui catalyse la recombinaison entre deux sites de

reconnaissance, les sites LoxP.
LoxP est une séquence d’ADN de 34 pb avec à ses extrémités des palindromes de 13
nucléotides (il est donc impossible de trouver cette séquence dans un génome eucaryote).
Les deux sites peuvent être très éloignés l’un de l’autre et être tout de même recombinés par
la Cre. Cette recombinase fonctionne dans les cellules eucaryotes génétiquement modifiées
comportant deux sites loxP. En fonction de leur orientation, il peut se produire une délétion
ou une inversion (93). (figure 19)
http://www.adbt.com/DESS_EGPR/index.php
Figure 19 : La délétion d’exon
Deux sites loxP sont insérés de part et d’autre d’un exon du gène à invalider (triangle vert). L’exon est
dit « floxé ». Cette construction peut être utilisée pour remplacer le gène sauvage par recombinaison
homologue dans des cellules ES. On vérifie que des souris transgéniques homozygotes (pour l’exon
floxé) sont parfaitement saines. Ces souris peuvent alors être croisées avec des souris transgéniques
exprimant la Cre recombinase dans un tissu particulier (le génome de ces souris contient le gène Cre
sous la dépendance d’un promoteur tissu spécifique).
La première étape est de générer des souris exprimant LoxP. Elles sont crées par
recombinaison homologue formant ainsi des mutations silencieuses.
Dans un deuxième temps, des souris exprimant Cre sont synthétisées. Pour ce faire, un
vecteur d’expression codant pour cette recombinase est injecté dans des embryons. Ces
derniers lorsqu’ils sont positifs au vecteur sont réimplantés dans la souris et sont à l’origine
des souris Cre +/+ (86).
33
Enfin dans un troisième temps, les souris Cre +/+ et LoxP +/+ sont croisées. Seules les
souriceaux exprimant Cre +/+ et LoxP +/- sont viables. Ces souris expriment Cre dans un tissu
spécifique (un promoteur tissu spécifique a été cloné en amont du gène codant pour Cre)
ce qui va entraîner l’élimination du gène d’intérêt inséré entre les deux sites LoxP.
L’induction de la Cre entraîne donc une délétion (93) (figure 20).
http://www.ncbi.nlm.nih.gov/books/bv.fcgi?rid=hmg.figgrp.2741
Figure 20 : Le système de recombinaison Cre-loxP.
Le système de recombinaison Cre-loxP peut être utilisé pour inactiver un gène désiré.
(A) La recombinaison homologue standard utilise des cellules ES, dans lesquelles trois sites LoxP
sont introduits avec un marqueur M sur le locus cible A. Après la transfection, on génère des souris
LoxP. Le gène codant pour la recombinase Cre est ajouté (enzyme qui induit des recombinaisons
entre deux sites LoxP) ce qui permet de donner plusieurs produits qui sont ensuite sélectionnés. Des
recombinants de type 1 sont utilisés pour générer des souris dans lesquelles le locus cible est situé
entre deux sites LoxP.
(B) Un croisements entre une souris dont le gène d’intérêt A est situé entre deux régions LoxP et une
souris transgénique comportant un gène codant pour la recombinase Cre lié a un promoteur tissu
spécifique est réalisé. Les souris issues de ces croisements, qui comportent les deux constructions,
expriment la recombinase Cre dans le tissu désiré, et la résultante de la recombinaison contenue
entre les deux site LoxP dans ces cellules spécifiques inactive ainsi le locus cible A.
34
1
/
4
100%