L Les défi s de la médecine personnalisée en oncologie DOSSIER THÉMATIQUE

290 | La Lettre du Cancérologue • Vol. XXI - n° 6 - juin 2012
DOSSIER THÉMATIQUE
La médecine personnalisée
Les défi s de la médecine
personnalisée en oncologie
The challenges of personalized cancer medicine
M. Kamal*
* Unité d’investigation clinique,
institut Curie, Paris.
L
e domaine de la médecine a connu, ces
10 dernières années, des avancées considérables
sur les plans scientifique et technologique,
qui ont frayé la voie à la médecine personnalisée
(MP). Le US National Cancer Institute définit la
MP comme “une forme de médecine qui utilise les
informations moléculaires et environnementales du
patient (gènes, protéines) pour prévenir, diagnos-
tiquer et traiter”.
Les résultats cliniques suite aux différents traite-
ments de chimiothérapie dans plusieurs types de
cancer se sont considérablement améliorés ces
30 dernières années. Cependant, la survie globale
des patients a relativement stagné, ce qui a encou-
ragé le développement de nouvelles stratégies
thérapeutiques (1). La recherche fondamentale et
translationnelle a permis l’identifi cation de certains
mécanismes cellulaires et moléculaires responsables
du développement de la tumeur et des métastases
dans différents types de cancers humains. Ces décou-
vertes scientifi ques ont concrètement contribué au
développement des thérapies ciblées, c’est-à-dire
des médicaments qui ciblent d’une manière effi cace
une ou plusieurs anomalies moléculaires spécifi ques.
En oncologie, le potentiel thérapeutique de la MP
est incontestable. Les succès révolutionnaires de
l’imatinib ou du trastuzumab ont constitué une base
tangible pour la thérapie ciblée.
Le séquençage complet du génome humain (2) a
permis d’obtenir une quantité immense de nouvelles
informations, qui ont souligné les innombrables
variations génétiques entre les individus. Récem-
ment, les nouvelles technologies “-omiques”
− incluant le séquençage à haut débit, les SNP (Single
Nucleotide Polymorphism) ou la CGH (Compara-
tive Genomic Hybridization), qui met en évidence
les arrangements moléculaires, ainsi que l’ana-
lyse du transcriptome et la RPPA (Reverse Phase
Protein Array) − ont rendu possible le profilage
moléculaire du patient dans des délais et des coûts
raisonnables. La MP s’impose ainsi comme une
option attrayante dans la lutte contre le cancer (3).
La mise en œuvre de la MP requiert la connaissance
des anomalies moléculaires et génétiques respon-
sables de l’oncogenèse, la disponibilité des thérapies
ciblées et l’identifi cation de sous-groupes de patients
qui peuvent bénéfi cier de ces thérapies ciblées via
le développement d’algorithmes de stratifi cation.
Cependant, l’application clinique de la MP va au-delà
de la connaissance scientifique et des thérapies
ciblées et nécessite la prise en compte de plusieurs
facteurs scientifi ques, techniques, réglementaires,
éthiques et fi nanciers (4).
Hétérogénéité
et évolution des tumeurs
Chaque type de cancer inclut des sous-types
présentant des signatures biologiques différentes
qui se distinguent par leur pronostic clinique et leur
réponse au traitement. Toute tumeur est unique,
dans le sens où différentes tumeurs issues d’un
même tissu peuvent avoir des caractéristiques
moléculaires complètement distinctes (5-7).
Cet argument a souvent été utilisé pour l’identifi -
cation de signatures moléculaires ayant pour but de
classifi er les différents types de cancer (8). Ce type
de classifi cation est utilisé dans certains types de
cancers, comme celui du sein (9), mais sa validité
n’est pas encore établie.
Deux facteurs cruciaux sont à prendre en compte
pour établir le profil moléculaire de la tumeur :
la complexité et l’hétérogénéité du tissu tumoral,
qui contient à la fois des cellules tumorales et
des cellules stromales normales adjacentes (5).

La Lettre du Cancérologue • Vol. XXI - n° 6 - juin 2012 | 291
Résumé
La médecine personnalisée est définie comme “une forme de médecine qui utilise les informations moléculaires et
environnementales du patient (gènes, protéines) pour prévenir, diagnostiquer et traiter”. En oncologie, son potentiel
thérapeutique est incontestable. Les avancées technologiques des 10dernières années, en particulier la percée des
technologies à haut débit, ont permis d’identifier une multitude de mutations somatiques et d’autres anomalies
génétiques dans plusieurs types de cancers humains. Cependant, plusieurs problèmes continuent à freiner aujourd’hui
l’implémentation active de la médecine personnalisée. Les différentes classifications moléculaires et les signatures
génétiques pronostiques et prédictives ne sont pas assez robustes pour prédire l’efficacité d’une thérapie ciblée.
Deplus, si le nombre de thérapies ciblées augmente quotidiennement, les résultats des essais cliniques ne s’avèrent
pas satisfaisants. Finalement, la transition vers la médecine personnalisée engendre plusieurs défis incluant la
standar disation des tests moléculaires, l’identification et la validation de biomarqueurs, la stratification des patients,
le coût de la thérapie ciblée, sans compter plusieurs considérations réglementaires et éthiques.
Mots-clés
Biomarqueurs
Thérapies ciblées
Techniques à haut
débit
Hétérogénéité
tumorale
Éthique
Summary
Personalized medicine is
defi ned as “a form of medicine
that uses information about a
person’s genes, proteins, and
environment to prevent, diag-
nose, and treat disease.” It is
generally accepted today that
personalized medicine has
great potential in the treatment
of many types of cancer.
The new high throughput
screening techniques allowed
the identification of several
actionable molecular altera-
tions in different kinds of
cancer and paved the way for
personalized medicine in the
fi eld of oncology. However, the
molecular classifi cations and
prognostic signatures are not
powerful enough to predict the
effi cacy of targeted therapies.
In addition, targeted thera-
peutic agents are increasingly
available for clinical applica-
tions, yet these promising drugs
have produced disappointing
results when tested in clinical
trials, mainly due to the lack
of predictive biomarkers and
appropriate statistical designs.
For a successful transition
towards personalized medicine
in oncology, several challenges,
including the standardization of
molecular screening assays, the
identifi cation and validation of
relevant biomarkers, patient
stratifications in addition to
fi nancial, regulatory and ethical
issues need to be addressed.
Keywords
Biomarkers
Targeted therapy
High throughput screening
techniques
Tumor heterogeneity
Ethics
Les biopsies du tissu tumoral doivent ainsi être
impérativement caractérisées, et la proportion des
cellules tumorales par rapport aux cellules normales
doit être bien établie. Un taux important de cellules
tumorales est indispensable pour pouvoir corréler
les altérations moléculaires identifi ées à la tumeur
elle-même. Dans l’essai BATTLE, première étude
prospective randomisée et achevée qui se fonde
sur l’analyse du profi l moléculaire des patients pour
orienter la décision thérapeutique dans le cancer
du poumon, des échantillons histologiques de plus
de 200 cellules tumorales étaient pris en compte
pour l’identifi cation des biomarqueurs d’intérêt (10).
Dans certains essais nationaux, comme SAFIR01 ou
SHIVA, au moins 50 % de cellules tumorales sont
requises dans l’échantillon.
En réalité, la situation est beaucoup plus complexe.
Plusieurs études de séquençage à haut débit ont
démontré une hétérogénéité intratumorale.
Des biopsies d’un même site tumoral peuvent avoir
des caractéristiques moléculaires signifi cativement
distinctes (11-14). Une hétérogénéité intratumo-
rale concernant l’amplifi cation de HER2 chez des
patientes atteintes de cancer du sein invasif est
corrélée à une diminution de la survie sans progres-
sion. Les variations de l’amplification de HER2
(Human Epidermal growth factor Receptor 2) dans
certains sous-clones de cellules tumorales peuvent
donc être associées à la progression tumorale (14).
Dans une autre étude récente, des analyses géno-
miques de plusieurs biopsies provenant d’un même
carcinome rénal primitif et de différents sites méta-
statiques ont révélé des signatures pronostiques
complètement différentes (13). L’hétérogénéité
intratumorale représente un défi majeur pour la
mise en œuvre de la MP. La caractérisation molé-
culaire de plusieurs biopsies d’une même tumeur
semble être nécessaire pour éviter de sous-estimer
cette hétérogénéité, qui peut expliquer la résistance
à certaines thérapies ciblées. Plusieurs mutations
dans les exons 19 et 21 de l’EGFR (Epidermal Growth
Factor Receptor) détectées dans le cancer bron-
chique non à petites cellules (CBNPC) semblent être
prédictives de la réponse au géfi tinib (15). La muta-
tion T790M de l’EGFR a été corrélée à la résistance
au géfi tinib (16). Dans de rares cas, cette même
mutation, présente dans une population mineure
de cellules tumorales de CBNPC, alors que les autres
cellules tumorales révèlent des mutations sensibles
au géfi tinib, semble pouvoir expliquer pourquoi
les répondeurs potentiels s’avèrent réfractaires au
traitement (12).
Un autre aspect de l’hétérogénéité tumorale est
la variation des caractéristiques moléculaires de
la même tumeur avant et après traitement, et de
la tumeur primitive par rapport à ses métastases.
En effet, une étude récente réalisant le séquen-
çage ciblé de 1 264 gènes impliqués dans le cancer
colorectal a démontré des différences génétiques
signifi catives entre les tumeurs primitives et les
métastases hépatiques (17).
Les analyses génomiques permettent aujourd’hui une
étude exhaustive et spatio-temporelle des anoma-
lies moléculaires de plusieurs biopsies d’une même
tumeur et de ses métastases. La véritable interro-
gation porte sur la faisabilité de toutes ces analyses
dans des délais et des coûts raisonnables afi n de
permettre une décision thérapeutique appropriée.
Identifi cations de biomarqueurs
et transfert vers la clinique
Actuellement, la majorité des études visant la décou-
verte de biomarqueurs sont rétrospectives ; elles sont
utiles pour connaître l’incidence, la prévalence et la
validation clinique des marqueurs d’intérêt, ainsi
que pour l’optimisation des tests (18). L’utilisation
de biomarqueurs en clinique se fonde sur une alté-
ration moléculaire spécifi que (une mutation pilote,
une amplifi cation ou une translocation génique, une
modifi cation épigénétique, etc.) qui n’est souvent
pas suffi sante pour sélectionner la thérapie ciblée
adéquate. En effet, plusieurs altérations moléculaires
sont nécessaires pour l’activation ou l’inhibition
d’une voie de signalisation spécifi que, elle-même
régulée par plusieurs mécanismes de rétrocontrôle
négatif ou positif. Des voies de signalisation alter-
natives sont souvent activées suite à l’utilisation des
thérapies ciblées (1). Les biomarqueurs prédictifs
sont donc indispensables pour pouvoir stratifi er les
patients selon leurs profi ls moléculaires spécifi ques
afin de prédire l’efficacité d’une ou de plusieurs
thérapies ciblées.

292 | La Lettre du Cancérologue • Vol. XXI - n° 6 - juin 2012
Les défi s de la médecine personnalisée en oncologie
DOSSIER THÉMATIQUE
La médecine personnalisée
Les différentes études utilisant le séquençage à haut
débit, qu’il s’agisse de l’exome ou du génome, ont
permis l’identifi cation de centaines de milliers de
mutations différentes, dont les conséquences et
les implications dans l’étiologie du cancer sont loin
d’être établies (19-21). La majorité des mutations
détectées sont passagères et ne jouent pas un rôle
actif dans le développement du cancer. L’identifi -
cation de mutations pilotes, activement responsables
de l’oncogenèse, constitue un grand défi . Le séquen-
çage d’un grand nombre d’échantillons (plusieurs
centaines) est nécessaire pour identifi er les muta-
tions les plus fréquentes, mais une validation fonc-
tionnelle in vitro et in vivo reste indispensable pour
pouvoir désigner une mutation comme “pilote” (22).
D’autre part, les autres techniques à haut débit
comme le SNP, la CGH et les analyses protéomiques
produisent également une multitude de données qui
doivent être intégrées et analysées (23). L’intégration
des données provenant de plusieurs échantillons et
techniques est nécessaire pour établir d’une manière
fi able une vision complète des anomalies molécu-
laires activement responsables du développement
du cancer (24). L’intégration des données est très
complexe et fastidieuse, du fait de l’hétérogénéité
des modèles expérimentaux, des protocoles, de
la qualité des données et de leur représentation.
La fréquence et la distribution de faux-positifs et
de faux-négatifs doivent être résolues pour pouvoir
intégrer et interpréter les différentes données (25).
Le rôle de la bio-informatique devient donc crucial
pour la gestion des différentes données génomiques
et le développement d’algorithmes élaborés pour
intégrer les données cliniques du patient et son profi l
moléculaire dans le but de prédire des corrélations
entre les anomalies moléculaires et l’évolution des
différents types de cancer (23).
Les différentes analyses moléculaires et bio-
informatiques sont très coûteuses et longues, et
doivent impérativement être suivies par une vali-
dation clinique. La validation des différentes signa-
tures moléculaires doit être réalisée par des équipes
indépendantes et sur des populations indépendantes
pour ne pas surestimer la performance de ce type
de signature, ce qui a été le cas dans certaines
études (26).
À ce jour, plusieurs biomarqueurs ont été identifi és
mais seule une petite partie d’entre eux est validée
en clinique (27). Récemment, des altérations molécu-
laires comme l’amplifi cation de HER2 dans le cancer
du sein (28, 29), les mutations de BRAF dans le méla-
nome (30) ou de l’EGFR dans le CBNPC (31) ont été
validées en clinique grâce à leurs valeurs pronostiques
et prédictives de l’effi cacité des traitements qui les
ciblent. D’autre part, certaines anomalies − à l’image
des mutations de l’IDH1 (isocitrate deshydrogénase)
dans le glioblastome multiforme − sont des muta-
tions pilotes pour lesquelles aucune thérapie ciblée
n’existe pour le moment (32).
Chaque biomarqueur identifi é a très peu de chances
d’être ratifié pour une utilisation clinique (1).
Une fois validés, les tests de détection des diffé-
rents biomarqueurs doivent être standardisés pour
éviter la variabilité d’un laboratoire à un autre. Cette
standardisation des tests doit prendre en compte
plusieurs aspects : le type de prélèvement à analyser,
la méthode utilisée pour la biopsie, les protocoles
d’extraction de l’ADN, de l’ARN et des protéines, le
stockage des échantillons, le choix des gènes et des
protéines à tester, le type de plateforme utilisée ainsi
que l’interprétation des données. Plusieurs mesures
d’assurance qualité doivent être ainsi développées et
le personnel du laboratoire, formé. Ces différentes
mesures ajoutent des dépenses importantes à
estimer.
La validation des biomarqueurs en clinique est aussi
un prérequis pour la stratifi cation des patients selon
des anomalies moléculaires spécifi ques afi n qu’ils
puissent bénéfi cier du traitement ciblé qui serait le
plus effi cace et approprié à leur cancer.
Essais cliniques
pour la médecine personnalisée
Un autre enjeu de taille est le développement de
thérapies qui ciblent ces aberrations moléculaires et la
conception des essais cliniques optimaux pour la MP.
Les médicaments qui ciblent des altérations molé-
culaires spécifi ques ont indéniablement fait leurs
preuves dans le traitement de certains types de
cancer. Cependant, si le nombre de thérapies ciblées
augmente quotidiennement, leurs applications
cliniques ne s’avèrent pas satisfaisantes, faute de
biomarqueurs et d’essais cliniques appropriés (33).
Dans le cadre de la MP, un biomarqueur doit être
testé dans des études prospectives randomisées pour
déterminer sa validation clinique. La majorité des
modèles d’études cliniques actuelles ne sont pas
appropriés pour évaluer des thérapies ciblées (1).
Plusieurs traitements ciblés ont un index thérapeu-
tique plus large que les agents de chimiothérapie
et produisent souvent des effets “hors cible” qui
conduisent à une surestimation de la sensibilité du
traitement. D’autre part, la majorité de ces trai-
tements sont cytostatiques et non cytotoxiques,

La Lettre du Cancérologue • Vol. XXI - n° 6 - juin 2012 | 293
DOSSIER THÉMATIQUE
ce qui nécessite un choix de end points adaptés.
La conception d’essais cliniques se basant sur une
population de patients sélectionnés selon un profi l
moléculaire spécifi que pour recevoir la thérapie
ciblée s’avère indispensable pour la validation des
biomarqueurs en clinique et l’évaluation même de
la thérapie ciblée (34). L’utilisation des techniques
génomiques de haut débit et d’imagerie dans les
essais cliniques standardisés faciliterait l’identifi ca-
tion des biomarqueurs prédictifs. Les biopsies doivent
être accessibles en routine pour les analyses molé-
culaires à haut débit. Les biomarqueurs potentiels
doivent être validés dans des études consécutives
portant sur la même population de patients, voire
dans une même étude intégrative avec 2 stades de
recrutement de patients selon des modèles statis-
tiques adaptés comme la randomisation adapta-
tive (35). La stratifi cation des patients selon des
biomarqueurs validés permet de meilleures pers-
pectives quant au résultat de la thérapie ciblée en
question. L’étude BATTLE constitue le premier essai
avec randomisation adaptative qui consiste à ajuster
la randomisation aux nouvelles données biologiques
obtenues en temps réel, ce qui permet d’enrichir les
groupes de répondeurs potentiels (10). Cette étude a
porté sur 4 biomarqueurs avec, au total, 11 anomalies
mesurables. Dans un premier temps, les patients ont
été répartis en 4 groupes de traitement (erlotinib,
vandétanib, sorafénib et erlotinib + bexarotène)
et la progression tumorale a été évaluée quelques
semaines après traitement par imagerie. Les analyses
moléculaires à partir de biopsies de ces patients ont
permis d’établir des catégories spécifi ques afi n de
diriger les nouveaux patients inclus vers un traite-
ment en fonction de la meilleure réponse constatée
par rapport au biomarqueur identifi é (10). L’exemple
de l’étude BATTLE constitue un virage important
pour la conception de nouveaux modèles d’essais
cliniques adaptés à la MP en cancérologie. Un défi
de taille dans ce contexte est d’anticiper l’évolution
de la technologie qui permettra d’étudier plus de
biomarqueurs en temps réel et, par conséquent,
d’adapter les modèles statistiques à cette nouvelle
génération d’essais.
Défi s socio-économiques
La mise en œuvre de la MP requiert un apport fi nan-
cier considérable dans les différentes infrastructures
médicales pour assurer :
➤
l’incorporation des différentes technologies à
haut débit ;
➤
la formation du personnel à l’utilisation de ces
technologies en routine ;
➤la standardisation des tests ;
➤
la mise en place d’un réseau bio-informatique
pour l’analyse des données et la corrélation avec
la clinique.
D’un autre côté, on s’attendrait à ce que la MP
engendre une réduction des coûts associés aux effets
indésirables et à la toxicité des médicaments (36).
En réalité, le rapport coût/effi cacité de la MP est
diffi cile à évaluer aujourd’hui, d’autant plus que les
nouvelles technologies de profi lage moléculaire à
haut débit évoluent rapidement et leur prix est revu
à la baisse. L’évolution rapide de ces technologies
qui deviennent moins coûteuses tout en gagnant
en performance constitue un défi majeur pour
leur utilisation en routine clinique. De nouvelles
versions des puces et des panels de biomarqueurs
sont fréquemment mises sur le marché et néces-
sitent, au niveau des plateformes concernées, des
essais de mise au point et de validation avant toute
utilisation clinique. Cet effort est coûteux et exige
un temps de travail important pour le personnel.
Une attention spéciale doit être accordée à la gestion
de l’évolution rapide des technologies à haut débit
tout en assurant la qualité des tests utilisés dans les
différentes plateformes.
De nombreux enjeux éthiques et sociaux sont
aussi associés à la MP, en particulier ceux liés à la
pharmaco génomique. Des problèmes éthiques de
confi dentialité et de respect de la vie privée se posent
et nécessitent une protection légale adéquate.
Le profi lage moléculaire des patients ayant pour but
initial de pouvoir administrer le traitement le plus
approprié à chacun d’entre eux peut révéler des infor-
mations sur des facteurs de prédisposition au cancer
ou à d’autres maladies impliquant ainsi les autres
membres de la famille (37). La question du droit de
savoir ou non devient donc capitale. L’utilisation
de données génétiques peut également mener à la
discrimination génétique par les assurances ou les
employeurs en fonction de la réponse d’un individu
à un médicament ou des facteurs de prédisposition.
L’éducation et la sensibilisation des patients ainsi que
du personnel médical aux différentes facettes de la
MP et à la signifi cation exacte des tests génétiques
et leurs implications deviennent indispensables.
L’accès équitable à la MP représente également
un problème éthique important, surtout au sein
du système de santé public français déjà endetté.
En dehors de la couverture sociale des traitements,
la prise en charge des différents tests de screening

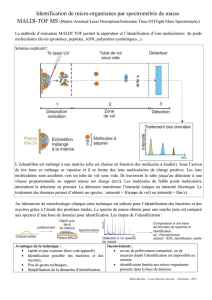
Figure. Les différents défi s de la médecine personnalisée.
Screening
moléculaire
du patient
Implémentation
de plateformes
de technologies
à haut débit
Standardisation des tests
Assurance qualité
Bio-informatique
Transparence et validation
des résultats
Disponibilité
du traitement adéquat
Développement
de nouveaux
médicaments
Considérations éthiques
Droit de savoir
Formation du personnel
Coût et couverture
par les assurances
Considérations éthiques
Consentement
Standardisation
de l’imagerie
et des biopsies
Profi l
moléculaire
Traitement ciblé
294 | La Lettre du Cancérologue • Vol. XXI - n° 6 - juin 2012
Les défi s de la médecine personnalisée en oncologie
DOSSIER THÉMATIQUE
La médecine personnalisée
doit être bien précisée. De plus, le fait que les médi-
caments développés ne puissent être disponibles
pour tous les patients peut représenter une faille
dans l’égalité et un problème éthique en soi (38).
L’offre même d’un médicament issu de la MP peut
poser problème, surtout si le patient refuse de subir
le screening, mais désire quand même avoir accès
au médicament. La stratifi cation des patients selon
le profi l moléculaire risque de créer de nouvelles
inégalités, dans le sens où certaines populations
de patients pourraient avoir un accès réduit à de
nouvelles thérapies. D’autre part, les entreprises
pharmaceutiques investiraient plus dans le déve-
loppement de nouveaux médicaments ciblant les
anomalies génétiques de populations larges (33, 39).
Conclusion
La mise en œuvre de la MP en routine nécessite
une collaboration étroite et synchronisée entre le
personnel médical, les patients, les scientifi ques
et les différentes plateformes techniques, bio-
informatiques et biostatistiques afi n d’assurer un
bénéfi ce clinique signifi catif (23). Les différents résul-
tats moléculaires obtenus doivent être transparents,
intégrés et validés par des réseaux bio-informatiques
adéquats et des scientifi ques. Les différents tests
de screening sur des échantillons tumoraux bien
défi nis doivent être standardisés pour assurer la
reproductibilité et la qualité des résultats, ce qui
peut s’avérer très coûteux, notamment pour la
formation du personnel impliqué. La mise en place
active de la MP en routine clinique nécessite ainsi
une réorganisation très coûteuse de toute l’infras-
tructure médicale. D’autre part, les considérations
socio-économiques et éthiques doivent être bien
identifi ées pour assurer la protection des individus et
leur couverture par les systèmes d’assurance publics
et privés (fi gure). Le développement des nouvelles
thérapies ciblées indispensables pour la pérennité de
la MP requiert également un bouleversement de la
conception des essais cliniques et un effort majeur
pour l’identifi cation de biomarqueurs et leur vali-
dation clinique dans des études prospectives (1). ■
1. Wistuba II, Gelovani JG, Jacoby JJ, Davis SE, Herbst RS.
Methodological and practical challenges for personalized
cancer therapies. Nat Rev Clin Oncol 2011;8(3):135-41.
2. International Human Genome Sequencing Consortium.
Finishing the euchromatic sequence of the human genome.
Nature 2004;431(7011):931-45.
3. Borden EC, Raghavan D. Personalizing medicine for cancer:
the next decade. Nat Rev Drug Discov 2010;9(5):343-4.
4. Diamandis M, White NM, Yousef GM. Personalized medi-
cine: marking a new epoch in cancer patient management.
Mol Cancer Res 2010;8(9):1175-87.
5. Fidler IJ. Tumor heterogeneity and the biology of cancer
invasion and metastasis. Cancer Res 1978;38(9):2651-60.
6. Jones S, Zhang X, Parsons DW et al. Core signaling
pathways in human pancreatic cancers revealed by
global genomic analyses. Science 2008;321(5897):
1801-6.
7. Varela I, Tarpey P, Raine K et al. Exome sequencing identi-
fi es frequent mutation of the SWI/SNF complex gene PBRM1
in renal carcinoma. Nature 2011;469(7331):539-42.
8. Sotiriou C, Piccart MJ. Taking gene-expression profi ling to
the clinic: when will molecular signatures become relevant
to patient care? Nat Rev Cancer 2007;7(7):545-53.
9. Ross JS. Multigene classifi ers, prognostic factors, and
predictors of breast cancer clinical outcome. Adv Anat Pathol
2009;16(4):204-15.
10. Kim ES, Herbst RS, Wistuba II et al. The BATTLE trial:
personalizing therapy for lung cancer. Cancer Discov 2011:
1;44-53.
11. Campbell PJ, Yachida S, Mudie LJ et al. The patterns and
dynamics of genomic instability in metastatic pancreatic
cancer. Nature 2010;467(7319):1109-13.
Références bibliographiques
 6
6
1
/
6
100%