Réplication - Faculté des Sciences de Rabat

Laboratoire de Microbiologie et Biologie Moléculaire
--------------------------------------
Université Mohamed V - Agdal• Faculté des Sciences
B.P. 1014 - Rabat - MAROC
Filière SVI - S6
Module de Génétique et Biologie Moléculaire –M21
Elément 2: Biologie Moléculaire –Pr. Bouchra BELKADI
Partie 2:
Expression génétique
Réplication
Faculté des Sciences - Rabat
Année universitaire 2009-2010

Plan
Introduction : Le Dogme Central
La réplication
I - Généralités de la réplication
II - Réplication continue et réplication discontinue
III –Réplication chez les procaryotes
IV- Fidélité de la réplication : PROOFREADING
V - La réplication et la division cellulaire
VI –La réplication chez les Eucaryotes

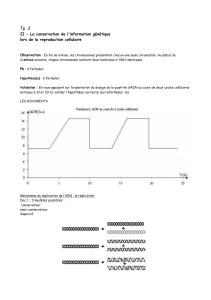
Macromolécules et informations génétiques
Génome: ensemble de gènes
Gène: unité fonctionnelle de l’information génétique.
Composition: acides désoxyribonucléiques (ADN),
séquences de bases puriques (Adénine, Guanine) et
pyrimidiques (Thymine, Cytosine)
INTRODUCTION

Formation de
la liaison
phosphodiester

PROTEINE
ARN
ADN
Matrice ADN
ADN polymérase III
Protéines de réplication
dNTP
ATP
Mg 2+
Matrice ADN (brin sens)
ARN polymérase
Facteurs de transcription
NTP
Mg 2+
Matrice ARNm
Ribosomes
Facteurs de traduction
Acides aminés
ARNt
Synthases
ATP,GTP, Mg 2+
Le dogme central
Transcription Traduction
Réplication
Le flux de l’information génétique
est unidirectionnel
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
1
/
37
100%