Base des études de liaisons
publicité

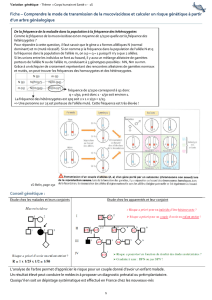
G15, Pr. Mandel Cours du 26/10/11 de 14h à 16h Groupe 02 M1 Pauline Muller et Thomas Thuet ________________________________________________________________________________ Génétique des maladies multifactorielles et prédisposition génétique I. Introduction Ce sont des maladies causées par plusieurs facteurs (environnement, génétiques, …). Ex : diabète, infarctus du myocarde, maladies auto-immunes, schizophrénie, SIDA ( environnement et facteur génétique => en Europe, 2% des gens sont à peu près résistants par délétion d'un gène codant pour le co-récepteur du VIH) Mêmes certaines maladies monogéniques sont influencées par l'environnement (famille, prise en charge thérapeutique, …). Est-ce qu'un facteur génétique est fort ou pas pour une maladie donnée ? une maladie génétique est souvent familiale épidémiologie : quelle est la fréquence de la même maladie chez les parents de 1er degré (père, mère, frère, sœur, fils, fille) ? Exemple: diabète de type I, PR II. Génome humain et variabilité : 3 milliards de paires de bases (pb) par génome, 2 copies de génome (soit 6 milliards de pb). Polymorphisme = fréquence allélique à une position donnée > 5%, environ tous les 400-500 pb. => SNP (Sigle Nucleotid Polymorphism) : 8 à 10 millions dans le génome Variants rares = fréquence allélique < 1% paradoxalement sont plus nombreux => 10 millions de différences d'un génome à un autre 0,1% de variation entre 2 copies de génome (N.B : entre nous et le chimpanzé, il y a 1% de variation). La variabilité est due parfois à la présence de 1 ou 3 copies au lieu de 2 = délétions et duplications => CNV (Copy Number Variant) variation du nombre de copies touche 10% du génome G15 Page 1 sur 7 III. Maladies multifactorielles et maladies monogéniques : ■ maladie monogénique = mutation du gène donne la maladie • Le risque de développer la maladie est : très très fort pour les maladies monogéniques si mutation nul si absence de mutation donc RR = ∞ (car 1/0) (ex: Huntington, mucoviscidose et CFTR...) • Dans le cancer du sein, il existe une mutation BRCA1 => le RR est de 12 (car il y a 85% de risque de développer le cancer si mutation présente, et 7% si absence de la mutation). ■ maladie multifactorielle = facteurs génétiques contribuent au risque mais ne provoquent pas la maladie à eux seuls => ils augmentent de 20 à 50% le RR => RR généralement faible par rapport à celui des maladies monogéniques (contre-exemple : maladie d'Alzheimer et un allèle ApoE4 où le RR = 3 et 2 ApoE4 où RR=9 à 10) 1. Médecine prédictive et personnalisée : ■ médecine prédictive = savoir, en étudiant le génome, si le patient est plus à risque de développer telle ou telle maladie => thérapeutique prédictive ou modification des facteurs de l'environnement pour diminuer le risque exemples: prise en charge du diabète, HTA, hypercholesterolémie.. ■ médecine personnalisée = le patient est déjà malade ; en fonction de la disposition génétique du patient, savoir si la thérapeutique aura une meilleure efficacité ou si cela va provoquer des effets indésirables 2. Maladies communes multifactorielles : Ex : spondylarthrite ankylosante (maladie rhumatismale sévère)=> variants du locus HLA (HLA B27 très pré-disposant pour la maladie) A partir des années 90-95, il y a une recherche systématique des variants aux prédispositions à ces maladies (PCR, séquençage) On effectue l'étude de gènes candidats = gènes responsables de pré-disposition ex : les gènes concernant la synthèse, les récepteurs, la dégradation de la sérotonine sont des gènes candidats pour la schizophrénie, gènes intervenant dans le métabolisme des lipides et du cholestérol pour les maladies cardio-vasculaires, gènes impliqués dans l’inflammation des cellules endothéliales pour l’athérome. Malgré une littérature confuse et contradictoire, de grandes découvertes sont faites G15 Page 2 sur 7 Apo E4 et Alzheimer => Apo E4 code pour un transporteur de cholestérol NOD2 (gène impliqué dans la réponse immunitaire innée) et la maladie de Crohn (maladie inflammatoire du côlon avec part importante des facteurs environnementaux) Depuis 2007, la révolution technologique permet d’analyser de grandes cohortes concernant des dizaines de milliers de patients, où plus de 500 000 voire 1 million de variations du génome sont analysées pour savoir où se situent les gènes associés à une pathologie. GWAS (Génome Wide Association Study) : stratégie suivie pour les maladies génétiques. A .Est-ce que la maladie a une part génétique ou non ? ■ Le premier élément indicateur est la récurrence familiale : → diabète de type 1 : 0,3 à 0,4% dans population générale, 3 à 8% si maladie chez parent de 1er degré donc RR multiplié 10 à 20 fois → schizophrénie : 1% dans population générale, 4 à 10% si maladie chez parent de 1er degré donc RR multiplié 7 fois en moyenne → asthme : 3,8% dans la population générale, 9,2% si maladie chez parent de 1 er degré, donc RR multiplié par 2,5. ■ Le fait qu’il y ait une agrégation familiale suppose un risque plus élevé, mais est-ce que tout ce qu'il y a dans une famille est génétique ? => attention famille partage le même mode de vie donc le même environnement !!!! • Pour séparer la famille de l'environnement, on a fait des études sur les jumeaux monozygotes, qui ont donc le même matériel génétique. • On s'intéresse à la concordance. On regarde l'incidence de la maladie chez les vrais et les faux jumeaux (même environnement, avec pour les uns 100% identiques au niveau du génome, pour les autres 50% de variation génétique partagée et 50% de différences). • exemples : hypertension artérielle => 2 fois plus de concordance pour les vrais jumeaux (10%) que pour les faux (5%) il y a donc une influence génétique, mais pas très forte (25% de concordance) polyarthrite rhumatoïde => 50% de concordance chez les jumeaux monozygotes, 10% chez les dizygotes Epilepsie idiopathique => 80% de concordance chez les monozygotes. C’est donc fortement génétique. cancer du sein => pas de différence entre monozygotes et dizygotes (c'était avant la découverte de BRCA1 et BRCA2, qui expliquent 7 à 10% des cancers du sein) maladie de Parkinson => idem que cancer du sein N.B : la maladie de Gaucher (maladie lysosomale avec déficit enzymatique) multiplie par 4 ou 5 le risque de la maladie de Parkinson (mutation faux-sens donnant une protéine anormale) maladie d'Alzheimer => < 60% de concordance chez les monozygotes, environ G15 Page 3 sur 7 25% chez les dizygotes autisme => 60% de concordance chez les monozygotes, < 10% chez les dizygotes Concernant l'autisme, maladie peu précise au niveau clinique, on pense que c'est probablement une maladie génétique souvent liée à des néo-mutations (caractère familial beaucoup moins présent). Si la mutation arrive pendant la spermatogenèse ou l'ovogenèse, les 2 monozygotes vont développer la maladie. Au contraire, chez les dizygotes ce sont des spermatozoïdes et des ovules différents, donc la néo-mutation peut apparaître chez l'un mais pas chez l'autre. B. Identifier des gènes de prédisposition : ■ études d'association ou cas-témoins : on recherche la fréquence des variants chez les malades idem chez les contrôles on compare les fréquences et on conclue ; si c’est statistiquement significatif, c’est associé à la maladie. => mais cela ne se fait que gène par gène (gènes candidats sont choisis en se basant sur des hypothèses physio-pathologiques) ■ études de famille : on balaye, avec une moins grande précision, l'ensemble du génome et cela permet de voir que dans telle ou telle région il y a tel ou tel gène à risque on essaye de le localiser par des études de liaisons. => problème !! cela ne permet que d’identifier de grandes régions, mais on a du mal à identifier quel gène en particulier. Base des études de liaisons : Mutation ou variant à risque : On suppose que sur le chromosome on a une mutation/un variant. • mutation concerne les maladies monogéniques responsable de la maladie • variant =polymorphisme concerne les maladies multifactorielles ■ hypothèse : variant pré-disposant à la polyarthrite rhumatoïde, présent sur le bras court d’un chromosome. → transmission du génome à l'enfant, avec recombinaisons homologues (au minimum une par bras de chromosome) → si le variant est donné à 2 des enfants de la 1ère génération, il y a 40 Mb en commun autour du variant → à la 2ème génération, il n'y a plus que 25 Mb en commun autour du variant G15 Page 4 sur 7 → à la 10ème génération, il n'y en a plus que 2 à 3 millions → à la 100ème génération, il n'y a plus que quelques centaines de kb en commun autour du variant cela est utilisé pour rechercher les variants de pré-disposition, car si ce variant expose à la maladie, il est plus fréquent chez les personnes ayant la maladie. Et si les parents ont un ancêtre en commum, ils ont un variant en commun, et on pourra alors explorer tout un fragment de chromosome en communs (études sib-pairs : quelles sont les régions en communs chez ces personnes ayant la même maladie ? est elle en commun avec plusieurs familles ?) Etudes d'association (= études cas-témoins) : On compare la fréquence allélique (analyse d'une seule variation!) ou haplotype (combinaison d’allèles correspondant à des polymorphismes très proches dans le génome donc la probabilité que la recombinaison sépare ces variants est quasi nulle) On peut donc analyser plusieurs variations grâce aux haplotypes = nombreux variants présents dans une région limitée transmis en bloc EN L'ABSENCE de recombinaison. Cela permet alors d'avoir plusieurs informations. Par exemple si une personne a le même haplotype qu’un autre : ancêtre commun probable. On compare la fréquence allélique (ou d’haplotypes) entre patients et contrôles. On mesure ensuite le risque relatif RR, ou plutôt l'ods ratio (OR) qui est à peu près équivalent au RR dans les maladies peu fréquentes. Présence allèle à risque + Absence allèle à risque - contrôles m -+ m-- malades m++ m+- Quelle est la probabilité d’être malade (m+), ayant l’allèle à risque (+) = m++, et celle de ne pas être malade (m-) sachant qu’il n’y a pas d’allèle à risque (-) = m-- OR = [m(++) x m(--)] / [m(+-) x m(-+)] (environ égal au RR) Si par exemple OR=3, si on a l’allèle : le facteur de risque est multiplié par 3. On définit aussi le risque attribuable (défini au niveau d’une population) = proportion des cas imputables au facteur de risque. RA = [f x (RR-1)] / [1 + f x(RR-1)] (f: fréquence de l’allèle) Si on a par exemple 50% de personnes ayant le facteur de risque, grâce au RA on montre que ce facteur de risque peut expliquer 10% de la maladie. Attention !!! => ce calcul ne tient pas compte des allèles protecteurs, qui diminuent le risque. Interprétation de résultat positif = statistiquement significatif : G15 Page 5 sur 7 → allèle est directement impliqué dans le risque pathologique, ce qui augmente le risque, auquel cas il faut un test fonctionnel exemple : HLAB27 et spondylarthrite ankylosante : pour prouver que ce variant est responsable de la maladie, on a crée des rats transgéniques ayant HLAB27 ; quand ils étaient exposés à une contamination bactérienne, le risque de présenter la maladie était élevé. ou → allèle est en déséquilibre de liaison avec un allèle proche impliqué fonctionnellement dans le risque Déséquilibre de liaison = association préférentielle entre 2 allèles Explication : dans une région, on a un variant donné. Au départ, sur un chromosome, d’autres variations sont présentes à côté ; le variant est transmis en bloc (haplotype). Donc si le variant est plus fréquent chez un malade, on a également un haplotype plus fréqeunt chez le malade. Au cours du temps, des mutations surviennent dans la région, et apparaissent ainsi de nouveaux variants. (plus un variant est apparu tôt, plus il est fréquent dans la population.) Si on a un variant responsable d’un risque accru d’une certaine maladie, il est survenu sur un chromosome avec d’autres variants ; donc si ce variant est augmenté chez le malade, les autres variants aussi sont augmentés dans la maladie. Donc on ne peut pas vraiment savoir quel est le variant causal. Il y a donc un déséquilibre de liaison entre le variant causal et les variants testés. Exemple : Si un variant est plus présent chez les malades, cela ne veut pas dire que c'est le variant fonctionnel, mais qu'il est peut-être en déséquilibre de liaison avec celui-ci. A 18,00% 42,00% 60% C T G 12,00% 28,00% 40% 30% 70% A et G = 60/40% C et T = 30/70% S'il y a équilibre de liaison, AC = 18%, etc... => les allèles sont indépendants les uns des autres. C T A 30,00% 30,00% G 0,00% 40,00% => chaque fois qu'il y a un C, il y a un A associé mais tous les A ne portent pas de C => ici, il y a un déséquilibre de liaison complet entre G et C G15 Page 6 sur 7 Exemple : très nombreux variants HLA. On a remarqué que certains variants étaient impliqués dans le rejet de greffe En 83 on s’est aperçu que dans certaines maladies, certains haplotypes étaient plus fréquents chez les malades avec un OR plus important pour la spondylarthrite ankylosante : 90% ont HLAB27 alors que parmi les contrôles seulement 10% ont HLAB27. Psoriasis : HLACu6, diabète ID :HLA DR3-DR4…ce sont des maladies auto-immunes. Or : l’hémochromatise ou l’hyperplasie surrénalienne ne sont pas des maladies auto immunes, mais ce sont des maladies monogéniques dont le gène est présent au milieu de HLA. Hémochromatose : mutation du gène HFE responsable du transport du fer ; cette mutation est survenue proche de l’allèle HLA A3 donc HLA A3 est beaucoup plus fréquent chez les patients ayant l’hémochromatose. Idem dans l’hyperplasie surrénalienne. Il s’agit de déséquilibre de liaisons. G15 Page 7 sur 7