Sujet BAC 2016 - biotechnologies stl - Izi-Bac

1
Correction Sujet Biotechnologies – bac STL option
biotechnologies 2016
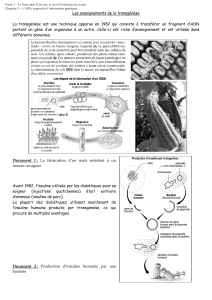
Q1 : Le plasmide pBR322 est adapté à une utilisation en génie génétique car il présente les
caractéristiques suivantes :
- Présence d’une origine de réplication (ori)
- Présence d’au moins un gène de sélection (ici 2 : tet R et amp R)
- Présence de sites de restriction (EcoR I, Hind III, BamH I)
Q2 :
Q3 : Le site bamH I est le seul situé au milieu d’un gène de sélection permettant ainsi de trier les
plasmides ayant insérés le gène d’intérêt ou non. En effet, le gène de sélection deviendra inactif si le
gène d’intérêt a été inséré, le gène n’ayant pas pu être traduit de façon continue en protéine.
Q4 : Le clone A correspondant à la bactérie transformée avec le plasmide pBR322 est résistant à
l’ampicilline et à la tétracycline car la bactérie a intégré le plasmide mais celui-ci ne contient pas le
gène de la proinsuline ; ainsi les gènes de sélection ampR et tetR ont pu être traduits en protéine
conférant à la bactérie une résistance pour les 2 antibiotiques.
Le clone B contient le plasmide recombinant. Ainsi, le gène d’intérêt a pu s’insérer au niveau du site
BamH I empêchant la traduction du gène tetR et inactivant ainsi le gène de résistance tetR. On
obtient ainsi une absence de résistance à la tétracycline pour ce clone.
Le clone C contenant uniquement le gène de la proinsuline circularisé ne contient pas les plasmides
pBR322 ou recombinant. Ainsi le clone n’a pas acquis de résistance à l’ampicilline et à la tétracycline.
Le clone D n’a pas du tout été transformé (pas de plasmide ni de gène de la proinsuline). Ainsi il n’a
pas pu acquérir de résistance d’où une absence de sensibilité aux 2 antibiotiques pour ce clone.
EcoR I et Hind III BamH I = site de restriction
Gène proinsuline inséré
Origine de réplication
Gène ampR
Gene tetR
Schéma annoté du plasmide
recombinant obtenu
en partenariat avec epicureweb.fr 1
Gabarit - Copie.indd 1 21/06/2016 09:07

2
Q5 : Le clone de type A est résistant à l’ampicilline et à la tétracycline ainsi il est capable de pousser
en leurs présences. Je relève donc les colonies ayant poussées sur les deux milieux soit les colonies :
1-3-4-6-7-8-9-10-12.
Q6 : Les colonies de bactéries transformées par le plasmide recombinant sont celles résistantes à
l’ampicilline mais sensibles à la tétracycline. Ainsi elles doivent avoir poussées sur le milieu 1 mais
pas sur le milieu 2 ; ce sont donc les autres colonies : 2-5-11.
Q7 : La durée de la phase de latence pour le milieu M1 est de 45 min
La durée de la phase de latence pour le milieu M2 est de 25 min
Q8 : Détermination des vitesses spécifiques de croissance :
Pour M1 je choisis 2 points en phase exponentielle (140 ; 3) (50 ;0.5)
µexpo = ln140-ln50/3-0.5 µexpo = 0.412 min-1
Pour M2, je choisis 2 points en phase exponentielle (40 ;0.75) et (60 ;1.05) soit
µ expo = ln60-ln40/1.05-0.75 µexpo = 1.35 min-1
Q9 : le milieu M2 contient davantage d’éléments nutritifs que le milieu M1 (glucose, extrait cœur
cervelle). La phase de latence est plus longue dans le milieu M1 car la bactérie doit synthétisée les
enzymes nécessaires à la dégradation des éléments nutritifs (ici l’extrait de viande et les peptones)
tandis que dans le milieu 2 il y a du glucose qui peut être directement dégradé d’où un début de
croissance plus rapide.
Q10 : Le milieu permettant une croissance optimale du clone D’E coli est le milieu 2 car il contient
davantage d’éléments nutritifs ce qui en fait un milieu permettant une croissance plus rapide du
clone (phase de latence plus courte) et une croissance en phase exponentielle plus élevée.
Q11 : La phase stationnaire composée de billes de gels poreuses permet le passage à l’extérieur de
l’insuline mature (molécule d’intérêt) tandis que le peptide C lui pénètre dans les billes. La phase
mobile, ici du tampon, permet la création d’un flux entrainant les différentes molécules. Ce type de
chromatographie se nome chromatographie d’exclusion, elle permet une séparation en fonction de
la taille et de la forme.
Q12 : La masse moléculaire de l’insuline mature est : 100*51 = 5100 Da
La masse moléculaire du peptide C est : 100*31 = 3100 Da
Q13 : le gel Sephadex G25 est le plus adapté à la purification de l’insuline mature car il retient les
molécules de 1000 à 5000 Da or le peptide C qui doit être retenu ici fait 3100 Da : il rentre donc dans
cette intervalle. De plus l’insuline mature sera ici rejetée des billes car sa taille est de 5100 Da donc
supérieure à l’intervalle de masses moléculaires des composés ralentis par le gel Sephadex G25.
en partenariat avec epicureweb.fr 2
Gabarit - Copie.indd 2 21/06/2016 09:07

3
Q14 :
Q15 :
L’absorbance à 450 nm est proportionnelle à la concentration d’insuline présente car chaque
molécule d’insuline fixée permettra la dégradation par une peroxydase d’une molécule de TMB en un
substrat coloré. Ainsi, plus il y aura d’insuline, plus il y aura de substrats colorés (d’où une
proportionnalité entre l’absorbance et le produit coloré).
Q16 :
TMB
Substrat
coloré
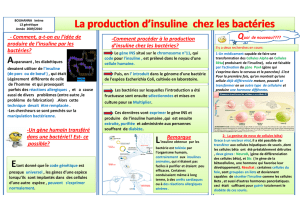
Obtention d’une souche d’E coli recombinante :
utilisation de l’enzyme BamH I. Le clone B est
retenu c’est à dire les colonies poussant sur la
boite 1 mais pas sur la boite 2
Optimisation de la culture de la souche
sélectionnée : utilisation du milieu 2 présentant
une phase de latence plus courte et une vitesse
spécifique de croissance plus élevée.
Purification de l’insuline produite par la bactérie
recombinante : utilisation d’une
chromatographie d’exclusion avec le gel
Sephadex G25
Dosage de l’insuline humaine purifiée : lecture
des cupules après réaction à 450nm avec une
absorbance proportionnelle à la concentration
d’insuline présente dans l’échantillon
en partenariat avec epicureweb.fr 3
Gabarit - Copie.indd 3 21/06/2016 09:07
1
/
3
100%