TP2 - ESI

TP2

Devoir 1
Des questions??

Traduction
•Voir le site pour une petite astuce:
http://www.ncbi.nlm.nih.gov/Taxonomy/Utils/wprintgc.cgi
•Remarquez dans quel ordre sont disposés les
codons
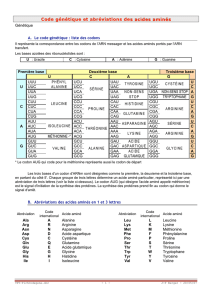
•1. Un nucléotide (ACGT) peut être associée à un chiffre.
•2. On peut compter en base 4 de la même façon que l'on compte
en base 10.
•3. Un codon peut très bien être la représentation d'un nombre en
base 4.
•Par exemple, si T=0 , le codon TTT = 0*16+0*4+0*1 = 0

Format de sortie suggéré
Frame+1 :
MVRRPWRGEAPSPAPTRAPRPPGPTGHPQPSWPRTQTPPLTLLLPAGCSCPSPPPRPTSRTS
Frame+3 :
MAPSSQGRGSRGLREV
Frame-2 :
MSAARPGVPPRPKHKPWRARGPALFWSPQTQREPTMVLSPADKTNVKAAWGKVGAHAGEG

Vérification: Emboss
•Getorf
% getorf -minsize 100
Finds and extracts open reading frames (ORFs)
Input sequence(s): SeqTest
Output sequence [eclaci.orf]:
 6
6
 7
7
 8
8
 9
9
1
/
9
100%