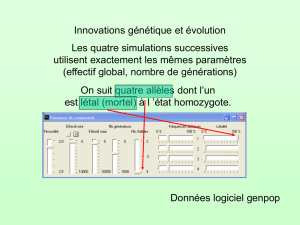
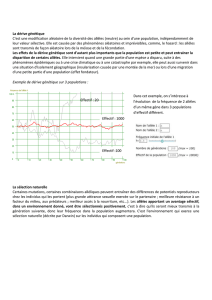
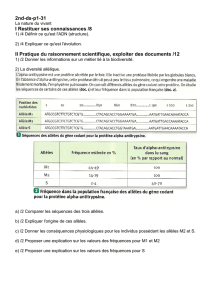
"Sélection Naturelle".

Analyse moléculaire
de la sélection naturelle
1. Qu’est-ce que la sélection naturelle ?
2. Quels sont les effets de la sélection naturelle ?
3. Comment détecter la sélection naturelle ?

1. Qu’est-ce que la sélection naturelle ?
Charles Darwin Alfred Russel Wallace
« Pouvons-nous douter… que les individus ayant quelque avantage, aussi léger soit-il, sur les
autres, auraient une meilleure chance de survie et de procréer leurs caractères? D'autre
part, nous pouvons être sûrs que n'importe quelle variation du moindre degré nuisible serait
indéniablement détruite. C'est cette conservation des variations favorables, et le rejet des
variations nuisibles, que j'appelle "Sélection Naturelle". Charles Darwin (1859) »
Sélection Naturelle: processus par lequel des variations favorables vont
tendre à augmenter en fréquence au sein d’une population, et inversement
pour des variations défavorables.

Sélection Naturelle: survie ou reproduction différentielle, en moyenne,
des différents phénotypes dans une population.
Agit sur les phénotypes
Comme les phénotypes sont, en partie, déterminés pas des génotypes, la
sélection naturelle cause des variations de fréquences d’allèles
Elle ne peut opérer que si
• les individus d’une population ne sont pas tous identiques (variation)
• une part de cette variation affecte des traits liés à la capacité à
survivre et/ou se reproduire
Elle ne cause une évolution que si :
• les différences de survie/reproduction sont dues à des variations
génétiques héritables
1. Qu’est-ce que la sélection naturelle ?

1. Qu’est-ce que la sélection naturelle ?
Une des quatre forces évolutives
Mutation
Migration
Sélection
Dérive
Impliquée dans :
l’évolution adaptative
les processus de spéciation
déterminisme des systèmes de reproduction …

Peut opérer de deux façons:
• par différences de survie entre génotypes
• par différences de fertilité
(les deux sont importants)
Peuvent faire l’objet d’un compomis:
•Phyllomedusa bicolor : chant nuptial et prédation
par les chauve-souris
•Mantes, araignées Nephila fenestrata :
cannibalisme sexuel :
Valeur sélective: capacité d’un individu à
transmettre ses propres allèles à la génération suivante
1. Qu’est-ce que la sélection naturelle ?
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
 59
59
 60
60
 61
61
 62
62
 63
63
 64
64
 65
65
 66
66
 67
67
 68
68
 69
69
 70
70
 71
71
 72
72
 73
73
 74
74
 75
75
 76
76
 77
77
 78
78
1
/
78
100%