Développement d`un biosenseur utilisant le « Microarray » à

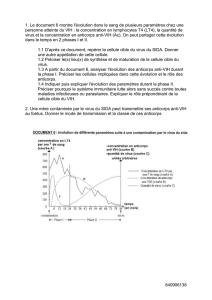
1
MAÎTRISER LA LUMIÈRE POUR NOS PARTENAIRES
CHALLENGING LIGHT FOR OUR PARTNERS
Présenté par
Isabelle Noiseux
Québec, juin 2008
Jean-Pierre Bouchard
Fabien Claveau
Shu Chen
Marci Vernon
François Babin
François Roy-Moisan
Jocelyne Osouf
Martin Briand
Michel Fortin
Patrice Desroches
Stéphanie Jutras
René Beaulieu
Développement d’un
biosenseur utilisant le
« Microarray » à anticorps
pour la détection de
bactéries
INO (Institut National d’Optique)
Québec, Canada
Micro
Micro-
-fabrication
fabrication
Micro
Micro-
-optics
optics
Optical
Optical
Design
Design
Microsystems
Microsystems
(MEMS/MOEMS)
(MEMS/MOEMS)
Micro
Micro-
-fabrication
fabrication
Micro
Micro-
-optics
optics
Optical
Optical
Design
Design
Microsystems
Microsystems
(MEMS/MOEMS)
(MEMS/MOEMS) Industrial
Industrial
Optics
Optics
BIOPHOTONICS
BIOPHOTONICS
Agrophotonics
Agrophotonics
Advanced
Advanced
Imaging
Imaging
Systems
Systems
Industrial
Industrial
Vision &
Vision &
3D Sensors
3D Sensors
Industrial
Industrial
Optics
Optics
BIOPHOTONICS
BIOPHOTONICS
Agrophotonics
Agrophotonics
Advanced
Advanced
Imaging
Imaging
Systems
Systems
Industrial
Industrial
Vision &
Vision &
3D Sensors
3D Sensors
Specialty
Specialty
Optical
Optical
Fibres
Fibres
Lasers
Lasers
Optical
Optical
Communications
Communications
Fibre
Fibre Optical
Optical
Components
Components
Specialty
Specialty
Optical
Optical
Fibres
Fibres
Lasers
Lasers
Optical
Optical
Communications
Communications
Fibre
Fibre Optical
Optical
Components
Components
¾Organisme privé sans but lucratif
¾R et D contractuel en optique et photonique

2
Objectif
CONTEXTE
¾Pourquoi ce projet?
NOTRE CONCEPT
¾Démarche technique
¾Développement de méthode
¾Test de quantification
VALIDATION
¾Optimisation du prototype
¾Validation avec salmonelle
¾Conclusions et perspectives
Contexte
L’identification et la quantification des bactéries
soulèvent des préoccupations dans un certain
nombre de marchés diversifiés, y compris
l’agroalimentaire, l’environnement, la sécurité et la
pharmaceutique
Marché de la microbiologie industrielle
-3,2 milliards en 2003
-4,9 milliards en 2008
Marché des essais diagnostiques
-28 milliards en 2003
-39 milliards en 2008

3
Contexte
L’objectif visé était d’élaborer un système qui respectait
les exigences suivantes :
¾Détection, identification et quantification simultanées
de plusieurs espèces de bactéries
¾La méthode doit être objective et ne doit exiger
aucune compétence particulière
¾Le test doit être simple, rapide et peu coûteux
¾Le coût d’achat doit être faible et le délai de mise en
œuvre minimal
Approche technique : Biosenseur
Anticorps
Anticorps
Sandwich
Sandwich
Support
Support
Anticorps
capturé
Anticorps
Anticorps
capturé
capturé
Antigène
Antigène
(
(bactérie
bactérie)
)
1) Élément
biologique
sensible
2) Élément
détecteur
3) Unité de
traitement
de signaux

4
Démarche technique : Microarray à protéine
Surface d’écriture
Z11
Z12
Z21
Z22
Élément biologique sensible
Impression de
l’anticorps sur la
lame porte-objet
à l’aide d’un
schéma
sélectionné

5
Notre démarche
Surface d’écriture
8
0.5
2.5
2.5
0.5
2.5
5.0
0.5
0.5
2.5
0.50.5
12
POS
NEG
NEG
7C4 0.5 mg/mL
7C4 1.0 mg/mL
7C4 2.0 mg/mL
7C4 3.0 mg/mL
7C4 4.0 mg/mL
Dimension en mm
Élément biologique sensible
0,5 mg/mL 4,0 mg/mL1,0 mg/mL 2,0 mg/mL 3,0 mg/mL
Images fluorescentes des anticorps imprimés
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
1
/
13
100%